
2024-05-16

基因组学和蛋白质组学经过多年的发展,已取得了令人瞩目的成就,如二代、三代测序技术已将基因测序带到了一个高度成熟与广泛适用的新时代,不仅显著提高了测序速度和通量,也极大地降低了成本;2023年Thermo公司推出的全新Orbitrap Astral质谱仪,更是引领蛋白质组学研究迈入到极微量、超高通量、超高覆盖度的阶段,使得全蛋白质组研究几近成为了可能。 代谢组学(Metabolomics)是“组学”大家族的最新成员,旨在全面表征生物样本中的小分子化合物。代谢组学却没能顺利的复刻基因组与蛋白质组的成功,仍然存在诸多技术难题。这是由于代谢物本身的特点和性质所导致:复杂多样的理化性质、广泛的浓度范围以及庞大的代谢组体量且尚未确定大小[1]。这些问题加在一起构成了代谢组学工作流程中许多问题的根源,其中最为关键和核心的就是代谢物定性问题,如何高通量和高深度的实现代谢物的精准定性,已成为代谢组学研究者难以跨越的障碍。 代谢物的定性到底有哪些困境呢?下面我们举一些栗子
1、小分子化合物巨大的数量和理化多样性
截止2024年,PubChem数据库共收录化合物1.18亿条,ChemSpider收录更是达到1.29亿!而这些化合物根据ClassyFire[2]的分类法,可以分为4,825种类别,不同类别之间的理化性质均有区别,甚至有的千差万别。再回过头来看A/G/C/T、20种氨基酸,是不是亲切的多。这样的复杂性,也决定了想通过一种方法来实现样品中所有代谢物的检测,几乎是不可能完成的事。 ClassyFire分类法
2、大量存在的同分异构体
根据百科词条:将分子式相同、结构不同的化合物互称同分异构体。同分异构体的存在,使得化合物无法仅依靠精确分子量实现准确定性。我们以代谢组学研究最常用的HMDB数据库为例,该库共收录代谢物22w+,我们筛选50-1,500分子量范围的共200,798条进行统计,发现仅有6%的化合物不存在同分异构体,53%的化合物同分异构体达到100以上,非常惊人的数字。 HMDB库50-1500分子量范围的同分异构统计 更糟糕的是,除了传统的同分异构体,分子质量十分接近的化合物,对于质谱来说要实现区分亦是个挑战,比如m/z 213.0975这个MS1信号,仅通过精确分子量来匹配(M+H),可以匹配上3个同分异构和另一个不同分子式但MS1在5ppm误差范围内的候选物。 所以仅用MS1精确分子量来做化合物定性,那要十分慎重了。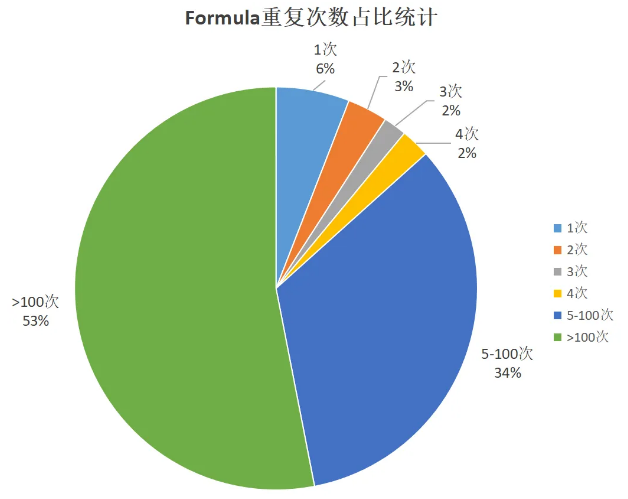
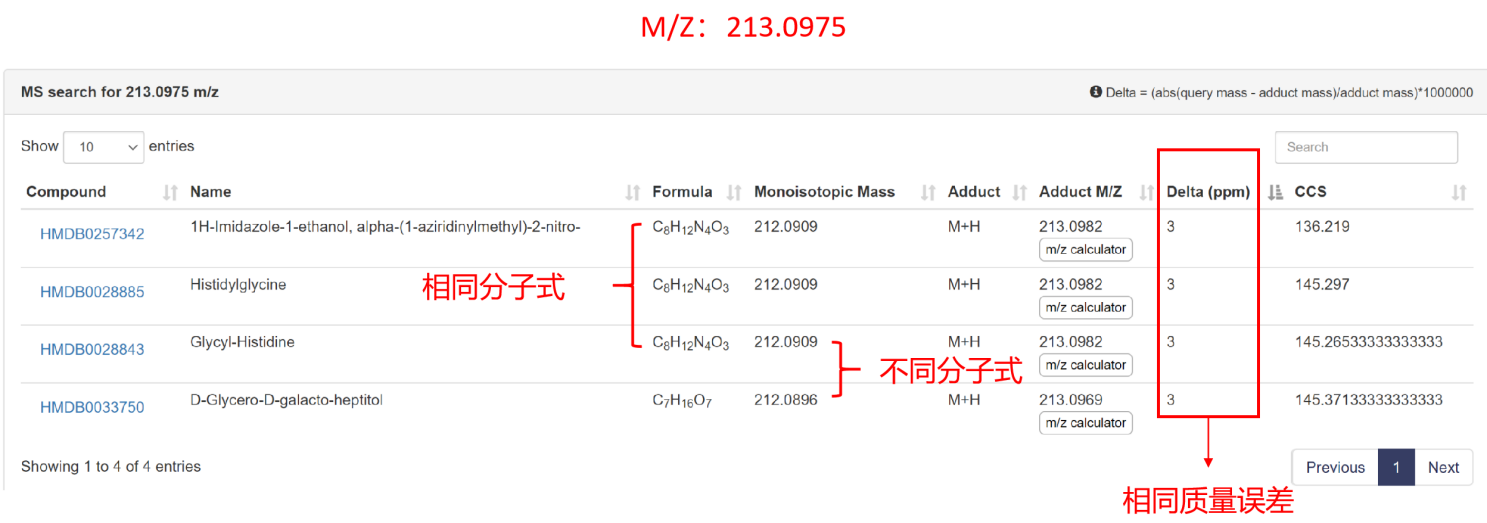
3、质谱信号的复杂性
代谢物的定性主要依靠色谱质谱信息来实现的,以应用最广泛的LC-MS平台为例,MS信号主要来源:代谢物、污染物、人工制品、背景噪音等,此外还有代谢物产生的同位素、加合物、多聚体、电荷、中性片段等[1],极其繁杂,要从这么多信号当中识别真正代谢物的MS信号,并实现代谢物的准确定性,绝非易事。 繁杂多样的质谱信号来源 在非靶向代谢组学研究中,代谢物定性的流程并不复杂,即通过采集到的样品中代谢物色谱质谱信息与标准物质的色谱质谱信息进行比对,完成匹配的即可实现定性。其中匹配的信息主要包括MS1(一级质谱,可获得精确分子量)、MS2(二级质谱,可获得碎裂特征信息)、RT(保留时间,主要用于区分同分异构体),还有CCS(碰撞截面积,离子淌度型质谱采集到的一种参数,主要作用仍然是区分同分异构体)。 但如何解决定性准确性和鉴定覆盖度难题呢?不得不重点介绍今天的主角——代谢数据库。代谢数据库主要包含上述3维或4维(是否含CCS)信息,数据库图谱信息的质量和覆盖范围对定性起着决定性作用。 常用的代谢数据库分为以下几种类型: 1.自建标准品库:自行购买或合成标准品,在自己的质谱平台采集,获得MS1、MS2、RT等信息,构建一个本地的标准品数据库; 2.商业数据库:已完成标准品信息采集并整合成直接可使用的收费数据库,大多也是实际标准品采集获得的信息构建,常用的有mzCloud、NIST、Metlin等; 3.公共数据库:部分单位或实验室,将自己平台采集的标准品图谱信息或直接整合好的数据库公开,供免费下载使用,MoNA库已将大多常用公共库做了整合,缺点就是信息较为混乱,直接使用问题较多; 4.计算机模拟数据库:根据化合物结构性质、代谢反应等信息,通过计算机模拟产生预测的图谱信息构建的库,有多种不同的预测策略和方法,随着AI模型的不断发展,该类型的库有望成为一大趋势。 计算机模拟建库在质谱解析应用中潜力巨大 从定性准确性角度,数据库1>2>3>4,但现实情况是可购买的标准物质十分有限,大概仅几千上万种,而且成本极其高昂,这对于数量如此庞大的代谢组,简直是杯水车薪,于是计算机模拟数据库成为了一种潜力巨大的解决方案,尤其在AI算法突飞猛进的今天[3]。该策略不仅可以通过AI模型来生成化合物的MSMS图谱,还可以实现RT、CCS值的预测,进一步过滤假阳性,提高鉴定的准确性。 计算机模拟建库在小分子和大分子定性方面,均已有着比较广泛的应用,比如在大分子蛋白组领域,大名鼎鼎的蛋白搜库软件DIANN[4]能实现高质量的Library-free DIA搜库,就依赖于其特有的Deep neural networks(DNNs)算法,生成模拟的质谱和RT信息实现蛋白的定性;脂质组方面,常用软件LipidBlast亦是通过计算机模拟产生的MSMS图谱库,来实现脂质定性[5]。包括国内代谢组大牛开发的MetDNA基于代谢反应网络的代谢物鉴定算法也有着不错的应用[6]。 通过计算机模拟的方式获得数据库来实现代谢物定性,是否能获得代谢组领域同行认可呢?S Alseekh联合国内代谢组顶级大牛罗杰、许国旺等人2021年在著名期刊《Nature methods》上发表了一篇文章[7],重新定义了代谢物鉴定分级level,其中将计算机模拟定性做了明确的划分: 新代谢物鉴定level 经典的代谢物定性分级方式发布于2007年[8],一经发布即受到广泛应用,直到今天仍然是代谢组学研究经典参考文献,但S Alseekh等人新发布的定性level的划分更加贴合如今的技术发展现状,比如将预测图谱的定性分级清晰纳入进来,有助于代谢物定性新技术的发展和应用。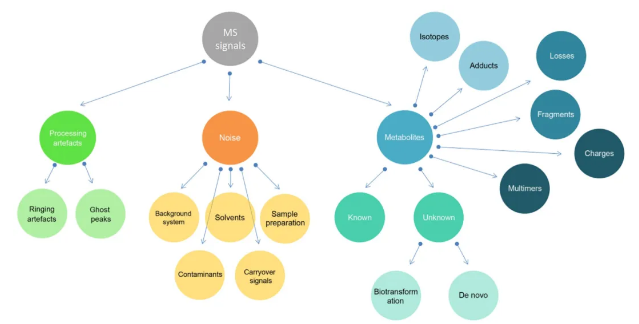
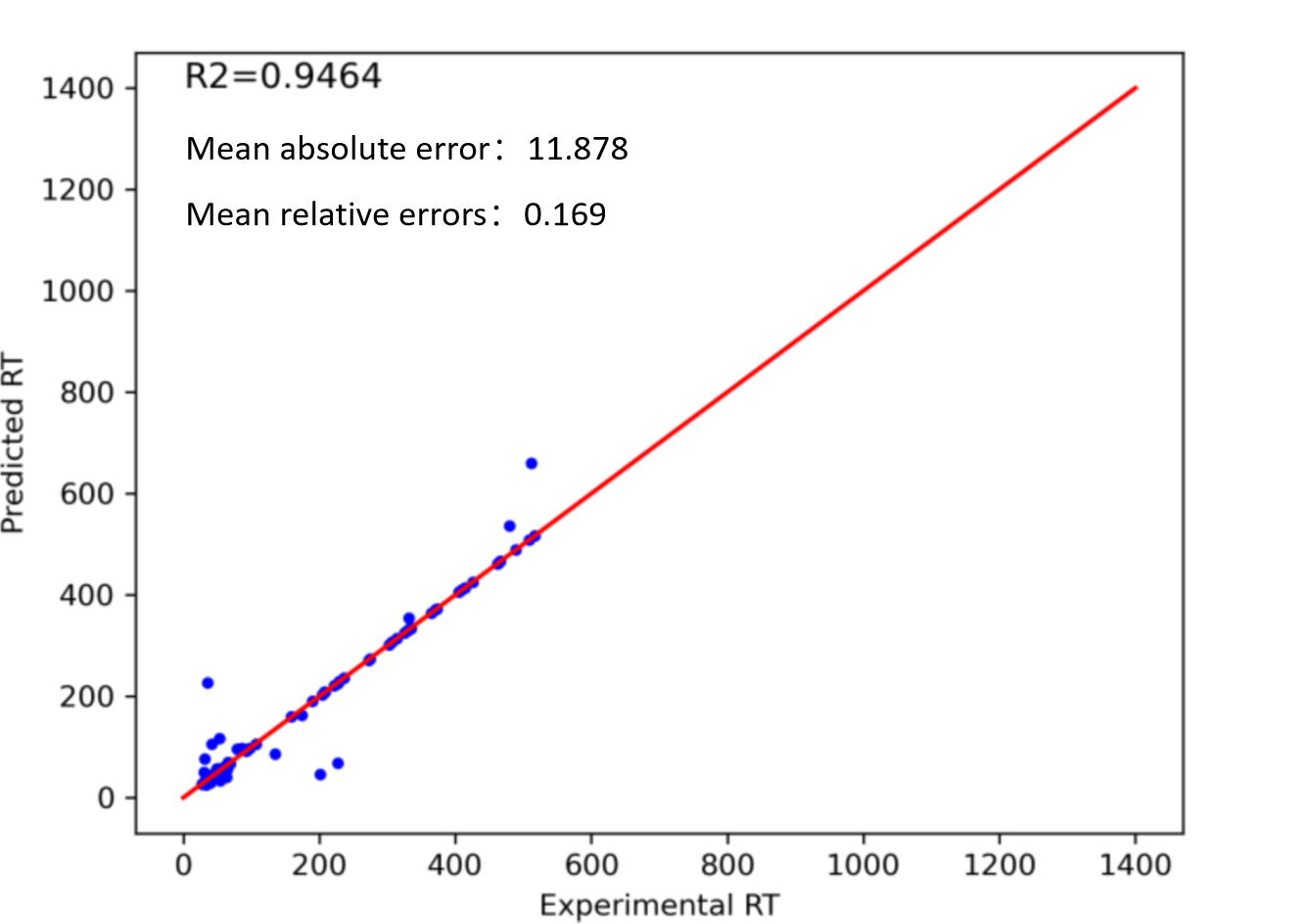
计算机模拟构建的代谢库在实际应用中是否真的靠谱呢,下面通过派森诺全新一代数据库PSNGM实测数据来一探究竟。 派森诺PSNGM一经发布就受到老师们广泛好评,尤其是我们的分类库结合的AI预测模型,让代谢物的定性上升了一个台阶。我们选取了使用PSNGM的肠道库、医学库以及植物库分析的项目数据进行了统计分析,我们对比了实验图谱匹配(level A-B(i))与AI预测库(B(ii))的定性结果,在三大库中,分别有62.2%、60.8%、61.2%的鉴定结果在标准品库定性结果中得以验证,说明AI库的准确性较高;同时相对于标准品库,AI库的应用能让鉴定数量有30%-38%左右的提升,很好的补充了标准品库的不足。 为了验证RT模型的可靠性,我们从自建库中随机挑选了100个标准品进行验证,RT预测的准确性达到了94%左右,平均误差在11.9s左右,这样的表现,RT的过滤将会大大降低假阳性的几率,让定性可靠性进一步提升。 代谢组学技术发展至今,仪器的性能反而不是如今的瓶颈问题,打造和构建高质量、高性能、高覆盖的代谢数据库成为了解码代谢组学研究的关键钥匙,派森诺打造的PSNGM代谢库期望能为代谢组学研究提供更好的支持,后续我们会陆续揭秘PSNGM代谢库及实测数据,期待大家的关注!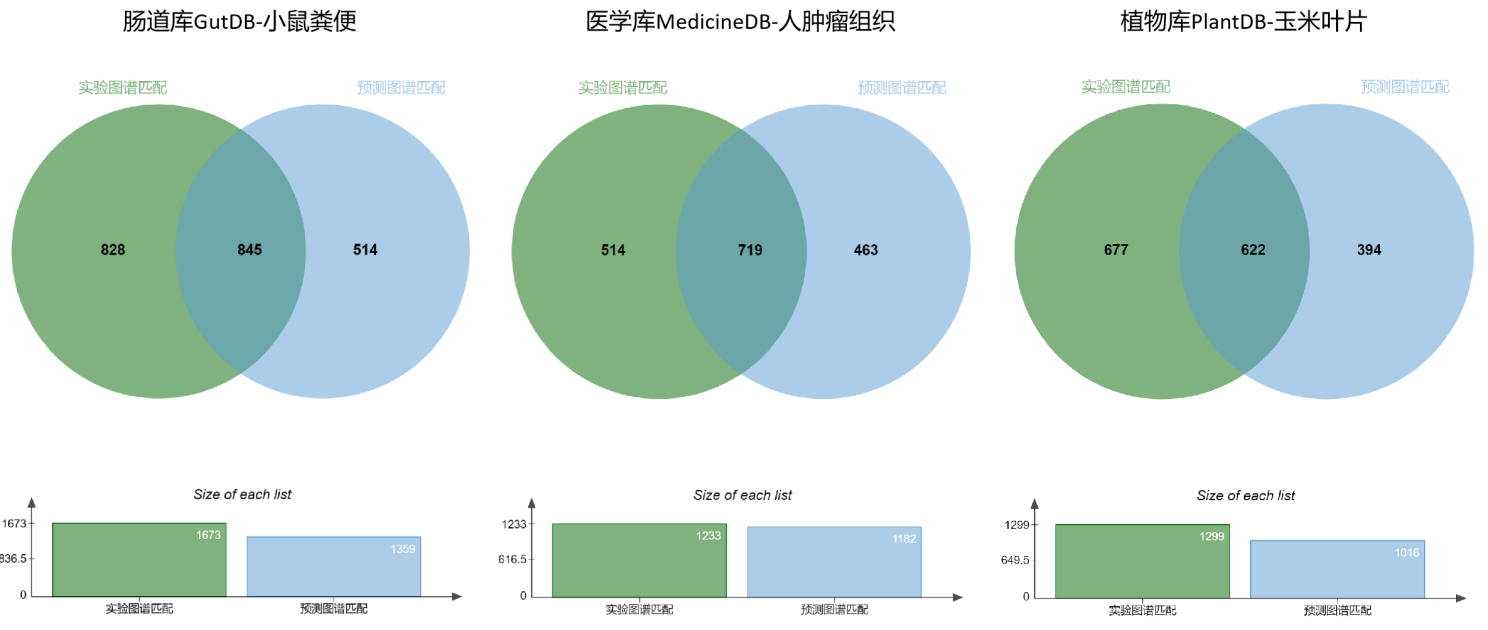
参考文献: 1.Godzien J, de la Fuente A G, Otero A, et al. Metabolite annotation and identification[M]//Comprehensive Analytical Chemistry. Elsevier, 2018, 82: 415-445. 2.Djoumbou Feunang Y, Eisner R, Knox C, Chepelev L, Hastings J, Owen G, Fahy E, Steinbeck C, Subramanian S, Bolton E, Greiner R, and Wishart DS. ClassyFire: Automated Chemical Classification With A Comprehensive, Computable Taxonomy. Journal of Cheminformatics, 2016, 8:61. 3.Cai Y, Zhou Z, Zhu Z J. Advanced analytical and informatic strategies for metabolite annotation in untargeted metabolomics[J]. TrAC Trends in Analytical Chemistry, 2023, 158: 116903. 4.Demichev V, Messner C B, Vernardis S I, et al. DIA-NN: neural networks and interference correction enable deep proteome coverage in high throughput[J]. Nature methods, 2020, 17(1): 41-44. 5.Kind T, Liu K H, Lee D Y, et al. LipidBlast in silico tandem mass spectrometry database for lipid identification[J]. Nature methods, 2013, 10(8): 755-758. 6.Shen X, Wang R, Xiong X, et al. Metabolic reaction network-based recursive metabolite annotation for untargeted metabolomics[J]. Nature communications, 2019, 10(1): 1516. 7.Alseekh S, Aharoni A, Brotman Y, et al. Mass spectrometry-based metabolomics: a guide for annotation, quantification and best reporting practices[J]. Nature methods, 2021, 18(7): 747-756. 8.Sumner L W , Amberg A , Barrett D ,et al.Proposed minimum reporting standards for chemical analysis Chemical Analysis Working Group (CAWG) Metabolomics Standards Initiative (MSI)[J].other, 2007, 3(3).DOI:10.1007/s11306-007-0082-2.