2024-05-23

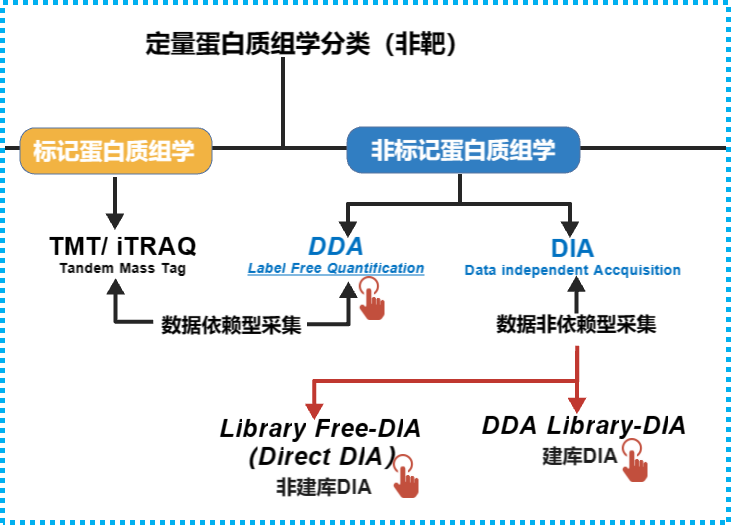
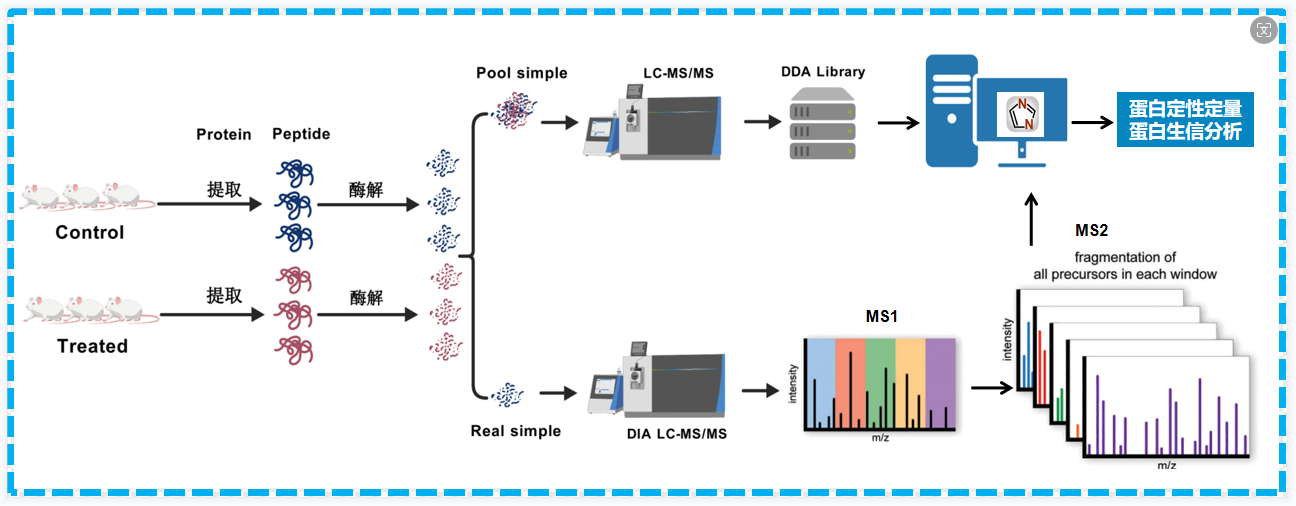
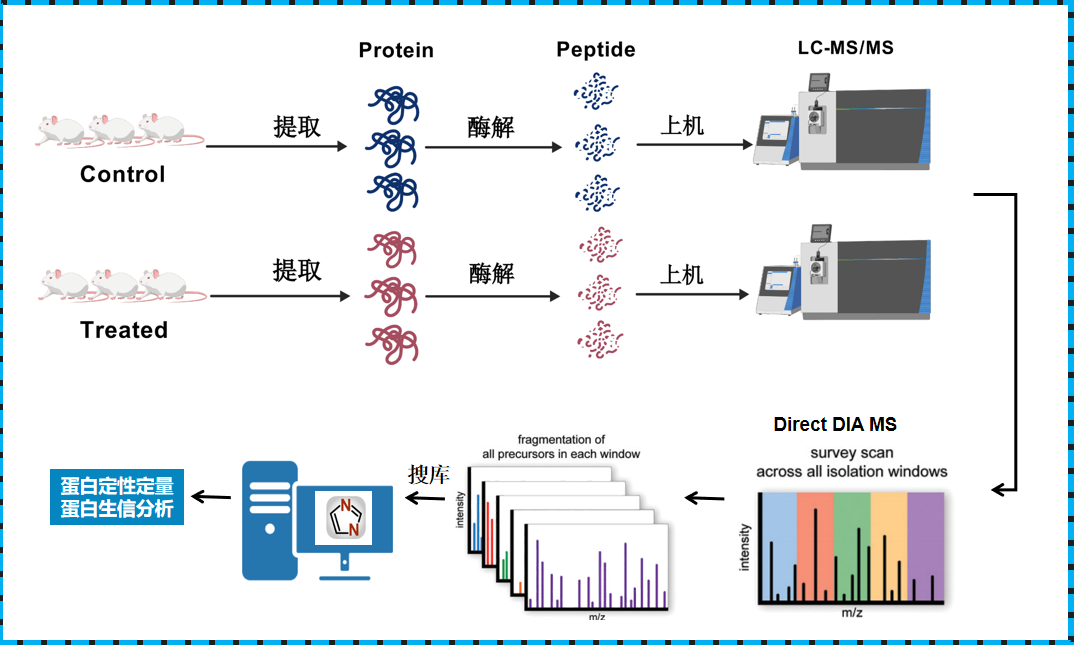
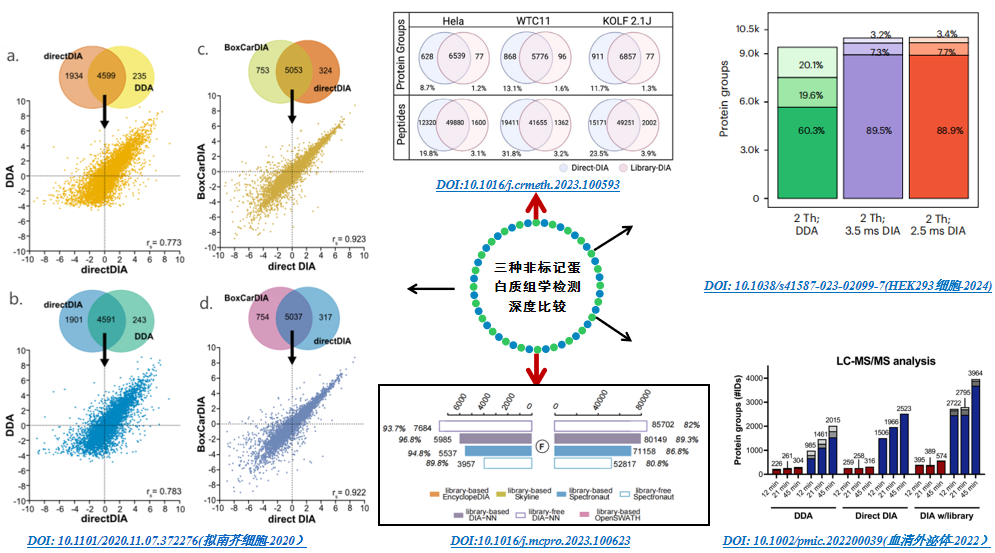
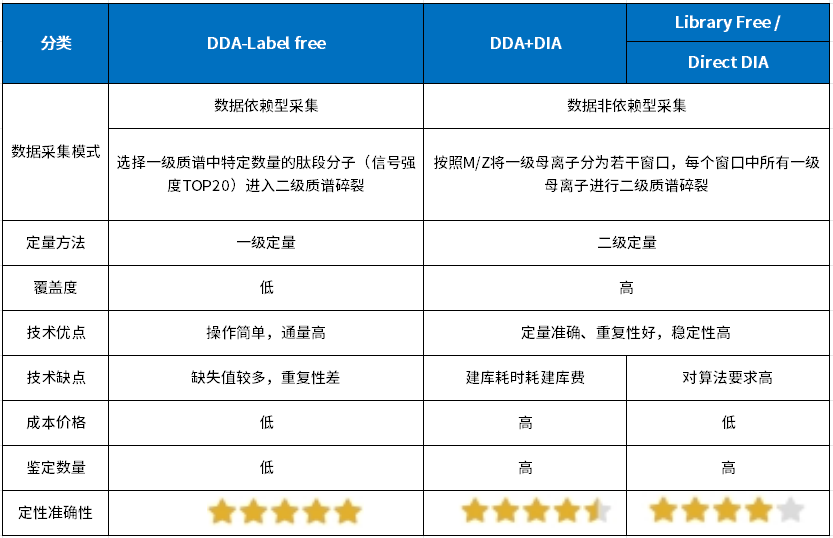
蛋白质作为生物功能的执行者,在系统生物学中发挥重要的作用。蛋白质组学与微生物组学、转录组学和代谢组学之间存在着密切的关系。这些组学技术相互补充、相互促进,共同推动生物学领域的发展。基于质谱的蛋白质组学研究方法多种多样,该如何选择?相信不少老师心中都有此疑问,让我们一起从不同的维度了解今天的主角-非标记定量蛋白质组学。 非标记定量蛋白质组学分类 蛋白质组学方法可以按照前处理以及定量原理不同,初步分为非标记定量蛋白组学和标记定量蛋白质组学。非标记蛋白质组学顾名思义即不对样本中提取的蛋白或者肽段进行同位素标签标记,直接通过液相质谱对蛋白质酶解肽段进行检测,通过比较不同样品间肽段的谱图强度,从而获得样本中相对定量蛋白质的表达水平。相比TMT/iTRAQ标记蛋白质组学来说,不存在因为标记试剂数量有限的样本检测通量问题以及样本标记产生批次效应问题。 图1 非标记蛋白质组学分类 非标记蛋白质组学根据数据采集模式和搜库方式,分为DDA-Label free、DDA Library +DIA以及Library free DIA(Direct DIA)三类。我们将从技术原理流程、蛋白检测数量、价格等方面全面解析这三类技术,帮助大家对这三种技术有更清楚的了解。 PART1 技术原理 当样本中提取的蛋白酶解成肽段后,肽段经液相色谱分级分离后被电离带上电荷进入质量分析器中记录谱图信号。 (1)数据依赖型采集原理(Data-dependent Acquisition-DDA):首先带电荷的肽段(母离子)会经过全扫描得到一级谱图,随后质谱会根据参数的设定对一级质谱图中的肽段母离子按照信号强度由高到低进行选择(TOP20),送到碰撞室进行碎裂,然后扫描碎片离子得到对应肽段母离子的二级质谱图,通过搜库软件对原始文件进行分析,一方面对一级质谱图峰面积进行积分得定量信息,另一方面对二级质谱碎片进行解析得到蛋白质定性信息。DDA依赖于每次扫描的母离子丰度排序进行二级质谱分析,这种模式下,一些有价值的但丰度较低的母离子可能不会被选择进行碎裂检测,导致无法实现定性。 图2 DDA-Labelfree非标记蛋白质组学流程图 (2)数据非依赖采集(DIA)原理:样品在进入二级质谱时,按照设定的质荷比m/z范围分为若干个窗口,每个窗口内的一级母离子被分割在一起,二级质谱采集的是每个窗口中所有肽段母离子一起打碎后的碎片离子,换句话说DIA不依赖于一级母离子的丰度排序碎裂,而是将全部一级母离子进行二级碎裂,分窗口采集的模式使得到的二级碎片信息更丰富。因此,DIA数据更完整,检测深度高且重复性好,通过多个二级质谱峰面积进行定量,准确度高。 相较于DDA模式有损、随机,DIA具有全景式扫描、数据无遗漏、定量准确性高重复性好 图3 DDA和DIA采集模式直观比较(来源community.sciex.com) DIA蛋白质组学由于采集谱图复杂的问题,导致分析困难。常规方法解决方法主要是:DDA Library +DIA,需要将样本先形成Pool样本,进而DDA分级上机建立谱图库,基于谱图库去匹配正式上机检测样本中肽段信息,建库的意义在于提供了一个全面、无偏见的参考数据库,这有助于在后续的实验中更准确地鉴定和量化生物样本中的蛋白质,这也导致DIA分析需要额外的建库成本,同时也比较耗时。另一种解决DIA谱图解析困难的方法则是Library free DIA(Direct DIA)。 图4 常规建库DIA蛋白质组学流程 (3)Library free DIA(Direct DIA)原理:该原理和传统DIA数据扫描原理一样,传统DIA依赖于DDA建库数据,当建库数据中未采集到有效二级谱图时则影响实验结果。为了解决DIA谱图复杂且解析困难的问题,不依赖数据库的Library free DIA(Direct DIA)蛋白质组学方法应运而生,与传统DIA分析策略相比不进行DDA分级建库,而是直接利用深度机器学习算法实现,通过搜索特定物种数据库蛋白序列生成预测谱图库并和DIA上机实际样本原始文件谱图进行匹配打分,同时去除假阳结果。无需构建图谱库的Library free DIA(Direct DIA)策略,省去了建库成本和时间成本,同时保留了DIA可重复的定量的优势,可以说是蛋白质组学的潜力股。 图5 非建库DIA蛋白质组学流程 PART2 检测数量 那么,非标记蛋白质组学这三种方法检测结果如何?纵观已发表的蛋白质组学文献。首先我们发现数据非依赖型采集方法(DIA)检测数量普遍高于数据依赖型采集(DDA)。根据上期我们Astral蛋白质组学的测试数据显示Library free DIA(Direct DIA)检测数据比(DDA--Label free)高出10%左右,文献中也呈现类似规律[1-3]。其次关于建库的DIA和非建库的Library free DIA(Direct DIA)二者检测数量的比较,这二者从技术本身来考虑没有绝对的优劣,现阶段随着仪器的更新和质谱方法的开发以及机器学习的使用和优化,Library-free DIA和 DDA Library DIA的鉴定数量相差不大,近期的一些研究表明,Library free DIA(Direct DIA)检出数量高于建库Library DIA的检测方法检出的数量[4-5],也从侧面说明Library free DIA(Direct DIA)技术的应用将成为大势所趋。 图6 文献非标记方法比较 PART3 不同非标定量方法比较总结 目前Direct-DIA由于其定量准确度高、无偏采集模式、价格较低、数据结果好等方面的优势,在蛋白质组学中大受欢迎,可谓“后起之秀”。具体技术的选择建议根据应用目的,经费和结果等多方面考虑选择更合适的技术。 非标记定量蛋白质组学文献案例分享 案例一 酒精相关肝病的非侵入性蛋白标志物[6] Nature Medicine(IF82.9)-Noninvasive proteomic biomarkers for alcohol-related liver disease 2022年,马克斯·普朗克生物化学研究所的研究团队利用基于质谱的Library-DIA蛋白组学和机器学习模型进行人群大队列研究,确定了早期酒精相关肝病(ALD)中的纤维化、炎症和脂肪变性三种疾病非入侵性蛋白质组合生物标志物,发表了题为“Noninvasive proteomic biomarkers for alcohol-related liver disease”的文章,通过收集ALD患者(459例),健康对照(137例)和ALD独立验证队列(63例)人的肝脏和血液进行DIA蛋白组学研究,发现肝脏生理病例相关的肝脏和血浆均发生显著的蛋白质组重塑现象,阐述了蛋白质组合生物标志物在肝病临床诊断和预后评估中拥有巨大的潜力。不但反映出肝脏在疾病状态下的特定蛋白质表达模式,提供更为准确、敏感的诊断工具。此外,通过监测这些生物标志物的变化,可以预测疾病的进展和治疗效果,为肝病的个性化治疗提供有力支持。 案例二 多组学方法揭示两种冠状病毒的分子机制[7] Nature(IF 64.8)-Multilevel proteomics reveals host perturbations by SARS-CoV-2 and SARS -CoV 2021年,慕尼黑工业大学科研工作者发表了题为“Multilevel proteomics reveals host perturbations by SARS-CoV-2 and SARS-CoV”的研究性论文。该研究同时针对SARS-CoV-2和SARS-CoV两种冠状病毒进行研究,探索了SARS-CoV-2和SARS-CoV两种病毒的相互作用。同时,检测了1200多个肺源性人类细胞系受到病毒感染的转录组、Direct DIA蛋白质组、泛素组和磷酸化蛋白组的影响。通过研究病毒蛋白质与宿主蛋白质之间的相互作用以及修饰位点以及通路变化,成功发现了SARS-CoV-2的致病特性,并深入探讨了冠状病毒家族的潜在药物靶点,得以精确地引导候选药物的开发,为抗击SARS-CoV-2和其他冠状病毒疫情提供了更为有效的治疗方案。这不仅对于抑制SARS-CoV-2的传播具有至关重要的意义,也为未来冠状病毒的防治工作奠定了坚实的基础。 案例三 数据非依赖型采集模式(DIA)中数据分析工具的比较分析[5] Molecular & Cellular Proteomics(IF7.0)-A Comparative Analysis of Data Analysis Tools for Data-Independent Acquisition Mass Spectrometry 2023年西湖大学研究团队发表了题为“A Comparative Analysis of Data Analysis Tools for Data-Independent Acquisition Mass Spectrometry”的文章,从三种不同类型的质量分析仪(TripleTOF, Orbitrap, and TimsTOF Pro)中获得的六个DIA数据集,并评估了5种DIA分析软件工具(OpenSwath、Encyclopedia、Skyline、DIA-NN和Spectronaut)的性能。发现DIA-NN的性能显著优于其他工具,研究人员接下来评估了在特定工具中唯一识别的多肽和蛋白质。比较发现当谱库的可比性有限时,无谱库方法优于基于谱库的方法。此研究为 DIA 数据分析工具提供了全面指导。 派森诺生物科技有限公司提供全面的蛋白组分析服务,旨在通过尖端的技术手段,为科研工作者深度解析蛋白质动态。我们诚挚邀请所有对精准定量蛋白质组学与功能分析感兴趣的老师们,共同探索生命奥秘,解锁科学新知。期待您的垂询,携手共创科研新篇章! 参考文献 [1]Mehta D , Scandola S , Uhrig R G .Library-free BoxCarDIA solves the missing value problem in label-free quantitative proteomics[J].Cold Spring Harbor Laboratory, 2021. DOI:10.1101/2020.11.07.372276. [2]Guzman UH, Martinez-Val A, Ye Z, et al. Ultra-fast label-free quantification and comprehensive proteome coverage with narrow-window data-independent acquisition. Nat Biotechnol. Published online February 1, 2024. DOI:10.1038/s41587-023-02099-7. [3]Kverneland AH, Østergaard O, Emdal KB, Svane IM, Olsen JV. Differential ultracentrifugation enables deep plasma proteomics through enrichment of extracellular vesicles. Proteomics. 2023;23(7-8):e2200039. DOI:10.1002/pmic.202200039. [4]Reilly L, Lara E, Ramos D, et al. A fully automated FAIMS-DIA mass spectrometry-based proteomic pipeline. Cell Rep Methods. 2023;3(10):100593. doi:10.1016/j.crmeth.2023.100593 [5]Zhang F, Ge W, Huang L, et al. A Comparative Analysis of Data Analysis Tools for Data-Independent Acquisition Mass Spectrometry. Mol Cell Proteomics. 2023;22(9):100623. DOI:10.1016/j.mcpro.2023.100623. [6]Niu L, Thiele M, Geyer PE, et al. Noninvasive proteomic biomarkers for alcohol-related liver disease. Nat Med. 2022;28(6):1277-1287. DOI:10.1038/s41591-022-01850-y. [7]Stukalov A, Girault V, Grass V, et al. Multilevel proteomics reveals host perturbations by SARS-CoV-2 and SARS-CoV. Nature. 2021;594(7862):246-252. DOI:10.1038/s41586-021-03493-4.