2024-06-11

单细胞测序以其庞大的数据量和精准到细胞级的高分辨率而备受瞩目。这一现象级的测序技术不仅在医学领域爆火,也逐渐走入农口。但是不同于人或者小鼠的样本,植物的样本面临着解离细胞困难,后续分析参考的数据库少,基因组比对率低等困难,尤其是在鉴定细胞类型时常常因为找不到合适的marker基因而头疼。
小编最近发现了一个非常好用的植物单细胞分析网站Plant Single Cell Hub,该网站是由华中农业大学植物科学技术学院金双侠教授课题组研发的植物单细胞分析一站式网站,网站链接http://jinlab.hzau.edu.cn/PsctH/。该网站一直在维护更新,功能也在不断的完善。其中不仅包含了植物细胞解离的方法、单细胞数据分析的脚本,还提供了用于细胞鉴定的marker基因,真是保姆级网站了!
下面就由小编来介绍一下这个网站正确的打开方法吧!
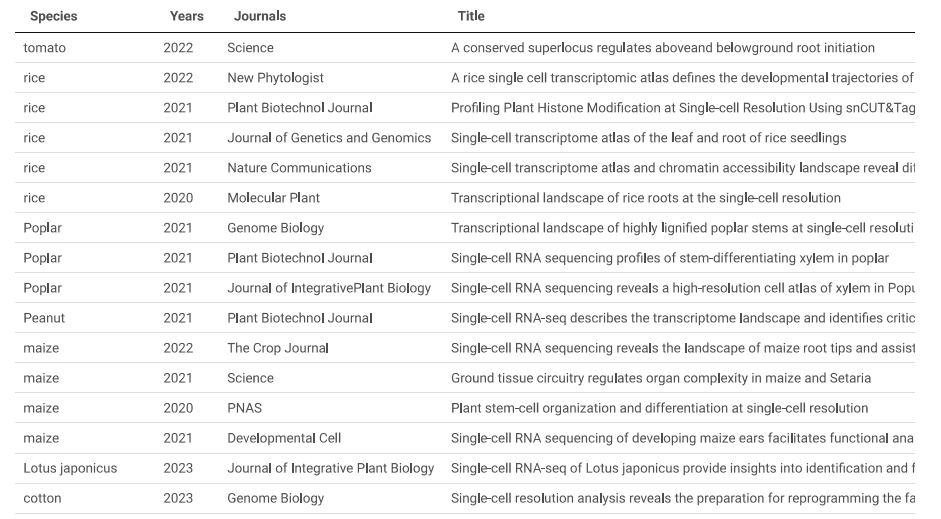
1.收录文献简介 在项目刚开题时,许多小伙伴可能都会遇到一个头疼的问题,那就是缺乏思路。这时候,我们通常需要阅读大量的相关文献。但是对于植物单细胞领域而言,相关文献相对较少,而且找起来也会耗费我们宝贵的时间。该网站收集了从2019年到2024年的植物(部分物种)单细胞文章,而且可以通过筛选物种、年份、关键词等,可以快速的查看相关的文献,帮助我们快速打开思路。 图1:收录文献数量柱状图 图2:文献概观图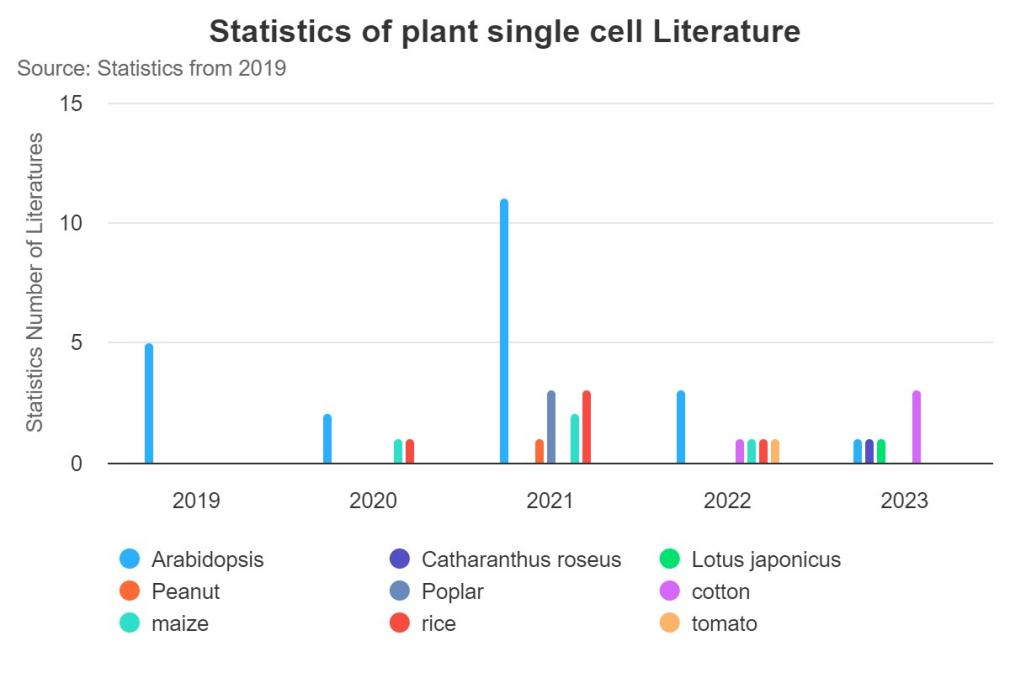
2.组织解离方法 植物细胞的结构与动物细胞不同,解离方法也有所不同。这个网站提供了一种基于原生质体的解离方法。网站提供的方法可以帮助我们了解植物样本解离的步骤。由于不同组织部分的结构差异较大,我们仍需根据自己的样本情况来设计和调整实验流程。 图3:实验流程示意图
3.一站式脚本生成 拿到测序公司的原始矩阵信息,该头疼就是如何构建分析流程了,特别是对于一些代码能力比较弱的小伙伴,该网站提供了一种非常对用户友好的脚本生成方法,如下图所示,你可以通过拖动滑块来自定义设置细胞过滤阈值、归一化方法、降维算法、marker基因阈值等多个参数,最后点击下载按钮,网站将生成并下载相应的R代码。如果你还没有构建自己的分析环境,该网站也给出了一种基于conda的分析环境构建方法,实在是太贴心了! 图4:分析参数配置图
4.marker gene 数据库 众所周知,单细胞数据的分群结果是建立在数学逻辑上的,缺乏生物学意义。在鉴定每个群的细胞类型时,我们依赖已有的细胞类型特异表达的marker基因。因此,准确的marker基因是确定细胞类型的关键。该网站提供了不同物种、不同组织的marker基因列表。这些marker基因经过了RNA原位杂交或GFP报告基因表达的验证,准确性较高。 图5:marker基因数据库 如果有小伙伴还没有自己的数据,但渴望练练手又找不到合适的数据,也不用担心。该网站提供了多个不同植物物种的单细胞数据供我们下载使用。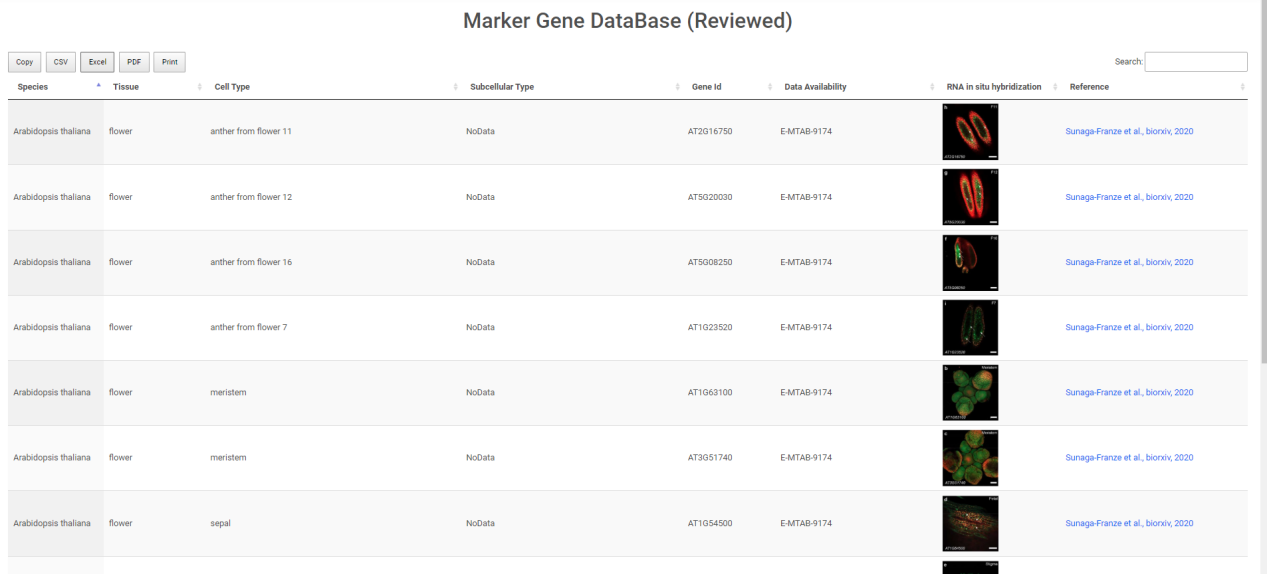
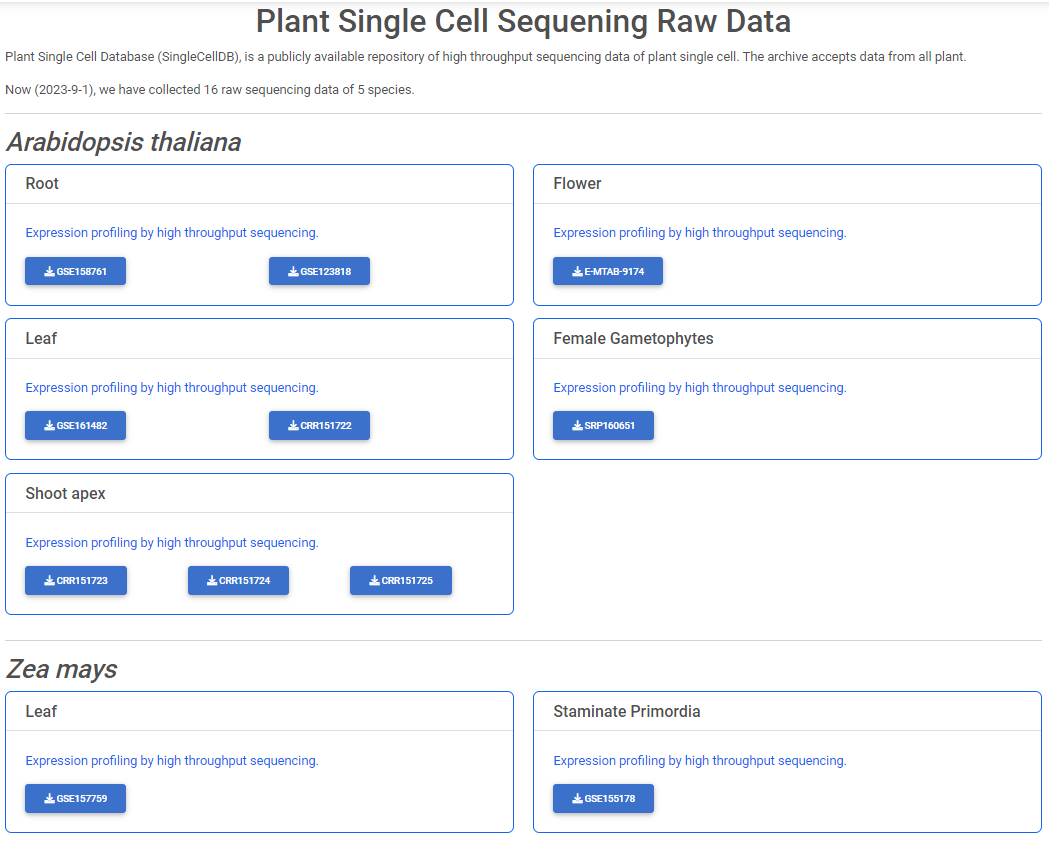
5.结 论 看完介绍,相信大家都会觉得这个网站虽小却五脏俱全。最让我惊喜的是,该网站所有的marker基因都经过了生物学验证。对于科研工作来说,准确性永远是至关重要的。希望我的介绍能给面对课题无从下手的小伙伴们一些帮助! 参考文献 Xu, Z., Wang, Q., Zhu, X., Wang, G., Qin, Y., Ding, F., … & Jin, S. (2021). Plant single cell transcriptome hub (pscth): an integrated online tool to explore the plant single‐cell transcriptome landscape. Plant Biotechnology Journal, 20(1), 10-12. https://doi.org/10.1111/pbi.13725


