2024-07-12

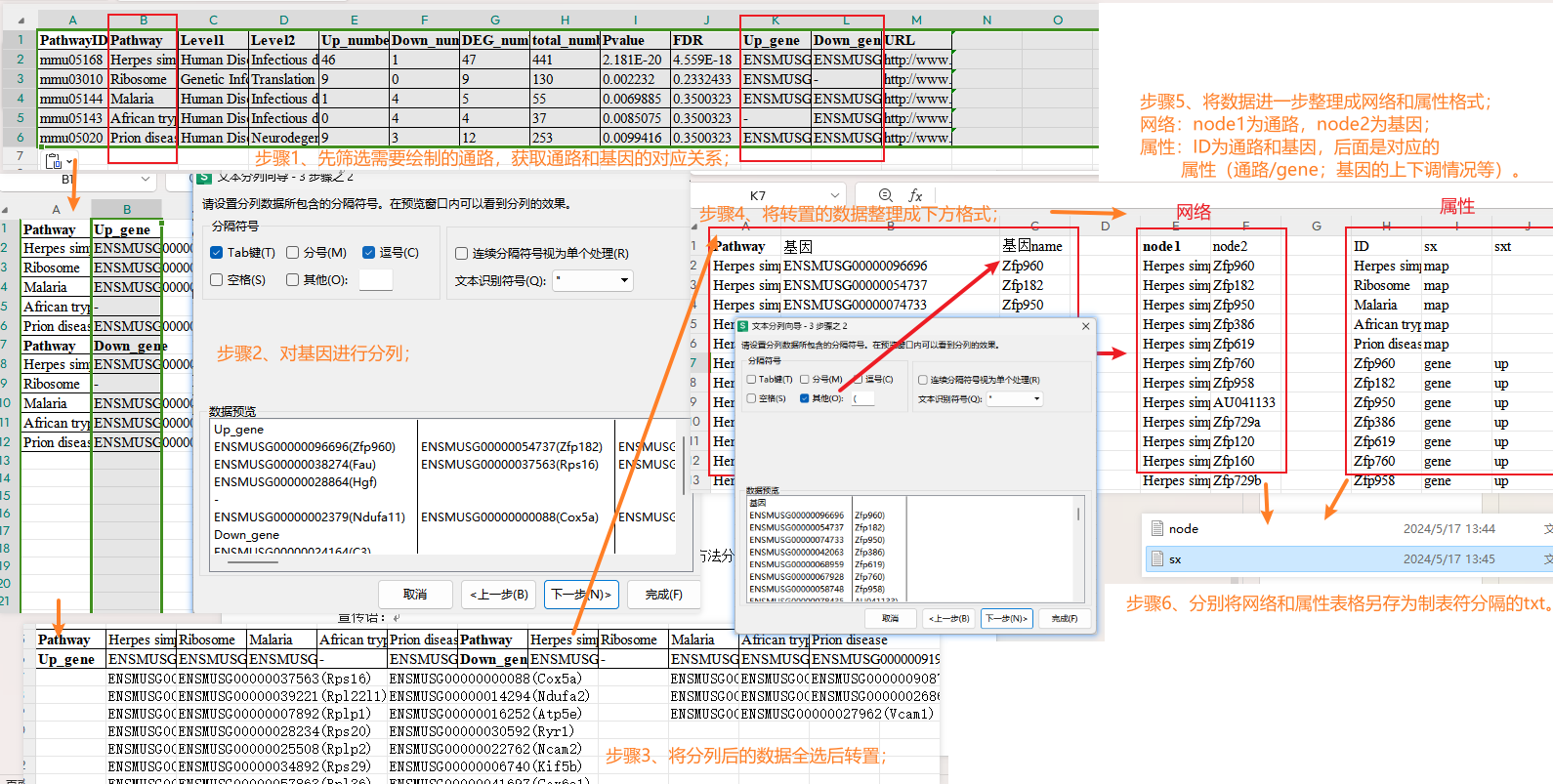
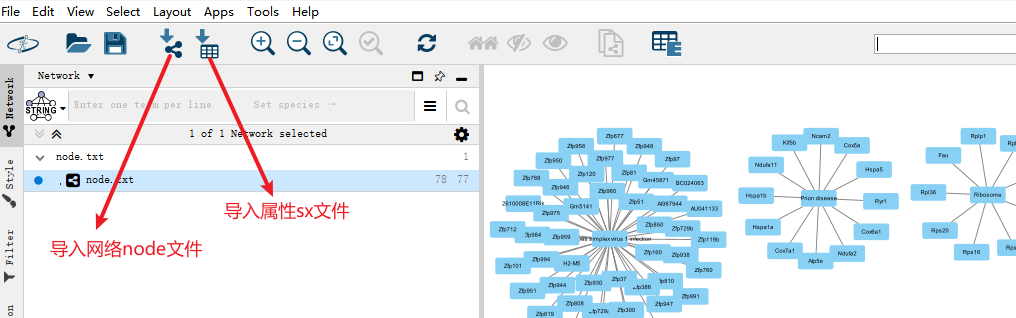
上两次(【干货】如何使用cytoscape玩转网络图?(一);【干货】如何使用cytoscape玩转网络图?(二))带大家了解在cytoscape上PPI网络的绘制、参数的调整、以及MCODE、cytoHubba、iRegulon插件的使用。 由于软件的更新,插件获取方法有了改变。今天小派再给大家来介绍插件的获取以及其它几个网络图的绘制,助大家都能够玩转cytoscape。 七、插件获取 cytoscape软件更新后(3_10后面版本)Apps里的App Manager...替换为了App Store。在App Store里可以方便的查看已安装的插件,以及对插件进行更新,但新插件的获取,需要链接到网站(App网站:https://apps.cytoscape.org/)下载安装。备注:网站可能需要科学上网才可以下载app。 cytoscape软件旧版(3_9_1)、新版(3_10_2)、java(jie)以及之前软文提到的MCODE、cytoHubba、iRegulon插件获取如下: 网盘链接:https://pan.baidu.com/s/1Jf-OgrzaCulGlA04PO-Ngg?pwd=3ub5 提取码:3ub5 cytoscape官网下载:https://cytoscape.org/ java官网下载:https://www.java.com/zh-CN/ 从App网站下载需要的插件后,通过cytoscape软件的Apps → App Store → Install Apps Form File安装即可。 八、通路网络图 做完组学分析后,可以得到比较组的富集结果,在富集结果中可能有些通路会是我们关注的重点,除了用热图展示通路里差异(基因/蛋白/代谢物)的表达情况,还可以使用通路网络图来展示差异的归属情况。 以下示例以转录组为参考,其它组学同理: 1、首先基于富集结果选择trem/通路(可以是显著性top的,也可以是自己关注的),此次以显著性top5通路为例; 2、选择需要绘制的通路和基因,整理成网络文件和属性文件(网络文件为通路和基因的对应关系,网络图里也可以加上作用对的描述;属性文件为node节点的描述信息),步骤参考下图; 3、打开cytoscape,导入网络和属性文件,然后对网络进行调整; 4、在style和Layout里调整网络(节点性状大小按照通路和基因设置,改变字体大小等)后,可以导出图片。 备注:代谢物/蛋白-通路、基因-通路-代谢物、代谢-基因-通路+PPI等网络都可以如此绘制。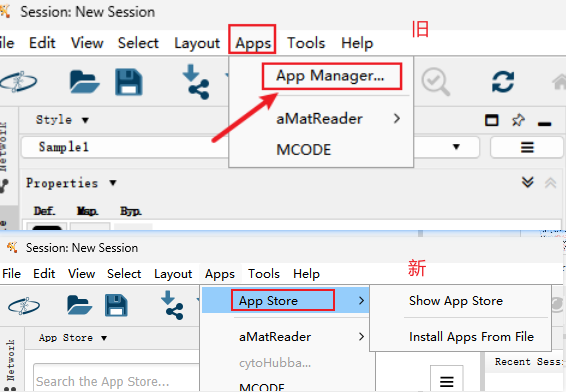


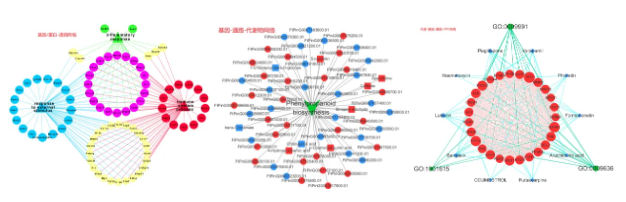
九、相关性网络图 做完组学后,我们会得到基因/蛋白/代谢物的表达量数据,在单个组学中我们能够筛到差异的结果,进一步我们可以进行关联分析。通过相关性分析,可以得到差异基因/蛋白/代谢物之间的关联性,或者得到其和表型指标的关联性,随后根据关联情况构建相关性网络。 以下示例以转录和代谢为例: 1、在我们这里做完联合分析后,在04文件夹里会得到基因和代谢物的相关性矩阵1(Cor),以及相关性系数在0.8以上的矩阵(mRNA_mete_diffrence_log2_dotplot)。由于差异分组中基因和代谢物的相关性较多,因此在标准分析中网络图我们选取相关性结果前100个关系对进行作图。有些时候老师关注的代谢物或基因不在网络图中,这样的话,我们可以基因这个矩阵2筛选我们关注的基因和代谢物,再绘制相关性网络图; 备注:其它组学也一样,需要得到相关性矩阵后,绘制网络图。相关性矩阵可以用一些在线小工具或R的cor()函数等得到,一般输入文件为2个表达矩阵(基因/蛋白/代谢物在样品里的表达矩阵,表型在样品里的数值矩阵,且样品需要一一对应)。 2、在筛选的时候,可以先整理个关注的基因和代谢物的列表,用excel的vlookup函数筛选,保留基因列和代谢物列都是关注的作用对,将基因和代谢物列分别作为node1和node2,cor和p值可以作为网络的筛选文件,另存为制表符分隔的txt作为网络文件,然后对基因和代谢物整理属性文件; 备注:基因ID如果需要用基因名字替换的话,也可以用vlookup查找替换。vlookup使用可以参考:https://mp.weixin.qq.com/s/KxxtA9rR1P_mV3Q6-aslEQ 3、打开cytoscape,导入网络和属性文件,然后可以在style和Layout里对网络进行调整(根据属性区分节点,根据相关性系数展示连线的颜色等),完成后可以导出图片。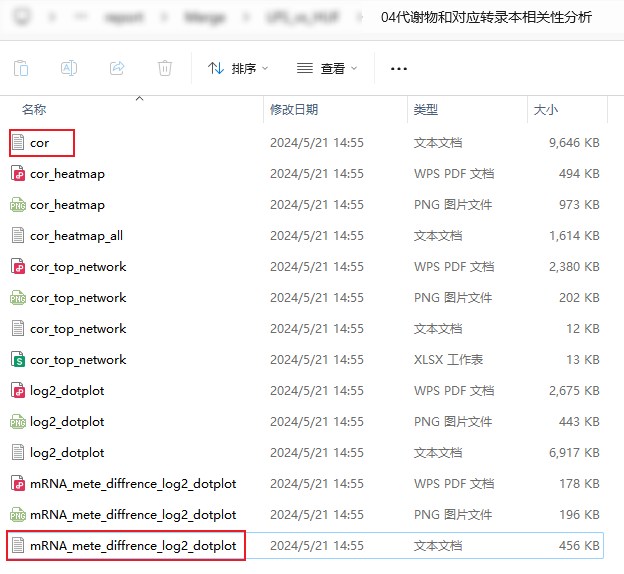
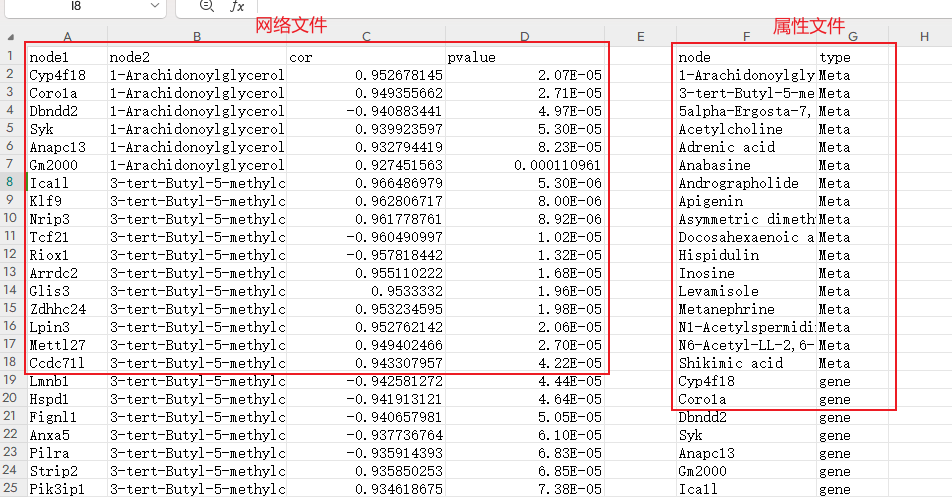
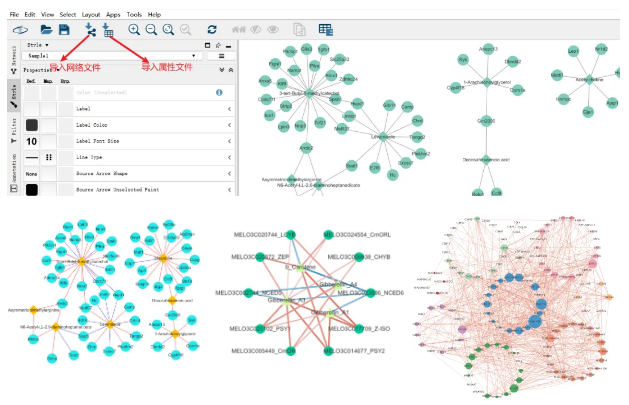
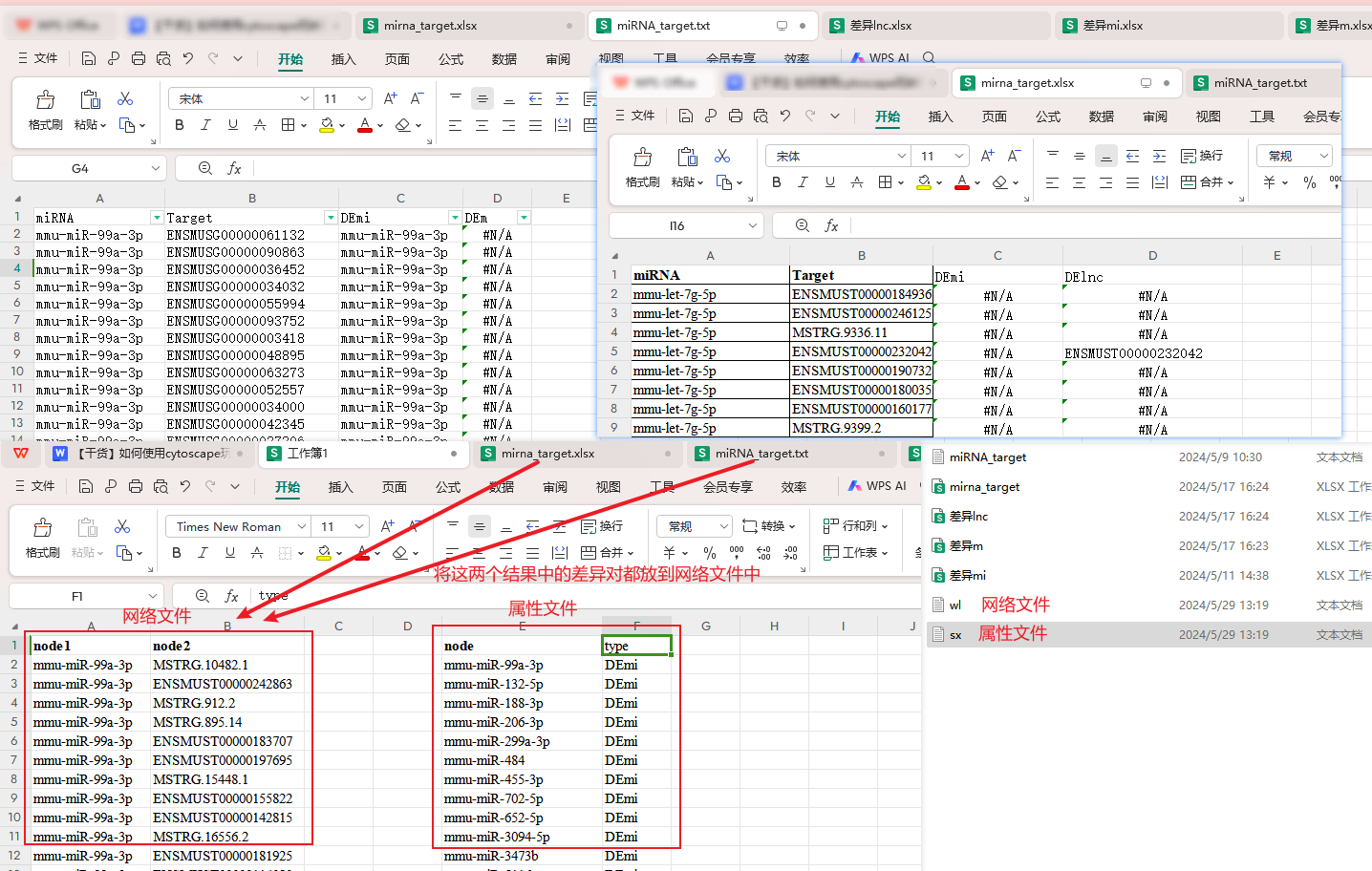
十、RNA调控网络图 在生物体内除了作为遗传信息传递的mRNA外,还有多种不同类型的RNA(lncRNA、miRNA等)参与复杂的基因表达调控网络。为了更全面的获得不同处理下,生物体内的RNA表达情况,我们可以通过全转录组测序、mRNA+miRNA测序、lncRNA+miRNA测序等技术,同时获得样品里多种不同RNA的表达及差异情况。根据不同RNA之间的靶向关系,我们可以进一步构建差异RNA调控网络。 以下示例以lncRNA+miRNA测序为例: 1、在lncRNA测序结果中有lncRNA和miRNA互作分析(结果为:report→result→LncRNA→6_miRNA_LncRNA→mirna_target表),在miRNA测序结果中有miRNA的靶基因分析(结果为:report→result→miRNA→4_miRNA_target→miRNA_target.txt压缩包里的表); 2、然后在两个表中可以通过vlookup将比较组的差异结果查找过来,然后将差异的作用对拿出来构建网络文件,以及可以将这些RNA的类别和上下调作为属性文件; 备注:可以筛选mi-lnc和mi-m里共有差异miRNA的互作对;可以将lnc和m的靶向也加进去;还可以筛选关注的RNA的作用对等。 3、打开cytoscape,导入网络和属性文件,然后可以在style和Layout里对网络进行调整,完成后可以导出图片。
![]()

小派用了三期来介绍cytoscape,希望大家都能有所收获哈!!


