2024-07-19

细胞之间的通讯是细胞相互协同完成复杂生命活动的重要方式。复杂的细胞外反应始于配体与其同源受体的结合和特定细胞信号通路的激活。所以绘制这些配体-受体相互作用是理解细胞行为和对邻近细胞反应的基础。随着单细胞测序技术的快速发展,目前已有多种工具能够做到在单细胞水平上系统地解析细胞间通信网络及功能。而对于我们科研人员来说,如何准确且优美的找到适合自己的工具及展现形式是十分重要的,接下来我会对目前最常用的几种方法进行介绍。
『一』、CellPhoneDB[1]
首先,就是大名鼎鼎的CellPhonedb啦。CellPhoneDB v1于2018年发表在Nature,升级后的v2发表在Nature Protocols, 灰常权威。
CellPhoneDB的主要优势在于:
1.分析思路:计算配体-受体相互作用时考虑了配体和受体的结构组成
2.数据库特点:CellPhoneDB使用了集成于UniProt、Ensembl、the IMEx consortium、PDB、IUPHAR等的受配体数据库,共存储了包括501种分泌蛋白和585种膜蛋白的978种蛋白质,共参与了1396种相互作用(最新的数据库好像有接近3000个互作对了https://github.com/ventolab/CellphoneDB),其中共有466种相互作用涉及了异聚体。
3.分析方式及可视化:该流程基于python,可以快速处理数据,另外还有网页版CellPhonedb可以使用。下游的输出结果也可以对接使用ktplots(R包,我们之前有介绍过[2]),cytoscape等进行美化。
4.引用量高,被广泛认可
但是也有局限性:
软件自身可视化形式有限,依赖其他工具提供更丰富的展现形式
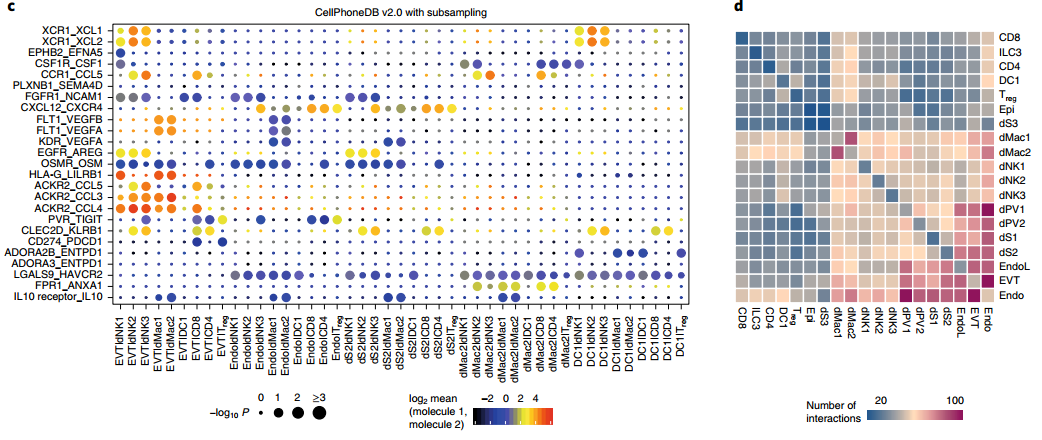
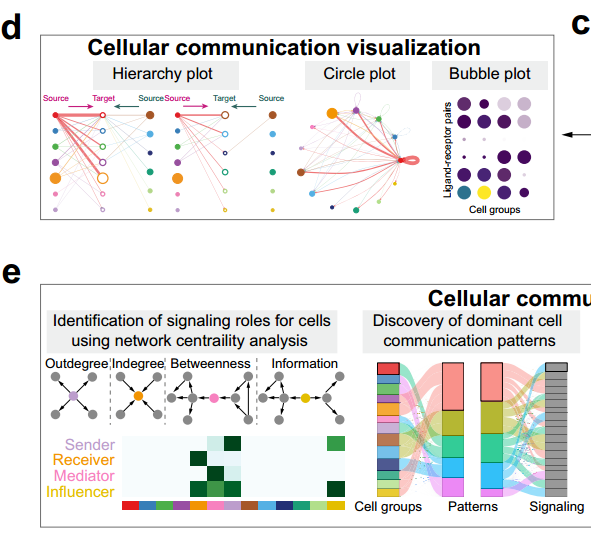
『二』、CellChat[3]
CellChat于2021年发表于Nature Communication,同样经历了两个版本的更迭,目前为v2,拥有丰富的展现形式,也同样广受好评。
CellChat的主要优势在于:
1.分析思路:计算配体-受体相互作用时不仅考虑了配体和受体的结构组成,还考虑了辅因子以模拟细胞通讯。
2.数据库特点:CellChatDB v2 包含约 3,300 个经过验证的分子相互作用,并将配体-受体对分类为包括:分泌信号(Secreted Signaling)、细胞外基质-受体(ECM-Receptor)、细胞-细胞接触(Cell-Cell Contact)和非蛋白质信号(Non-protein Signaling在内的不同类型。
3.分析方式及可视化:分析所用的包基于R语言,不同于CellPhoneDB的可拓展性,cellchat自身即封装了丰富的可视化函数,丰富的图片类型可以一站式解决您的可视化需求。
局限性:
产生的受配体相互作用较少
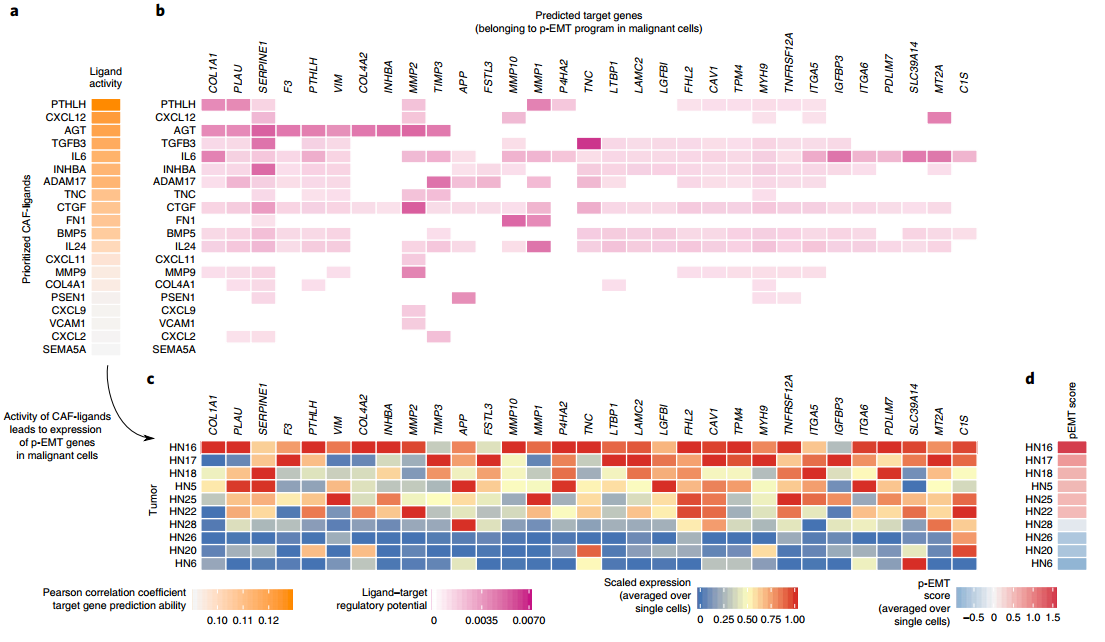
『三』、NicheNet[4]
NicheNet于2020年发表于Nature Methods,同样足够权威。
NicheNet的主要优势在于:
1.分析思路:不同于CellPhoneDB和Cellchat,NicheNet计算配体-受体相互作用时把配体,受体,靶基因联合起来进行分析,即进一步考虑了信号通路也就是受体被激活后的信号传导与下游靶基因.
2.数据库特点:整合了信号通路,转录因子等数据库,具有特色且考虑全面。
3.分析方式及可视化:分析所用的包基于R语言,分析内容扩展到下游信号通路等,可视化内容丰富。
缺点在于:
没有考虑受配体的结构组成
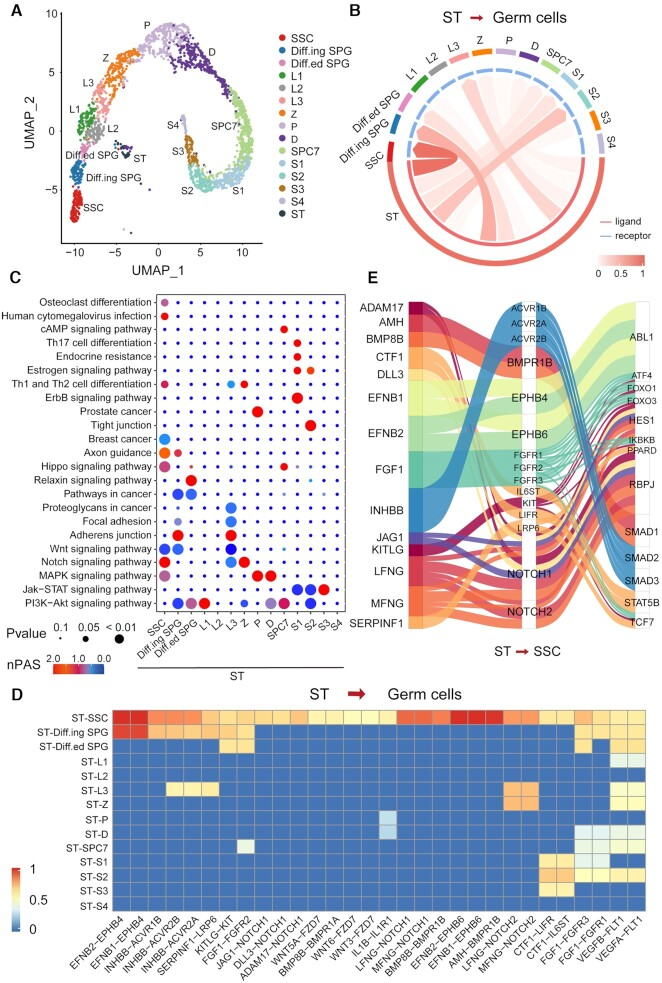
『四』、CellCall[5]
CellCall于2021年发表于Nucleic Acids Research,同样可靠。
CellCall的主要优势在于:
1.分析思路:计算配体-受体相互作用时考虑配体-受体-转录因子的先验知识,通过结合配体/受体的表达和某些 L-R 对的下游 TF 活动来推断细胞间通讯
2.数据库特点:CellCall所使用的是基于KEGG通路收集配体-受体-转录因子 (L-R-TF) 轴数据集
3.分析方式及可视化:分析所用的包基于R语言,可包含通路活性分析,可以分析某两种细胞之间通讯导致的受体细胞通路变化,且内部函数丰富,可视化形式多样
缺点在于:
由于算法和数据库的原因,能够检测部分非特定通讯,但可能会错过低强度通讯。
综上所述
除此之外,还有ITALK等方法,细胞通讯分析软件众多,食无定味,适口者珍,各位老师可以参考包括此文在内的众多知识分享来选择最适合自己的方法进行自己的课题分析,祝老师的CNS发表顺利!!!
参考链接:
1.CellPhoneDB: inferring cell-cell communication from combined expression of multi-subunit ligand-receptor complexes. Nat Protoc.
2.https://mp.weixin.qq.com/s/f-YJ5YIp26EdnyC52gpBDQ?from=industrynews&version=4.1.26.6024&platform=win&nwr_flag=1#wechat_redirect
3.Inference and analysis of cell-cell communication using CellChat. Nat Commun.
4.NicheNet: modeling intercellular communication by linking ligands to target genes. Nat Methods.
5.CellCall: integrating paired ligand-receptor and transcription factor activities for cell-cell communication. Nucleic Acids Res.


