
2024-07-20

转录是基因表达的关键调控步骤,是一个内在的不连续动态过程,其调控涉及核心启动子元件、转录因子和增强子,具有短爆发和长沉默期的特点。增强子是在发育和疾病过程中对基因调控和细胞识别控制至关重要的远端基因组元件,增强子在转录过程中会转录表达非编码增强子RNA(eRNA),越来越多的研究表明eRNA在基因调控中发挥重要作用,而且eRNA对细胞类型和状态具有高度特异性,可以通过研究eRNA转录表达而将其作为诊断标志物或治疗靶点,因此研究增强子对于解析细胞周期、发育和疾病期间的转录调控机制至关重要。 新生RNA(nascent RNA)是指正在转录的RNA分子,通过新生RNA研究基因的实时转录活动状态,揭示转录调控机制。新生RNA不仅具有半衰期短、丰度低的特征,而且不具有Poly(A)尾,只能从丰富的总细胞 RNA 中选择性标记和富集新生RNA。目前常规单细胞测序虽然能研究细胞之间异质性,但平均了单个细胞的不连续转录,无法捕获活跃的转录过程,使得在单细胞分辨率下对转录动力学和研究增强子-基因关系具有一定挑战性。 2024年6月5日,来自麻省理工学院的Phillip A. Sharp研究团队在Nature期刊上发表一篇Single-cell nascent RNA sequencing unveils coordinated global transcription的研究文章,文中介绍了一种新型单细胞新生RNA测序方法(scGRO-seq),通过点击化学(click chemistry)定量评估单个细胞的全基因组新生转录活动。作者将scGRO-seq应用于小鼠胚胎干细胞,通过分析获得细胞基因和增强子转录动态的全面视图,量化了转录爆发动力学和难以检测特异性基因的细胞周期期间的转录动力学,同时研究发现在增强子开始转录激活时间早于相关基因转录激活。总而言之,scGRO-seq可以填补转录时间控制以及增强子-基因相关功能研究空白,同时为深入探究细胞周期、发育和疾病中的基因调控机制提供新平台。 接下来,带大家解析这篇文章:
一、技术原理
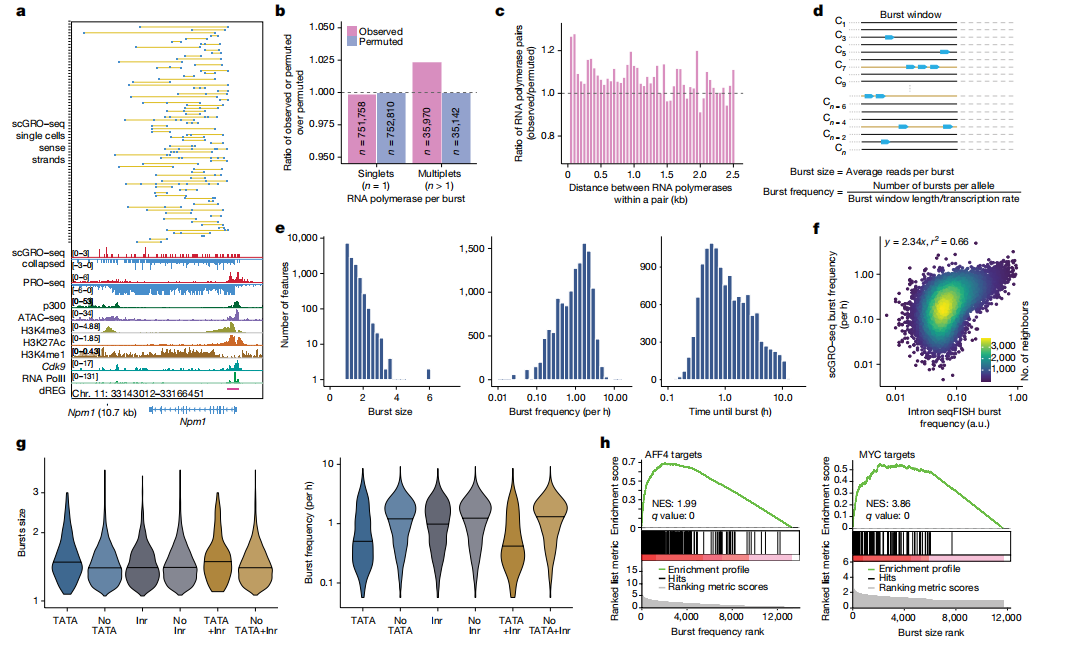
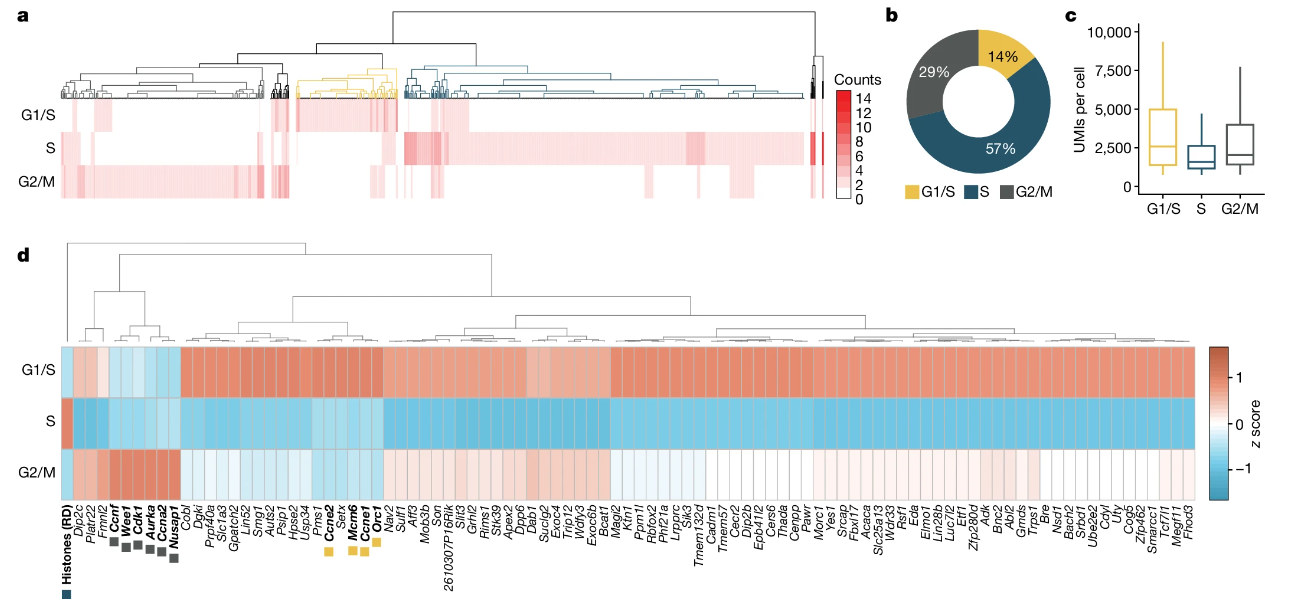
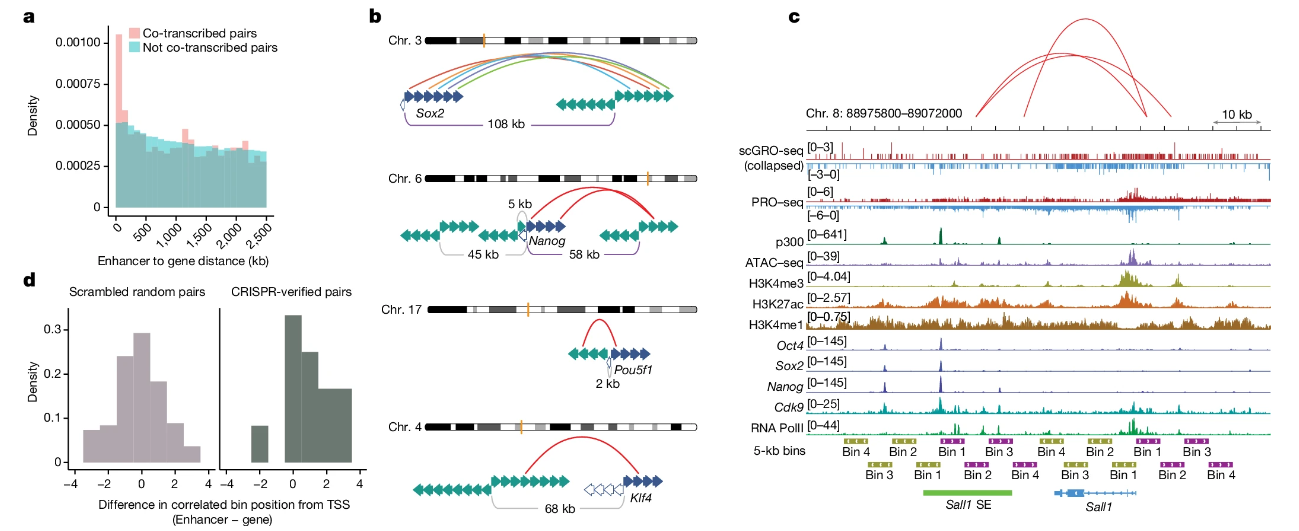
首先,作者开发了一种利用点击化学(AGTuC)的全基因组转录组测序方法,是一种基于细胞群的新生RNA测序方法,通过3′-(O-propargyl)-NTPs选择性标记新生RNA,偶联5′端带有AzScBc基团的DNA PCR handle,逆转录RNA-DNA偶联物并制备NGS文库。为了能够将该方法更好的应用于单细胞测序,作者进一步优化AGTuC实验条件,在标记过程中不会破坏核膜完整性,称为完整核AGTuC(inAGTuC)。作者利用inAGTuC测试了不同细胞核投入量下捕获新生RNA的效率并与PRO-seq进行相关性比较,结果发现 AGTuC 和 inAGTuC具有更高的效率、更低的成本、更短的文库制备时间和更低的样品起始量,是现有方法(PRO-seq)的可行替代方案,同时为单细胞新生 RNA 测序奠定了基础。 在inAGTuC的基础上,作者开发了新的化学物质应用于单细胞新生RNA测序,将其命名为scGRO-seq。首先分离细胞核,加入3′-(O-propargyl)-nucleotides进行核run-on反应,将附有3′标记的新生RNA完整细胞核通过流式分选至含有尿素的96孔板中(尿素可以裂解核膜并使 RNA 聚合酶变性并释放标记的新生 RNA),将带有5′-AzScBc基团的DNA接头(包括位于5'-AzScBc 、10-12bp单细胞barcode序列、4-6bp UMI序列和 PCR handle)和CuAAC MIX加入孔板中,在CuAAC的作用下,炔丙基标记的新生RNA与每个孔中唯一的5′-AzScBc DNA连接形成RNA-DNA偶联物,将孔板中RNA合并,加入TSO序列通过M-MuLV逆转录酶进行反转录,结束后通过Cas9去除空接头,最后进行扩增和测序。 图1 scGRO-seq流程图与验证 scGRO-seq直接测定转录爆发动力学 与传统荧光原位杂交检测转录动力学的方法不同,scGRO-seq能够以单核苷酸分辨率检测全基因组正在转录的RNA聚合酶,能够观察到RNA聚合酶在基因上的分布,反映了转录的动态过程(图2a)。为了验证scGRO-seq能够揭示转录爆发的过程,作者设计了一个无偏倚的模型,量化转录RNA聚合酶的发生频率,结果发现,在真实数据中出现更高的多聚体,表示多个RNA聚合酶同时转录(图2b),同时分析了多聚体之间的距离分布发现,真实数据中多聚体具有更紧密的RNA聚合酶分布(图2c),进一步说明了scGRO-seq能够捕获到转录爆发现象。作者通过scGRO-seq数据直接测定了转录爆发的动力学参数(图2d-e),比较发现scGRO-seq与内含子 seqFISH所得爆发频率数据具有良好相关性(图2f),同时发现核心启动子元件和转录因子可以调节转录爆发参数(图2g-h)。 综上所述,scGRO-seq能够直接和全面地观察转录爆发,有助于在单细胞水平上研究转录动力学。 图2 scGRO-seq推断转录动力学 scGRO-seq推断组蛋白基因的细胞周期 目前,用于确定的细胞周期状态的传统scRNA-seq方法存在一定局限性,首先是大多数scRNA-seq依赖于poly(A)尾进行捕获测序,因此无法检测到复制依赖性组蛋白基因(在S期专门转录的最佳细胞周期期特异性基因,无poly(A)尾);其次,这些基因高度特异表达于S期,在前期样本制备时可能存在一定滞后性,最终导致难以及时检测到特异性基因表达。而scGRO-seq能够解决传统scRNA-seq存在的问题,作者利用scGRO-seq对小鼠胚胎干细胞进行实验,结果发现,scGRO-seq检测到组蛋白位点体中复制依赖性组蛋白基因的活性转录,并且可对S 期细胞进行分类(图3a),通过不同细胞周期期特异性基因表达的分层聚类揭示单个细胞的三个重要簇,三个簇具有不同的细胞比例代表细胞不同周期阶段的长度(图3b),不同细胞周期转录水平存在差异(图3c)。对细胞周期阶段差异表达基因的分析发现,细胞在DNA复制过程中虽然转录水平降低,但会持续进行,不同基因存在不同转录循环模式(图3d)。 综上所述,scGRO-seq能够揭示整个细胞周期的动态转录程序。 图3 scGRO-seq推断组蛋白基因的细胞周期 相互依赖基因的共转录 作者利用scGRO-seq探究功能相关基因是否在稳态下进行共转录。通过可视化方法直观地展示了两个基因在单细胞水平上的共转录关系,表明这两个基因可能在时间上或功能上有某种联系,比如可能受到相同的转录因子调控,或者参与相同的生物学过程(图4a)。作者对转录过程中共表达的基因对进行分析发现了共转录基因对在细胞周期调控、RNA剪接、翻译控制等生物学过程中富集(图4b)。作者将scGRO-seq获得的共转录基因对与内含子seqFISH比较共转录相关性,结果发现两者具有高度相似的共转录谱(图4c)。 综上所述,具有高通量优势的scGRO-seq能够直接检测基因对或基因网络之间的转录协调性,是研究基因组功能的重要工具。 图4 功能相关基因的协调转录 增强子-基因时间协调 作者利用scGRO-seq探究基因和增强子之间的时间协调性。作者分析了基因前 10 kb 内的 scGRO-seq读段以及增强子周围向外转录的每条链上至少 3 kb 的读段,结果发现增强子-基因共转录主要在 200 kb 内显着富集(图5a),同一条染色体上的功能相关基因聚集在一起时,多个增强子与每个基因相关,可能是细胞周期调控的进一步作用所导致结果(图5b)。为了验证增强子的转录可能在其靶基因启动子转录之前开始的假设,作者通过CRISPR干扰验证多个增强子bin与该基因之间存在相关性(图5c),同时发现相关的增强子bin通常比基因bin距离其 TSS 更远,这意味着增强子转录可能在启动子转录之前开始(图5d)。 综上所述,scGRO-seq初步验证了增强子转录可能在启动子转录之前开始,但该时间模式对增强子-基因转录调控的影响需进一步深入探究。 图5 基因与增强子之间的时间协调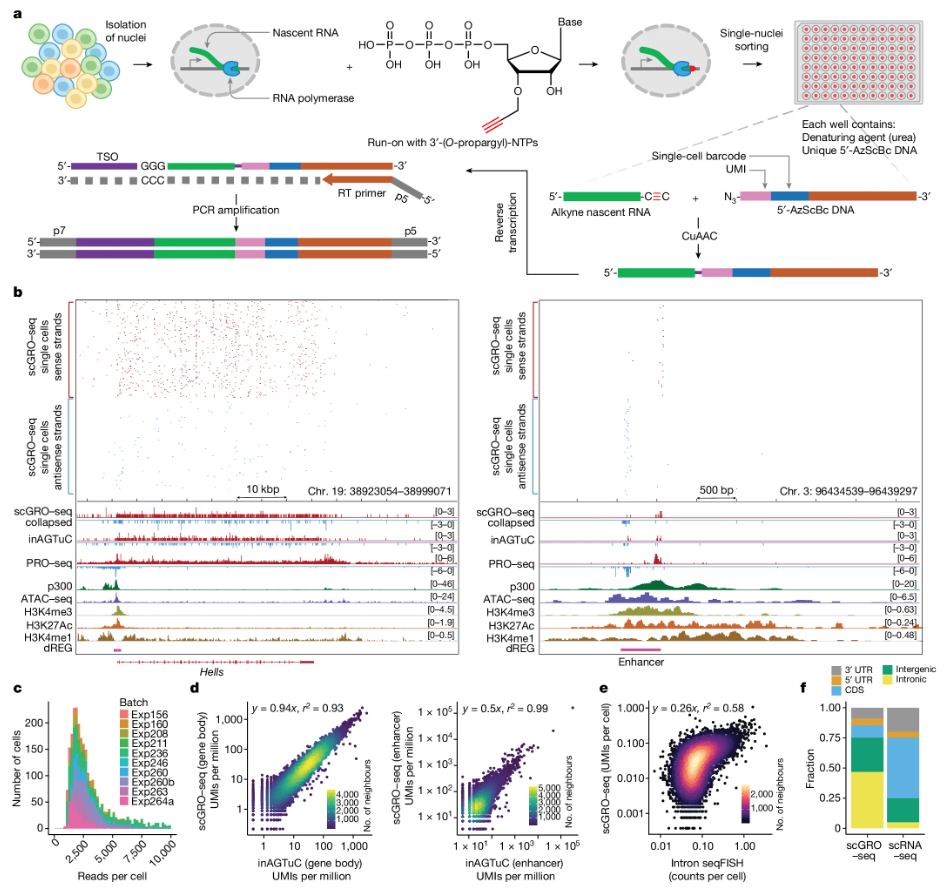

二、结 论
综上所述,scGRO-seq可以不依赖于Poly(A)尾检测新生RNA,通过CuAAC可以捕获约10%的新生RNA,作者通过点击化学取代了多轮新生的RNA纯化和核酸连接,优化了实验步骤,增加高通量液滴封装捕获效率,扩展了scGRO-seq的适用性。 scGRO-seq本质上是多模态的,是一个可以深入详细地理解转录调控的工具,它可以在天然环境中评估共转录和预测增强子-基因调控网络,文中作者已验证scGRO-seq可以确定表达基因的突发大小和频率、细胞周期阶段的转录动力学以及全基因组基因-基因和增强子-基因共转录检测等功能。 但目前的scGRO-seq仍具有一定局限性。通过低sarkosyl浓度实现的核完整性的保持未能促进暂停复合物中RNA聚合酶的运行,从而限制了启动子-近端暂停聚合酶的检测。在读取深度和细胞数量方面限制了对基因-基因和增强子-基因对的爆发动力学和共转录的分析。但尽管存在这些局限性,scGRO-seq的适应性和效率为研究不同生物学背景下的转录动力学和调控机制提供了新的途径,丰富了对基因表达调控及其在生理和病理条件下的影响的理解。