2024-08-01

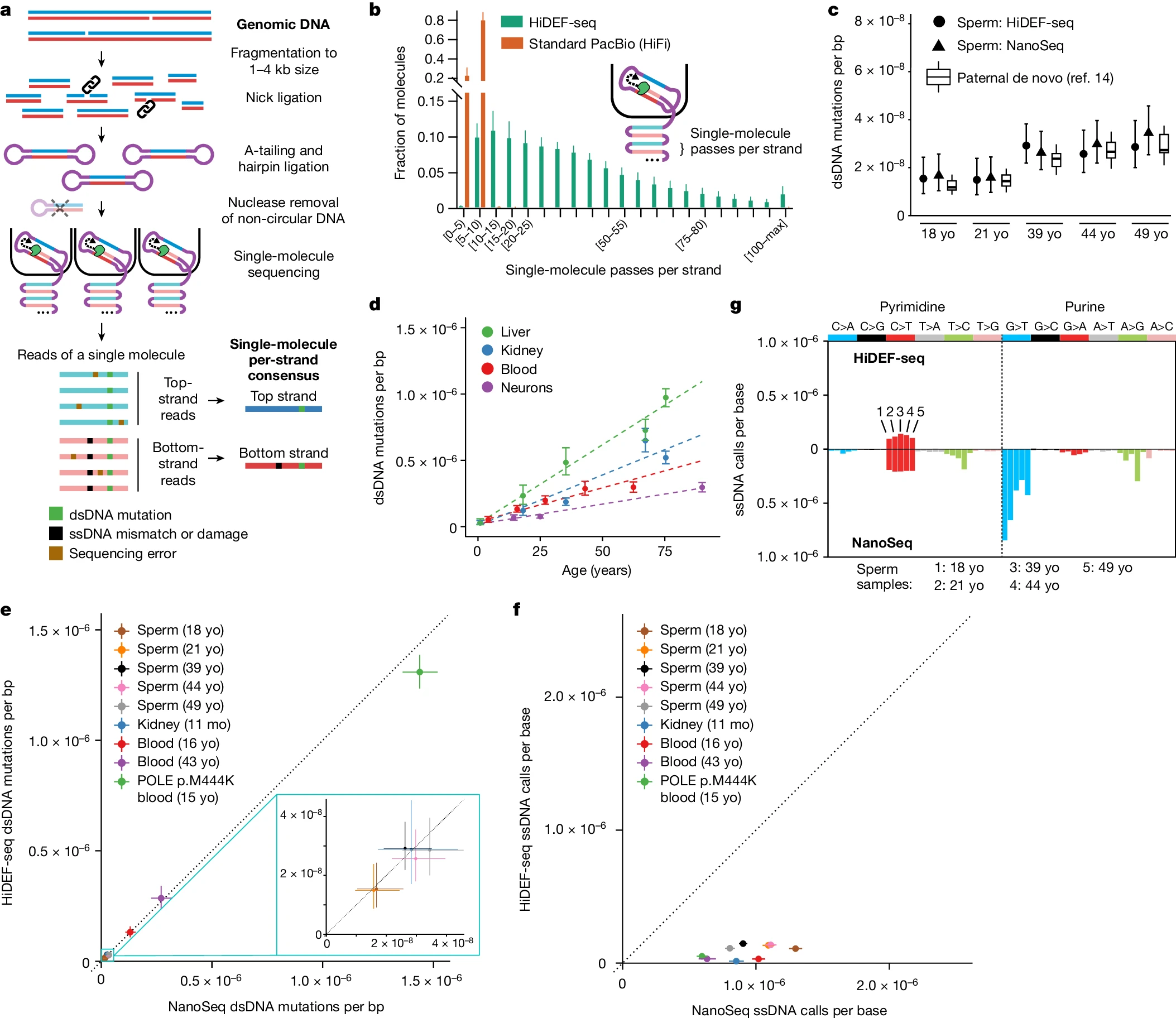
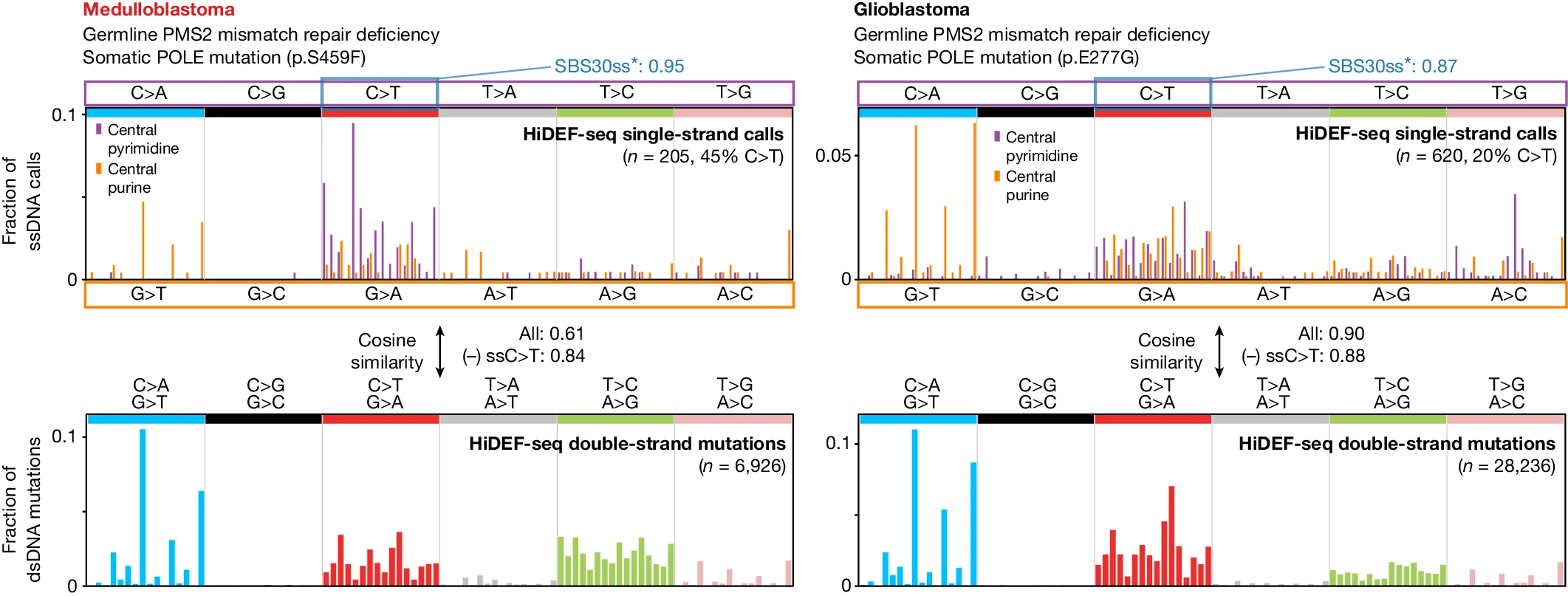
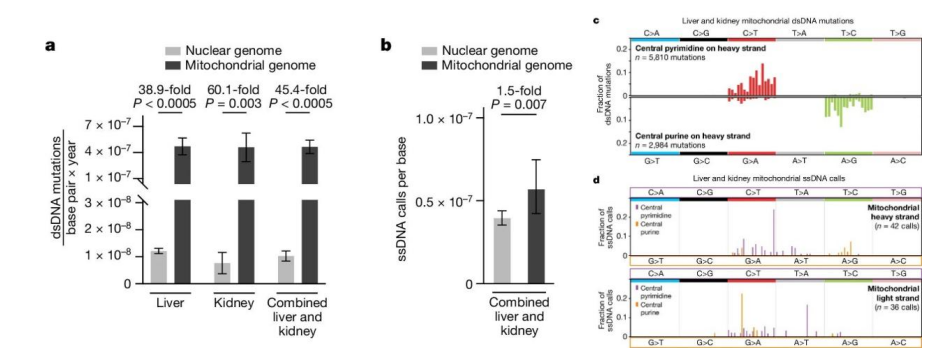
在人类基因组中,突变的积累是生命过程中不可避免的现象。这些突变往往起始于DNA双螺旋结构中的一条链上的核苷酸不匹配或损伤,如果这些单链DNA(ssDNA)事件未被及时修复或修复不当,它们将转化为双链DNA(dsDNA)突变,进而可能导致癌症和其他遗传性疾病。尽管现有的DNA测序技术在检测双链DNA突变方面取得了显著进展,但它们不能准确地解决这些前体ssDNA事件。这是因为目前无论是单细胞基因组测序、体外单细胞克隆、克隆群体的显微解剖或活检,以及双链测序的方法,通常需要在测序前对原始DNA分子进行扩增。这可能会掩盖真正的ssDNA事件或引入人为的ssDNA不匹配和损伤。 近日,来自美国纽约大学格罗斯曼医学院的Gilad D. Evrony带领团队开发了一项名为Hairpin Duplex Enhanced Fidelity sequencing (HiDEF-seq) 的单分子测序新技术,这一技术不仅能够以单分子的精度检测单个碱基的替换,还能识别DNA损伤,包括常见的胞嘧啶脱氨作用。这项技术的应用,为理解突变如何在各种情境下产生,尤其是在癌症和衰老的背景下,提供了新的视角和强有力的工具。这项工作为我们揭示了单分子测序技术在解析单链DNA事件方面的卓越能力。相关成果于2024年6月发表在Nature杂志发表。 技术原理 HiDEF-seq为了实现对单链事件的高精度检测,其具体流程大致如图1所示:首先对基因组DNA进行处理,利用Hpy166II限制性酶对基因组DNA进行消化。将DNA片段化至1-4 kb大小。然后分别进行nick ligation(缺口连接),A-tailing(A尾修饰)和hairpin ligation(发夹结构连接),连接上发卡结构的PacBio测序接头。使用Pacific Biosciences (PacBio)的单分子长读长测序仪进行测序。通过增加独立测序遍数来提高每条链的共识序列质量。由于PacBio的测序酶读长平均在100 kb左右,也就是说一个4 kb的片段平均可以被测25遍,正反两条链中的每一条都可以被测到至少12次,然后将每一条链被反复测序拿到的序列合并在一起分别合成一个单链的共识序列,这个过程中每个位置的序列都会被反复独立测数十遍,合成共识序列的时候就可以直接将几乎所有的测序错误都排除掉,这样就可以拿到一个高准确度的正反链的单链共识序列,最后再根据正反链互补配对的原则再对正反链的共识序列进行矫正得到一个双链突变的信息,就可以同时拿到单链和双链的突变信息和突变率。正因为Duplex测序和PacBio的HiFi测序的巧妙组合,使得HiDEF-seq成为目前最准确的单分子突变率检测技术,单链的突变率降低到千万分之一的水平,而双链突变率更是低于万亿分之一。 作者对HiDEF-seq与NanoSeq这两种测序方法的比较结果,主要目的是为了评估这两种方法在检测不同类型的DNA变异时的表现差异,评估不同测序技术的优势和局限性,特别是在检测低频变异或特定组织/细胞类型中的变异时。比较主要集中在两个方面:双链DNA(dsDNA)突变检测和单链DNA(ssDNA)事件(如错配和损伤)的检测。在双链DNA(dsDNA)突变检测方面,HiDEF-seq能够达到非常高的保真度,低于每10亿个碱基1个错误(10-9),且能够测量到预期的双链DNA突变特征以及随着年龄增长线性增加的突变负担。在检测双链DNA突变在这一指标上,NanoSeq与HiDEF-seq结果一致,表明两种技术在此方面的性能相当。在单链DNA(ssDNA)检测方面,HiDEF-seq在检测单链DNA事件时,平均负载比NanoSeq低18倍,考虑C>T调换时则低5倍。而在NanoSeq虽然在双链DNA突变检测方面表现出色,但在单链DNA事件检测方面可能存在较高的人工产物。这些结果表明,HiDEF-seq和NanoSeq在双链DNA突变检测方面的表现相似,均能准确地检测并量化双链DNA突变。但是,在单链DNA突变检测方面,HiDEF-seq相比于NanoSeq展现出更高的准确性,这使得HiDEF-seq成为目前检测单链DNA事件最准确的技术之一。 图1. HiDEF-seq概述 癌症易感性综合征样本分析 研究团队通过HiDEF-seq技术对来自不同癌症易感性综合征患者的样本进行了分析。结果显示,与非癌症易感性样本相比,某些综合征样本的单链DNA损伤程度显著更高。特别是POLE聚合酶校对相关的息肉综合征(PPAP)和先天性错配修复缺陷综合征(CMMRD)样本,单链DNA损伤的程度显著增加。此外,研究还发现了与POLE相关的单链DNA错配特征SBS10ss,并确认其与已知的双链DNA突变特征SBS10c高度相似,这证实了单链DNA错配可能是双链DNA突变的起始事件。缺乏错配修复和聚合酶校对的高突变肿瘤样本展现了独特的ssDNA损伤模式,这些模式与肿瘤中观察到的双链DNA突变谱具有较高的相似性。 图2. 癌症易感性综合征的ssDNA呼叫负担和模式 高突变肿瘤的分析 研究表明,在缺乏错配修复和聚合酶校正的高突变肿瘤中,单链DNA的错配模式明显不同于仅缺乏聚合酶校正的样本。这些肿瘤样本尤其表现出较高的单链DNA C>T错配,这些错配主要来源于胞嘧啶的脱氨损伤,而不是聚合酶的错误插入。此外,从这些肿瘤样本中提取的单链DNA错配特征SBS14ss,与COSMIC数据库中记录的双链DNA特征SBS14高度相似,这表明这些单链突变可能是引发双链DNA突变的初始步骤。 图3. 在错配修复和聚合酶校对方面都存在缺陷的高突变肿瘤 胞嘧啶脱氨损伤模式 HiDEF-seq技术能够以单分子精度检测胞嘧啶脱氨损伤,这是一种常见的DNA损伤形式,可导致双链DNA中的C>T突变。研究中发现,健康个体的血液DNA样本中存在低水平的单链DNA C>T突变,这些突变可能是由于胞嘧啶的自发脱氨或实验室处理过程中的热诱导造成的。此外,精子样本中观察到了更高的胞嘧啶脱氨损伤水平,这可能与精子生成或处理过程中的特定条件有关。通过对动力学数据的分析,研究进一步确认了这些损伤源自胞嘧啶脱氨。 图4. 精子和热处理DNA的ssDNA损伤特征 线粒体基因组的突变机制 HiDEF-seq技术为揭示线粒体基因组突变机制提供了新的见解。研究表明,线粒体基因组的突变率显著高于核基因组,这些突变主要发生在DNA复制过程中,可能与线粒体DNA复制时部分单链暴露有关。通过对肝脏和肾脏样本的分析,研究发现线粒体DNA的双链突变负荷与年龄存在正相关,并且在线粒体重链上观察到A>G和C>T突变的非对称模式。这些结果进一步支持了线粒体基因组在复制期间发生突变的观点。 图5. 线粒体基因组dsDNA和ssDNA的调用负荷和模式 结 论 HiDEF-seq技术的应用不仅在技术层面上取得了显著进展,还在生物学上为探究DNA突变的起源和机制提供了新的工具和视角。该技术能够详细揭示单链DNA事件,实时监测DNA损伤、修复和复制过程的动态,这对于理解突变在癌症和衰老等背景下的产生至关重要。研究发现,不仅为癌症易感性综合征中突变的起源提供了新的线索,还为未来的治疗和干预策略提供了潜在的靶点。此外,HiDEF-seq对线粒体基因组突变机制的新认识,也为相关疾病的研究和治疗开辟了新的方向。随着该技术的不断发展和应用,有望在癌症预防和治疗等领域实现更多突破。这项技术具有广阔的应用前景,将极大推动我们对DNA突变及其相关疾病机制的理解。