
2024-08-12

生物地球化学循环是当前地球科学领域研究的重点内容之一,描述了生物圈与地球其他圈层(如大气圈、水圈、土壤全、岩石圈)之间物质和能量的转移。微生物不仅是生态系统中最基础且最为丰富的生物组成部分,更是生物地球化学循环中无可替代的驱动力。作为地球化学元素转化的首要执行者,微生物通过其独特的代谢途径,深刻影响着碳、氮、磷、硫等关键元素的生物可用性与地球环境的化学平衡。 宏基因组能够揭示微生物参与复杂生物地球化学过程的遗传潜能与功能途径,通过挖掘宏基因数据,可以识别出微生物参与特定元素循环的关键基因、代谢通路和发挥作用的酶,为了更全面、更系统地解析微生物在多种元素循环中的作用(包括碳、氮、磷、硫、铁、砷、汞、硒),派森诺技术团队通过整合KEGG、Metacyc、CAZy、KOFAM、Pfam、TIGRfam、dbCan2、MEROPs、Mcyc、Ncyc、Pcyc、Scyc、FeGenie、Asgenedb等多个数据库中与元素循环相关的功能基因集,推出宏基因组生物地球化学循环分析。
PSN生物地球化学循环功能基因集 PSN生物地球化学循环功能基因集包含9大子库,总计收集107条功能通路,标记基因累计高达1225个。
PSN生物地球化学循环项目分析优势 ✔多库融合解析元素循环! ✔突破碳氮磷硫,全景元素动态尽在掌握! ✔分析图表大集合一站式交付! PSN生物地球化学循环功能基因集详细信息
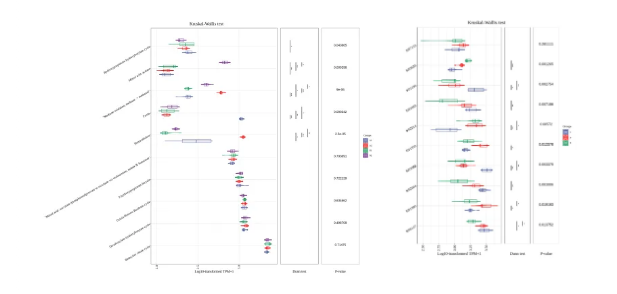
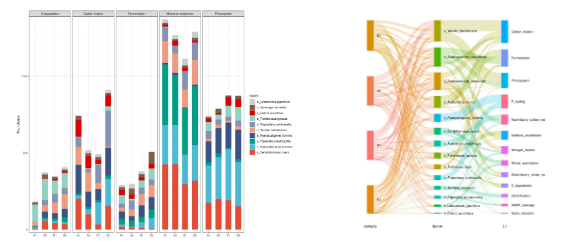
部分分析结果展示 ✔多库融合建设全面功能基因集,丰富图表搭建实用分析报告。 ✔PSN生物地球化学循环项目报告提供功能通路循环图、功能通路/功能基因丰度统计、差异比较、功能通路物种贡献度分析等多维结果。 ✔Nature、Science等国际顶级期刊的专业配色方案,实用又美观! 图1代谢通路循环图、功能通路组成堆积柱形图 图2 功能通路与功能基因组成热图 图3 功能通路LEfSe分析分类学分支图与LDA柱形图 图4 功能通路与功能基因差异分析 图5 物种对代谢通路的贡献度堆积柱形图、桑基图 生态领域多组学文章集锦 生态领域多组学|微观与宏观交织的生命探索之旅 生态领域研究热点解析|多组学视角下的环境微塑料污染研究 生态领域多组学 | 抗生素篇 生态领域多组学 | 盐胁迫篇 生态领域研究热点解析|多组学视角下的颗粒物污染研究 生态领域研究热点解析 | 多组学视角下的重金属污染 多组学研究合集 | 生态+水产+农业资源普查 生态领域研究热点解析 | 多组学视角下的农药污染 采样指南|春季采样忙,小派来帮忙!!! Nature Microbiology:叶际微生物组诱导宿主代谢防御水稻稻曲病的机制 Nature Communications:多组学联合分析揭示植物病原菌拮抗活性物吡唑三嗪的生物合成与调控机制 Microbiome:固氮内生菌可提高中国芒草生态适应性 Journal of Hazardous Materials:蜈蚣凤尾蕨内生菌介导了砷污染的生态修复机制 Environmental Science & Technology:海洋脱卤剂及其伴侣:2,4,6-TCP脱氯过程中的微生物作用和响应 Horticulture Research:高通量测序和土壤代谢组揭示多巴胺通过招募有益微生物来缓解苹果镉胁迫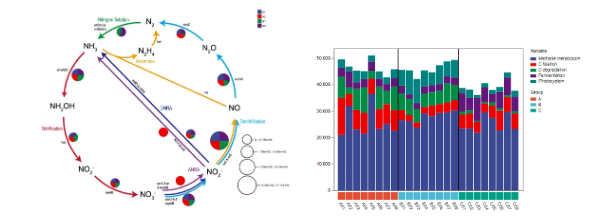


参考文献 Kanehisa M, Goto S. KEGG: kyoto encyclopedia of genes and genomes. Nucleic Acids Res. 2000 Jan 1;28(1):27-30. doi: 10.1093/nar/28.1.27. PMID: 10592173; PMCID: PMC102409. Garber AI, Nealson KH, Okamoto A, McAllister SM, Chan CS, Barco RA, Merino N. FeGenie: A Comprehensive Tool for the Identification of Iron Genes and Iron Gene Neighborhoods in Genome and Metagenome Assemblies. Front Microbiol. 2020 Jan 31;11:37. doi: 10.3389/fmicb.2020.00037. PMID: 32082281; PMCID: PMC7005843. Song W , Xiong H , Qi R ,et al.Effect of salinity and algae biomass on mercury cycling genes and bacterial communities in sediments under mercury contamination: Implications of the mercury cycle in arid regions[J].Environmental Pollution, 2021, 269(12):116141.DOI:10.1016/j.envpol.2020.116141. Parks JM, Johs A, Podar M, Bridou R, Hurt RA Jr, Smith SD, Tomanicek SJ, Qian Y, Brown SD, Brandt CC, Palumbo AV, Smith JC, Wall JD, Elias DA, Liang L. The genetic basis for bacterial mercury methylation. Science. 2013 Mar 15;339(6125):1332-5. doi: 10.1126/science.1230667. Epub 2013 Feb 7. PMID: 23393089. Song X, Li Y, Stirling E, Zhao K, Wang B, Zhu Y, Luo Y, Xu J, Ma B. AsgeneDB: a curated orthology arsenic metabolism gene database and computational tool for metagenome annotation. [J].NAR genomics and bioinformatics, 2022(4).DOI:10.1093/nargab/lqac094. Xue C X , Lin H , Zhu X Y ,et al.DiTing: A Pipeline to Infer and Compare Biogeochemical Pathways From Metagenomic and Metatranscriptomic Data[J].Frontiers in microbiology, 2021, 12:698286.DOI:10.3389/fmicb.2021.698286. Qian L , Yu X , Zhou J ,et al.MCycDB: A curated database for comprehensively profiling methane cycling processes of environmental microbiomes[J].Molecular ecology resources, 2022(5):22.DOI:10.1111/1755-0998.13589. Qichao,Lin,Lei,et al.NCycDB: a curated integrative database for fast and accurate metagenomic profiling of nitrogen cycling genes.[J].Bioinformatics (Oxford, England), 2018.DOI:10.1093/bioinformatics/bty741. Zeng J , Tu Q , Yu X ,et al.PCycDB: a comprehensive and accurate database for fast analysis of phosphorus cycling genes[J].Microbiome, 2022, 10(1):101.DOI:10.1186/s40168-022-01292-1. Yu, XiaoliZhou, JiayinSong, WenXu, MengzhaoHe, QiangPeng, YishengTian, YunWang, ChengShu, LongfeiWang, ShanquanYan, QingyunLiu, JihuaTu, QichaoHe, Zhili.SCycDB: A curated functional gene database for metagenomic profiling of sulphur cycling pathways[J].Molecular ecology resources, 2021, 21(3).