
2024-08-17

近年来,空间转录组(ST)方法的开发和应用急剧增加。与标准的单细胞方法不同,ST保留了捕获的转录组的空间背景,从而允许直接观察细胞和它们在组织空间中的相互作用的排列。而目前市场上可用的空间转录组技术,其中商业化的空间技术主要受限于相对较高的成本、有限的分辨率以及高特异性捕获等,非商业的空间技术又受限于较低的捕获效率以及繁琐的实验操作等,且均还难以实现端对端在三维空间中生成并计算分析ST。 2024年6月德国柏林医学系统生物学研究所在《Cell》上发表了一篇关于新的空间组学技术的文章(IF:45.5):Open-ST:High-resolution spatial transcriptomics in 3D。主要介绍:作为一种成本效益高的方法,Open-ST结合了高分辨率和高效的转录组捕获,以及广泛的开源实验资源和软件,可以用于在二维和三维中无缝的数据加工和分析。(所有协议和软件可在 https://rajewsky-lab.github.io/openst 上获取)
整体工作流程 通过测序包含barcode(32nt)、测序接头和聚(dT)区域的寡核苷酸来生成空间条形码序列及其相关的(x和y)坐标;处理寡核苷酸以允许聚(A)转录本捕获同时根据实验设计选择对测序芯片进行切割成所需大小(若选择3mm*4mm,则可以切割成360个);也能够从相同的冷冻切片分析组织形态(H&E)和 ST;在处理样本时,整体流程与常规ST技术相仿。蛋白酶和杂交缓冲液结合在一个溶液中,以促进组织的同时渗透和 RNA 捕获,进而减少单链DNA和RNA分子之间的静电排斥;利用qPCR检测确定最佳的渗透条件,以实现最大的mRNA捕获;在文库制备过程中依据前期的qPCR结果,以确定最佳PCR循环次数;原始H&E图像自动预处理,并使用微调的Cellpose模型将图像分割成单个细胞;在核分割后,通过核边界的径向扩展增加了细胞质上下文;使用第一次测序运行中的条形码坐标以及处理后的转录组数据,使用Spacemake在组织空间中映射。Open-ST能够无偏差地适应具有不同细胞大小和密度的组织,并自动从下游分析中排除没有细胞的区域。同时可以在单细胞水平上进行数据分析,并与成像数据集成。最后,鉴于高效的转录捕获和集成的计算流程,Open-ST适用于任何组织的三维空间重建(图1-2)。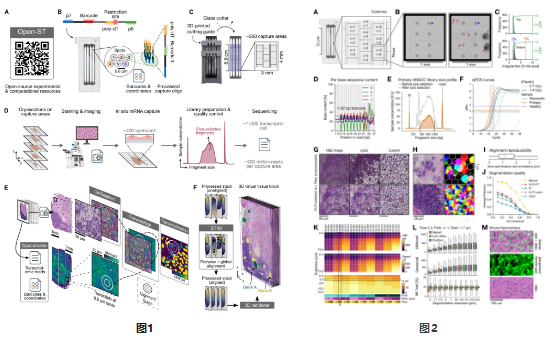
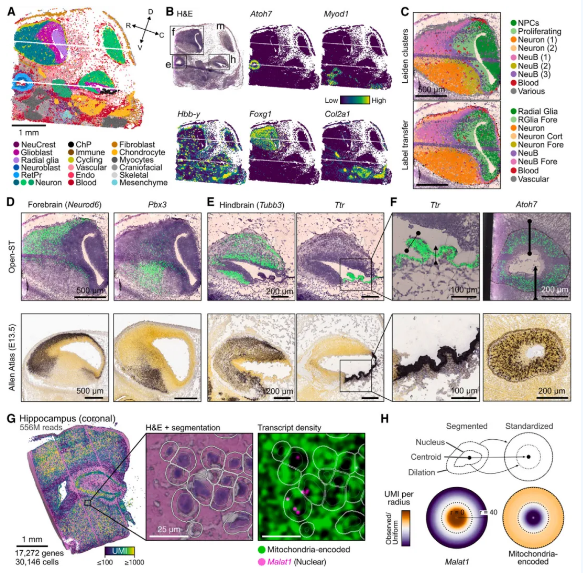
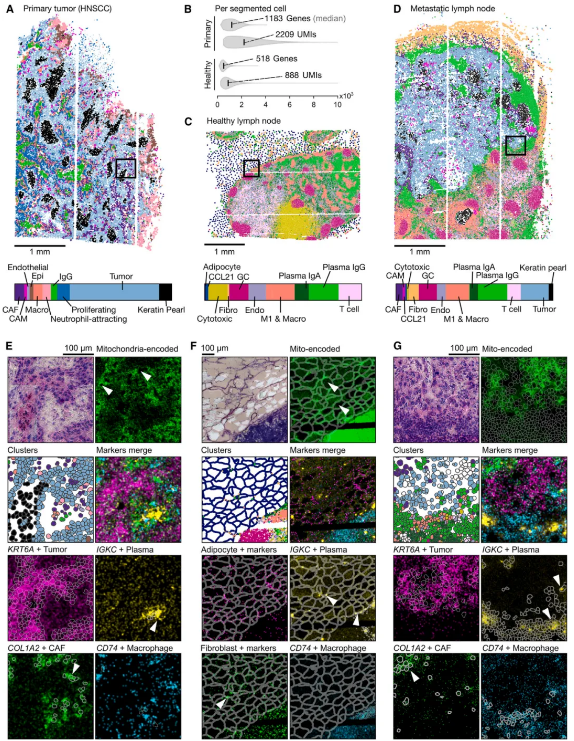
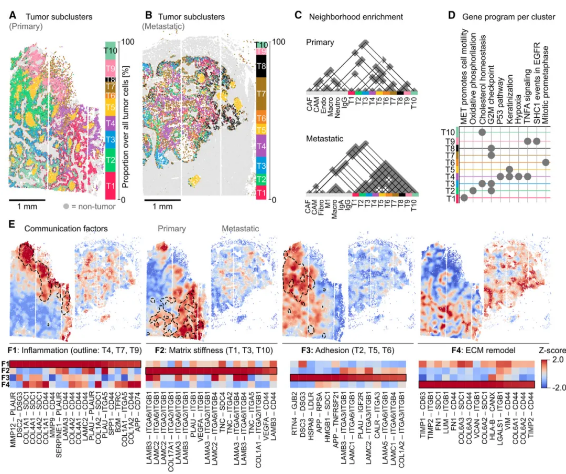
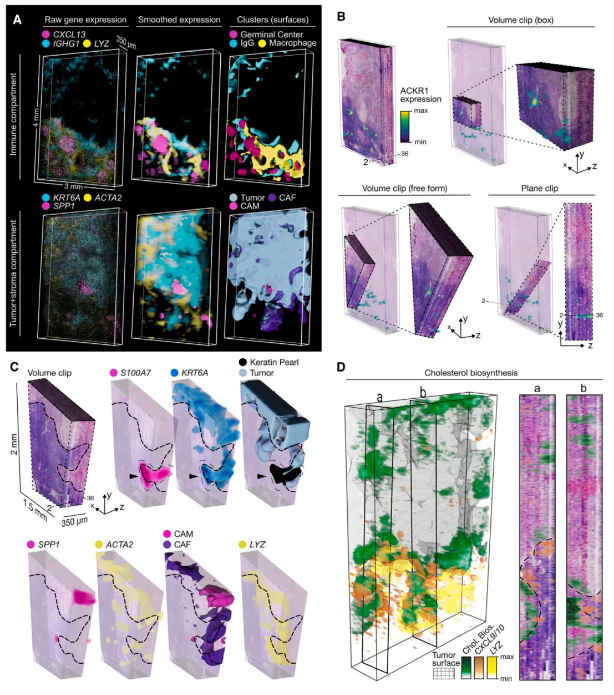
主要优势及其应用 高效且稳定地捕获转录本 Open-ST在多种组织(胚胎小鼠头部、成年小鼠海马体、人类原发性肿瘤)应用中,均展现了高比例的独特基因组映射转录本(65%-78%),并且只有一小部分读段由核糖体RNA 组成(2.5%-15.3%)。在每个细胞中平均捕获到621个基因和880个UMIs,其中42%的高质量细胞(所有分割细胞的 32%)含有超过 1,000个转录本。所有捕获的转录本中,82% 被发现在分割的细胞内。在整个数据集中,10,000个基因占到了捕获的转录组信息的95%。为评估了Open-ST数据的库复杂性,与现有的基于测序的ST方法进行了比较发现在相同测序深度的捕获效率方面始终优于其他空间技术,除了Visium显示出相当的性能(图3)。 图3 高精度捕获局部标记基因 Open-ST能够高精度捕获组织局部的标记基因。为了验证其性能,选取了E13小鼠头部(文献已报道)进行测试。基于Open-ST数据结果大致聚类反映了胚胎小鼠头部的主要解剖区域并共鉴定了三个神经元簇,分别以Tbr1、Lhx9或Nefl 表达为特征。进一步的簇包括成纤维细胞(表达胶原蛋白)、软骨细胞、肌细胞、间充质细胞和内皮细胞。同时初步聚类揭示了一组没有可识别标记的细胞。在这些细胞上,我们进行了子聚类,并鉴定了我们称之为神经嵴、颅面发育、肌肉骨骼发育、免疫和早期血管的细胞群。Open-ST捕获丰富的转录组信息的能力,进而能够对前脑、中脑和后脑的分割细胞进行更精细的聚类。前脑中细胞类型的多样性和空间分布与从单一细胞参考图谱中整合和转移细胞类型标签所获得的结果相似。此外,为了调查捕获定位,我们将转录本在H&E上绘制成虚拟原位杂交,强度与基因表达相关,结果与Allen发展中鼠脑图谱中的染色体原位杂交(ISH)染色高度一致。同时通过局部捕获也反映明显的核质结构,在研究成年小鼠海马体半球的冠状切片发现,通过细胞分割掩模上可视化核保留的Malat1和线粒体编码转录本计数,显示出了准确的转录本定位。Malat1在核中显著富集,而线粒体转录本在质室中富集(图4)。 图4 空间复杂细胞类型捕获能力 Open-ST具有较好的空间复杂细胞类型的捕获能力。通过对一个原发性头颈鳞状细胞癌肿瘤以及同一患者的健康和转移性淋巴结进行了空间测序。在所有三个数据集中,我们都鉴定出了对应于肿瘤、基质和免疫群体转录多样性的标记,其空间邻近富集与病理学家在 H&E图像中独立概述的注释相匹配。例如,“角蛋白珍珠”(KPs),作为鳞状细胞癌的特征,出现在原发性和转移性肿瘤组织中;肿瘤细胞和癌症相关的巨噬细胞(CAMs)在两种原发性和转移性组织中都表达了FABP5;癌症相关的成纤维细胞(CAFs)(COL1A1+)在两种肿瘤组织的微环境中被鉴定出来,其中一部分在转移性肿瘤中表达了高水平的ACTA2;内皮细胞(PECAM1、ACTA2)在原发性肿瘤中被鉴定出来,而淋巴结中的内皮细胞表达了 ACKR1;在转移性淋巴结内,CXCL8在CAM群体中表达,与肿瘤和KP簇共定位等。最后还检测到多个生发中心。这些生发中心表现出局部表达FDSCP,一种由生发中心树突细胞分泌的蛋白质以及IGHM。此外,根据IGHG3或IGHA1的强表达,将两个簇分类为免疫球蛋白IgG或 IgA浆细胞。无偏细胞类型标记特征鉴定了免疫和基质细胞在脂肪细胞群体内(FABP4、ADIPOQ、PLIN1、SORBS1、CIDEC 和 SIK2)(图5)。 图5 揭示原发性和转移性头颈鳞状细胞癌中的空间受限异质性 Open-ST揭示了在原发性肿瘤中紧密相邻的多种肿瘤细胞状态,它们的空间组织紧密遵循细胞-细胞通讯热点的分布。通过整合和聚类两个样本(发性头颈鳞状细胞癌和转移性淋巴结中肿瘤)中的42,132个肿瘤细胞,我们鉴定出了10种不同的转录组状态(T1-T10),为它们的异质性、空间分布和基质相互作用提供了详细的图谱。肿瘤转录组状态在原发性和转移性样本中显示出不同的空间组织模式。特别是在原发性肿瘤中,肿瘤状态被组织成连续的空间域,每个域普遍对应于特定的状态。相反,在转移性组织中,肿瘤状态看起来更加混合,没有明确定义的边界。基于它们的表达水平,通过非负矩阵分解实现,得到了四个非负因子,跨越了原发性肿瘤和转移,每个因子具有独特的相互作用特征:炎症(F1)、基质硬度(F2)、粘附(F3)和细胞外基质(ECM)重塑(F4)。这些因子在原发性肿瘤中显示出比在转移性中更高的空间组织程度,遵循更加混合的肿瘤状态组织。不同的肿瘤状态被限制在特定的细胞-细胞相互作用热点(图6)。 图6 VS Xenium In Situ Open-ST产生的空间基因表达和转录组簇与Xenium结果基本一致。使用相同的人类肿瘤样本,将Open-ST与基于成像的ST方法(Xenium In Situ,10X Genomics)进行了比较发现,两种方法都在两个样本中解析出相似的多样性和空间分布的细胞类型,尽管它们在切片轴上的间隔超过100um。在两种模式下独立注释为相同身份的簇显示出基因表达大体一致。采用相似的分割方法,两种方法每平方毫米产生了大约5200-5500个细胞。在Open-ST中,被细胞捕获的总转录本比例比Xenium低约20%。尽管存在这些差异,两种方法在整个样本中所有基因的总表达上高度相关。此外,无论是否进行子集化,Open-ST都能产生相似的细胞类型的分类和空间分布(图7)。 图7 三维虚拟组织构建以及探索三维其他生物学信息 Open-ST能够在连续切片中稳健地捕获转录本,非常适合高通量研究和组织的三维转录组重建。通过处理一个人转移性淋巴结,获得了跨越350 um组织深度的10 um切片,其中19个切片用Open-ST进行分析、11个切片仅进行 H&E 染色、1个切片进行免疫荧光染色以及5个切片保留用于验证。实验结果,共获得了超过100万个细胞的基因表达谱,每个分割的细胞中基因(313-624)和UMIs(438-1008)的捕获中位数都很高,利用STIM和 Visualization Toolkit,将连续切片对齐,重建并可视化三维虚拟组织块并通过平滑等值面渲染空间映射的UMI计数表明了在肿瘤区域的可重复捕获效率(图8)。 图8 Open-ST能够通过体积渲染和平滑表示来增强清晰度,进而在三维中探索基因表达。例如,我们通过特定标记基因的表达在三维中可视化了淋巴结的淋巴样、转移性肿瘤和基质组织区域的划分。重建的组织可以用于全面的探索性三维分析,同时查询转录组和成像模态。像体积裁剪和平面裁剪这样的渲染技术可以用来探索任何方向上的基因表达,而不受切片平面的限制。例如,我们可视化了ACKR1(一种局限于后毛细血管静脉的内皮标记)的基因表达连续性,通过在三维虚拟组织块中使用平面裁剪,可以跨 z 轴跟踪后毛细血管静脉,并揭示其表达是如何局限于转移性淋巴结的免疫区域,形成网络状结构。此外,我们检查了在初始切片中看似与主要肿瘤块断开的角化珠(KP),我们的三维重建揭示了它在后续切片中与肿瘤块的连续性,证明了在成像和转录组模态中的连续性。此外,三维重建的力量远远超出了对细胞类型和组织结构的研究;例如,检查三维中细胞具有高胆固醇生物合成基因集活性的空间组织(图9)。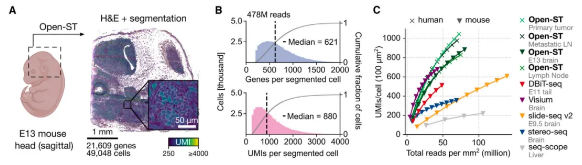


结 论 Open-ST,是一套实验和计算工具的框架,用于在亚细胞分辨率下高效捕获和分析组织空间中的转录本, 在空间测序结构复杂的临床相关人类原发组织方面的具有广泛适用性。在数据处理方面,使用Open-ST计算工具包可以应用于其他实验设置和处理过的样本。此外,模块化方法允许在必要时使用替代工具和算法进行任何(预)处理步骤。总的来说,在提供了详细的实验资源和开源计算工具情况下,Open-ST有望成为后期科研用户的强有力的工具以及ST分析的标杆。