
2024-08-27

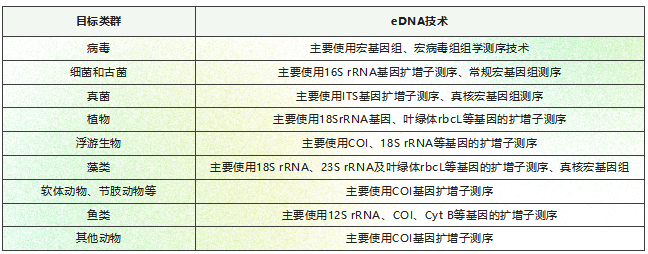
环境DNA(eDNA)技术是指利用如土壤、水体、粪便等环境样本中直接提取的DNA,这些DNA可能来自生物残体细胞、分泌物、排泄物、血液等。通过如下两种方式进行测序: (1)直接对DNA(或cDNA)进行建库测序; (2)扩增特征基因,对扩增产物建库,进行高通量测序,即为eDNA宏条形码技术。 第一种方式就是我们常说的宏组学。主要适合于病毒、古菌、细菌、真菌等在生物。第二种方式是扩增子技术,通过特定的标记基因,来检测携带该基因的类群在样本采集点附近的情况。表1.1汇总了常见类群分析使用的eDNA技术与标记基因。eDNA宏条形码技术近年来受到了广泛关注,并逐渐应用于生物多样性研究、生物监测、食性研究、古环境分析、珍稀濒危物种和外来入侵物种检测等生态学领域。 eDNA宏条形码技术适用的研究方向众多
表1.1 常见目标类群分析
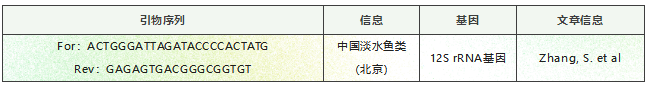
eDNA 宏条形码技术可对水体中多种类群的eDNA 短片段进行扩增,并与高通量测序技术相结合,允许同时对数十至数百个样本进行大规模并行测序分析,是有效的大尺度生物监测手段。使用eDNA技术监测鱼类信息是比较常见的研究类型。Miya等人统计了鱼类群体eDNA相关研究的信息,指出目前对鱼类监测使用较多的基因为12S rRNA基因,特别是MiFish-U/E引物对。关注鱼类群体常使用的基因包括12S rRNA、16S rRNA、18S rRNA、COI、Cytb等基因1。 Miya等人统计的eDNA宏条形码技术监测鱼类群体引物使用率 2022年,Zhang等人在Science Advances上发表eDNA研究2。文章中利用12S rRNA基因扩增子测序(表1.2),对北京市109个定点和定点的鱼类群落进行了调查,探讨了环境变量在城市空间尺度上对鱼类生物多样性的影响。研究鉴定出52个本地和23个非本地分类群,在水域和水域中既有常见物种,也有生境特有物种。结果显示水质对本地鱼类多样性的影响很大,但对非本地鱼类的影响很小。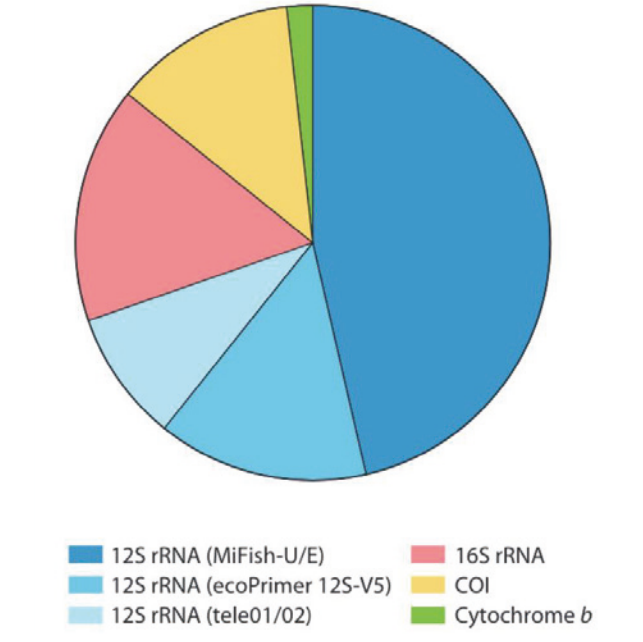
表1.2 上述研究引物列表
除了鱼类群体的监测外,水体样本eDNA技术还可以对浮游生物、底栖生物等类群进行监测分析。下面列举一些eDNA水体滤膜相关的研究工作。2019年,Wang等人在Microbiome上发表eDNA研究3。文章中使用了18S rRNA基因扩增子测序,分析了长江浮游和底栖硅藻的分子生物地理学信息。2020年Djurhuus等人在Nature Communications上发表eDNA研究,分析了海洋群落的季节变化和潜在的互作4。文章中使用了16S RNA、18S rRNA、12S rRNA、COI等基因的扩增子测序,结果数据集包含663个分类群(在科或更高的分类等级),范围从微生物到哺乳动物。文章探究了群落组成的变化,揭示了分类群之间的相互作用,并确定了这些群落与环境属性之间的相关性。利用网络分析解析了捕食者-猎物关系、营养联系和季节变化等。同年Carraro等人也在Nature Communications上发表eDNA研究5。文章中使用了COI基因扩增子测序,分析了包含Habroleptoides、Protonemura、Athripsodes等在内的生物多样性信息,分析了淡水生态系统生物多样性的空间格局。2022年,Zhang等人在Water Research上发表eDNA研究6。文章中利用16S rRNA基因V3区和18S rRNA基因V9区的扩增子测序,从26个位点鉴定出136种浮游植物,其中蓝藻31种,真核浮游植物105种。文章指出,与形态学方法相比,eDNA宏条形码技术在检测纳米/微型浮游生物(< 20 μm)方面具有明显的优势。2023年,Novotny等人在Science Advances上发表eDNA研究7。文章中利用16S rRNA基因扩增子测序,分析了浮游生物。表1.3汇总了上述文章中使用的扩增子基因和引物信息。此外,Burki等人对原生生物eDNA研究进行了综述介绍8,强调了使用eDNA条形码技术可以有效探究这些微型生物的多样性和丰富性。对于原生生物的eDNA研究常使用18S rRNA基因V4和V9区。 表1.3 上述研究引物列表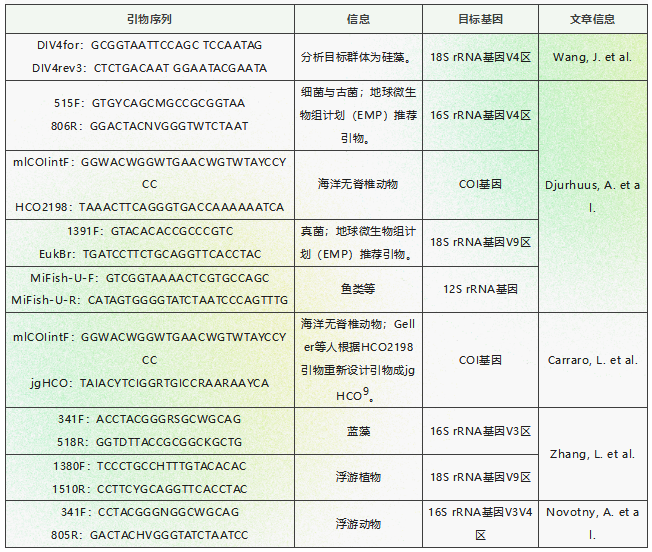
水环境的样本除了水体滤膜外,还可以是沉积底泥或者河岸土壤等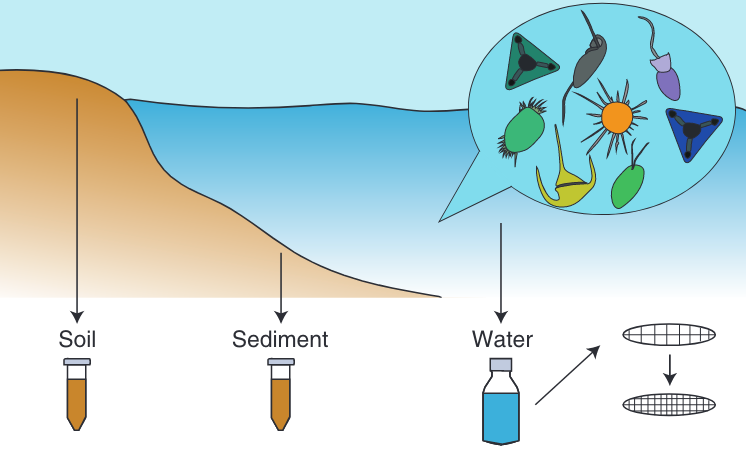
对于水体样本的采集,常用的方法是分级过滤。可根据关注的生物类群选择滤膜孔径大小,下方描述仅作参考,请结合实际关注的分类群和文献描述进行滤膜孔径选择: ” 细菌(bacteria):一般选择0.22 μm 超微型浮游生物(picoplankton):0.2-3 μm 微型浮游生物(nanoplankton):3-20 μm 小型浮游生物(microplankton):20-200 μm 中型浮游生物(Mesoplankton):180-2000 μm ” 注意:如考虑多个分类群,可以采用分级过滤的形式。如Uyaguari-Diaz等人10选择105 μm孔径滤膜进行预过滤,通过105 μm孔径滤的水体使用1 μm孔径滤膜过滤捕获真核生物细胞,通过1 μm孔径滤的水体再用0.2 μm孔径滤膜过滤捕获细菌。
派森诺生物在环境DNA领域项目累积了众多项目经验,并且拥有丰富的标记基因自建库,让分析结果更加精准。如果您对eDNA项目感兴趣,欢迎前来沟通进一步沟通:021-80118168
参考文献: 1.Wang, S. et al. Methodology of fish eDNA and its applications in ecology and environment. Science of The Total Environment 755, 142622 (2021). 2.Zhang, S. et al. Environmental DNA captures native and non-native fish community variations across the lentic and lotic systems of a megacity. Sci. Adv. 8, eabk0097 (2022). 3.Wang, J. et al. Molecular biogeography of planktonic and benthic diatoms in the Yangtze River. Microbiome 7, 153 (2019). 4.Djurhuus, A. et al. Environmental DNA reveals seasonal shifts and potential interactions in a marine community. Nat Commun 11, 254 (2020). 5.Carraro, L., Mächler, E., Wüthrich, R. & Altermatt, F. Environmental DNA allows upscaling spatial patterns of biodiversity in freshwater ecosystems. Nat Commun 11, 3585 (2020). 6.Zhang, L. et al. eDNA biomonitoring revealed the ecological effects of water diversion projects between Yangtze River and Tai Lake. Water Research 210, 7.Novotny, A. et al. DNA metabarcoding highlights cyanobacteria as the main source of primary production in a pelagic food web model. Sci. Adv. 9, eadg1096 (2023). 8.Burki, F., Sandin, M. M. & Jamy, M. Diversity and ecology of protists revealed by metabarcoding. Current Biology 31, R1267–R1280 (2021). 9.Geller, J., Meyer, C., Parker, M. & Hawk, H. Redesign of PCR primers for mitochondrial cytochrome c oxidase subunit I for marine invertebrates and application in all‐taxa biotic surveys. Molecular Ecology Resources 13, 851–861 (2013). 10.Uyaguari-Diaz, M. I. et al. A comprehensive method for amplicon-based and metagenomic characterization of viruses, bacteria, and eukaryotes in freshwater samples. Microbiome 4, 20 (2016).