
2024-08-30

BSA(Bulk Segregant Analysis,混合分组分析法)是基因定位研究中常用的一种方法。随着新一代测序(NGS)技术的日益成熟及其成本的显著下降,BSA与NGS的结合(即BSA-seq)已成为基因定位的首选方法之一。从专门针对EMS诱变的MutMap方法,到适用于不同群体(如F1、F2、RIL等)的多种分析技术(如QTL-seq、ED、G’等),BSA-seq在基因定位中的应用日益广泛和深入。 如果你研究的物种有一个较好的参考基因组和注释,研究的群体是F2代,并且性状符合3:1的分离比,表明是单基因控制,加之两个亲本的亲缘关系不近不远,那么恭喜你,你的基因成功定位的概率超过了90%,小论文发表的日子指日可待。然而,我得提醒你,沉溺于美好的幻想并不明智,赶紧起来统计性状吧!想想看,在常见物种中,这么容易定位的基因,早就被领域内的专家们攻克了。如果真的遇到这样理想的情况,先多查阅文献,确认你的基因是否已经被报道过。 大多数情况下,基因定位的过程并非一帆风顺。例如,在显性突变的情况下,如果在BCF1代观察到性状分离比为1:1,并且使用极端性状进行混池分析,理论上仅通过SNP-index分析方法就可以成功定位到候选区间。然而,现实中常常会遇到不尽如人意的结果:要么无法定位到区间,要么定位到的区间中没有目标基因。 那么,遇到这种情况该怎么办呢? 在这种情况下,我们推荐使用OcBSA方法来进行分析。这一方法由中国农业科学院蔬菜花卉研究所蔬菜分子设计育种创新团队的程锋研究员与马铃薯遗传育种与栽培创新团队的李广存研究员共同提出,相关研究成果以“OcBSA: an NGS-based Bulk Segregant Analysis Tool for Outcross Populations”为题[1],于2024年2月17日发表在《Molecular Plant》期刊上。 接下来,我们将详细解读这篇文章的内容,并在最后说明为什么OcBSA方法特别适用于前面提到的那个问题。
背景 由于基因组的高杂合性、自交不亲和性以及长世代等特性,对于这些植物来说,获得能够稳定遗传的自交系是十分困难的。因此,这些物种通常使用异型杂交的繁殖方式,以F1代分离群体作为主要研究对象,例如马铃薯、红薯、黑麦等作物,以及苹果、桃、葡萄、柑橘、茶树等果树。因为双亲的高杂合性,对于二倍体亲本,F1代中会含有四个单倍型。长期以来,基因组的高杂合性和异型杂交一直是分子育种以及物种改良的主要障碍。我们常用的BSA-seq分析方法(如∆SNP-index, ED等),对于这种情况就显得捉襟见肘。 在本研究中,作者开发了一套名为OcBSA的分析工具,主要用于解决在双亲高度杂合的F1群体中进行QTL定位的难题。OcBSA工具通过关注并分析携带致病突变的杂合亲本的两个单倍型组的遗传模式来实现基因定位。其核心假设是,这两个单倍型在两个样本池中的突变位点会表现出遗传偏倚,从而提供定位线索。 相比于其他QTL定位工具,OcBSA具有明显的优势。这一点已经通过计算机模拟的群体和多个先前报道的真实杂交群体(包括苹果、梨、茶树、葡萄、桃子、柑橘和水稻)得到验证。此外,OcBSA还在本研究中构建的F1代群体中成功应用,识别出了调控土豆花色的候选基因。
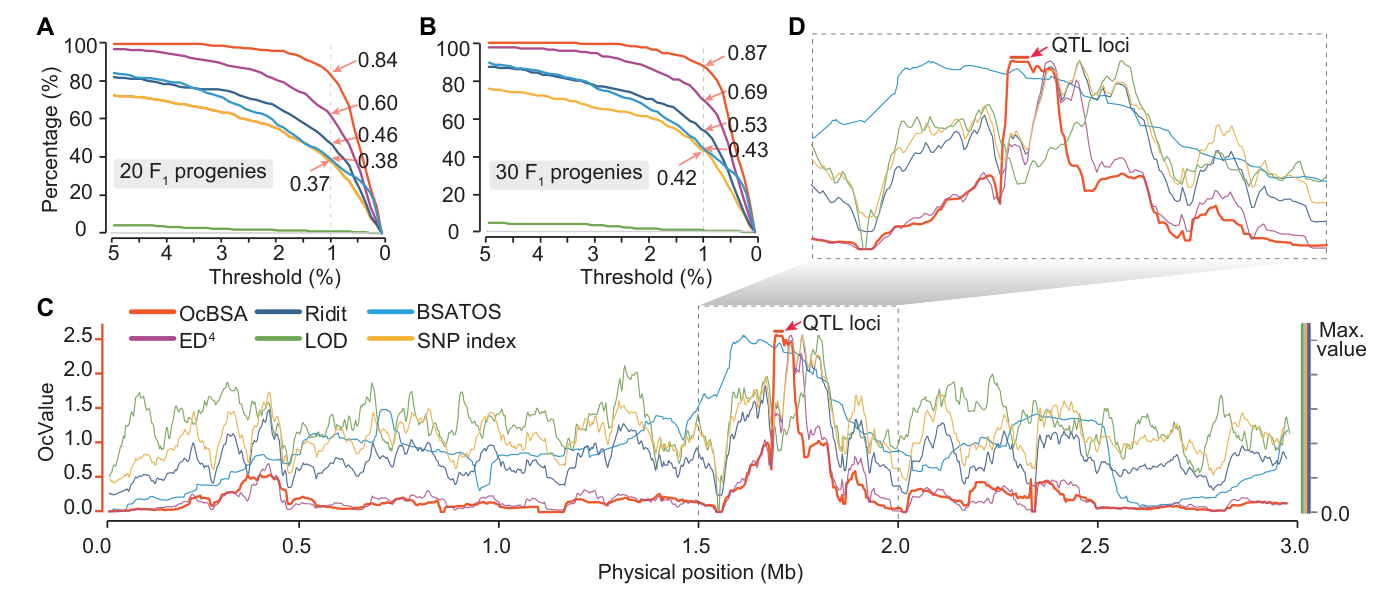
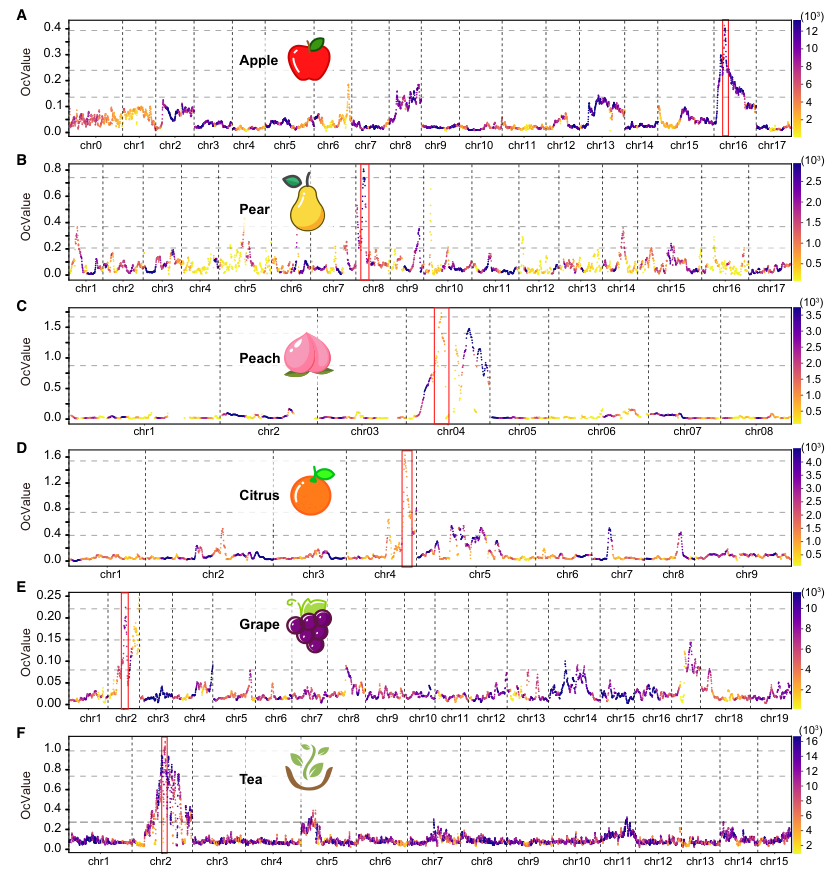
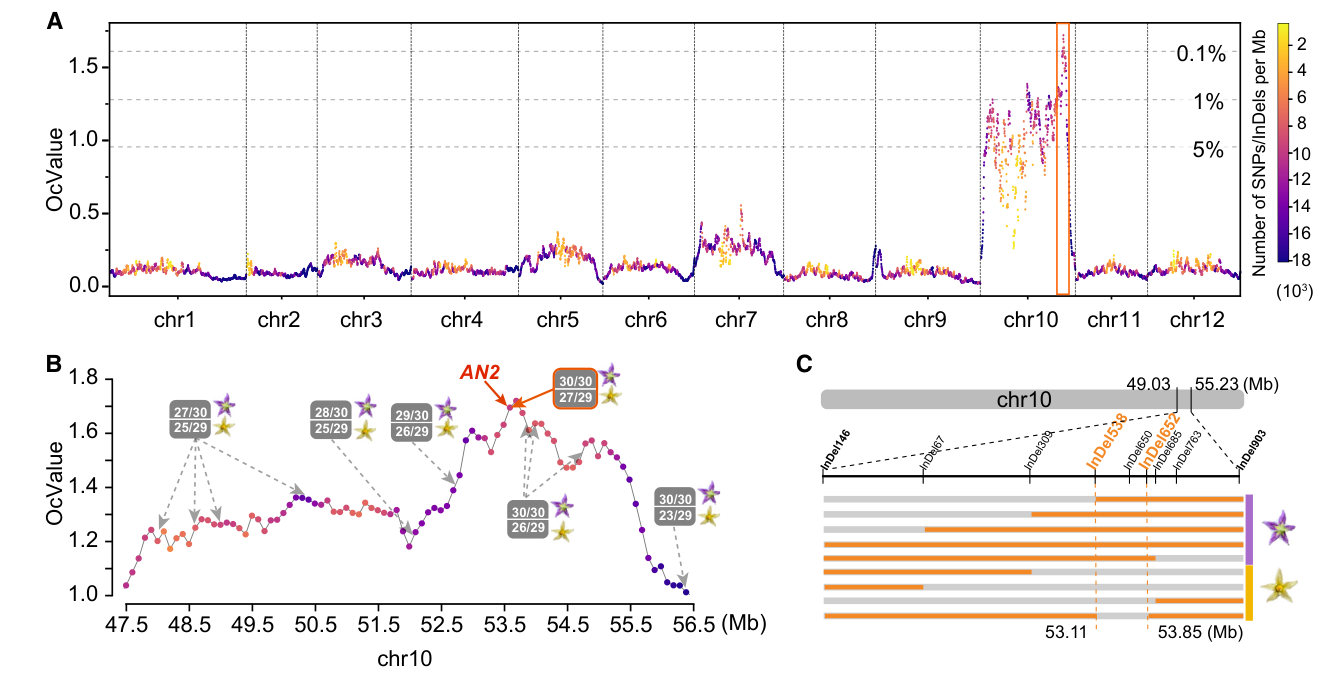
结果 OcBSA的算法与实现 相比于常见的双亲为纯合的F2代群体,由异交得到的F1代群体的双亲通常显示出高度杂合性。在显性遗传模型中,F1代群体的性状分离是由于来自携带显性突变的杂合亲本的两个单倍型在群体中发生了分离,而另一个亲本对性状分离并没有贡献。例如,土豆表皮颜色(如图1A所示)取决于亲本P1的单倍型h1中的突变。在F1代中,携带h1的个体呈现紫色,而携带h2的则呈现黄色,且表皮颜色与亲本P2的单倍型h3和h4无关。在基于目标性状共分离位点的基因型差异的数据分析中(如BSA或连锁作图定位),来自P2的随机单倍型会引入噪音(如图1A中的橙色峰),这会严重影响基因型频率的计算,导致目标信号的偏差或丢失。在这种情况下,当使用F1代群体中两个表皮颜色差异的混池进行BSA-seq分析时,由单倍型h3和h4引入的噪音会大大降低QTL定位信号的敏感性、准确性和显著性。因此,OcBSA的主要策略是从两个混池中去除h3和h4,创建两个新的混池,即OcPool 1和OcPool 2,这两个新池中仅包含h1和h2。这些新生成的虚拟池OcPool 1和OcPool 2,类似于使用亲本P1自交生成的F2代群体所构建的极端性状混池(图1B)。 根据以上规则,作者梳理出了OcBSA的完整工作流程。首先,将两个杂合亲本进行杂交,构建F1代群体,其中一个亲本(P1)携带有目标性状的显性突变。接着,收集具有差异性状的F1代个体,并将其分为两个混池。然后,对这两个混池以及两个亲本的DNA进行全基因组测序。在第二步(如图1C所示),将两个混池、P1和P2的reads比对到参考基因组上,以识别SNP(单核苷酸多态性)和InDel(插入或缺失变异)。对于在两个亲本之间存在多态性的SNP和InDel,做进一步分析。具体来说,保留在P1中为杂合而在P2中为纯合的位点,以便进行单倍型去除分析(如图1C所示)。此外,也保留在P1中为杂合但在P2中缺失的位点,以考虑P2中基因组区域的缺失变异。接下来的步骤是通过去除P2的两个单倍型,构建两个OcPool(如图1C和1D所示)。如图1C所示,两个池中的黄色框中的“m”代表来自P2的单倍型位点。作者通过排除覆盖这些P2基因型的reads来去除这些“m”单倍型。从两个原始混池中过滤掉这些reads后,得到两个OcPool用于进一步分析。在最后一步,作者通过计算遗传分化系数(OcValues),在用户自定义大小的窗口中滑动遍历整个基因组,以估计两个OcPool中单倍型h1和h2(或l和m)之间的遗传差异(如图1所示)。 图1. OcBSA 的原理、方案和工作流程 OcBSA整合多种分析工具(BWA,SAMtools和GATK),允许用户在Windows或Linux平台,使用python脚本将raw data的fastq格式文件或vcf文件作为输入文件进行完整分析。此外,OcBSA集成了批量引物设计工具,为用户之后的精细定位提供便利。 OcBSA 在使用F1代群体进行 QTL 定位方面优于现有工具 为了展示OcBSA在F1代群体中的优势,作者使用QMSim2模拟生成了一个包含1000个F1代个体的群体,全基因组共有100,000个变异位点。利用这个模拟群体,作者选择了一对极端性状的混池,每个混池分别包含20、30和40个子代个体,并进行了600次重复,总共生成了1800个极端性状混池。同时,使用OcBSA以及其他工具/算法(包括Ridit、ED、LOD、BSATOS和SNP-index)进行QTL定位,所有方法使用相同的滑动窗口(窗口大小为30 kb,步长为3 kb)。结果显示,OcBSA的表现远优于其他对比工具,并且随着池中个体数量的增加,准确性也得到了提高(见图2)。特别是,从图2C和2D中可以看出,OcBSA展现出了最优的抗噪音能力。 图2. 使用计算机模拟评估和比较OcBSA与其他工具的准确性 使用来自不同物种的F1代群体数据评估OcBSA 除了模拟数据,作者利用已报道的多种不同物种的F1代群体数据,以检验OcBSA的性能和广泛适用性。作者总共收集了7个来自不同物种的F1代群体数据集,包括苹果、梨、桃子、柑橘、葡萄、茶树和水稻。结果,OcBSA在所有7个数据集中均定位到QTL,并且结果与原报道的结果一致或有提高。 图3. 用OcBSA方法对6个物种的F1群体进行QTL定位的结果 OcBSA有效识别出马铃薯花色调控位点 为了进一步验证OcBSA的有效性,作者构建了一个马铃薯F1代群体,用于定位控制马铃薯花色的QTL。研究中,作者将两个杂合的二倍体马铃薯材料(一个为紫花,另一个为黄花)进行杂交,构建了一个包含252个单株的F1代群体。性状调查结果显示,F1代群体中紫花(132株)与黄花(120株)的比例约为1:1(χ² = 0.48, P = 0.45),表明花色性状由单基因控制。接下来,作者分别选择了30株紫花单株和29株黄花单株,进行混池测序,分别获得了61.84 Gb和64.39 Gb(约70×)的重测序数据。同时,作者对双亲也进行了重测序,分别生成了约15×的重测序数据。通过与参考基因组(DM8.1)进行比对分析,共鉴定出28,141,212个SNP和3,764,490个indel。接下来,作者将利用OcBSA对这些数据进行QTL定位。 结果显示,OcValue超过top 0.1%的峰值位点落在第10号染色体的53.7 Mb区域,区间范围为53.4–53.9 Mb,表明该位置存在QTL。为验证该QTL位点,作者在45.7到56.7 Mb的区间内设计了14个indel标记,并在两个混池样本中进行了基因分型(图4B)。结果显示,indel分型结果与OcBSA定位结果完全一致。进一步分析9个关键重组单株后,作者将定位区间缩小至约740 kb(图4C),该区域包含48个基因。这一区域与前人报道的花青素合成相关的QTL区域重合,并且另一项研究通过转基因验证发现,该区域的ANTHOCYANIN 2基因负责控制马铃薯紫花性状。这一结果再次证明了OcBSA在F1异交群体QTL定位中的准确性和有效性。 图4. 马铃薯F1群体花色的OcBSA QTL定位结果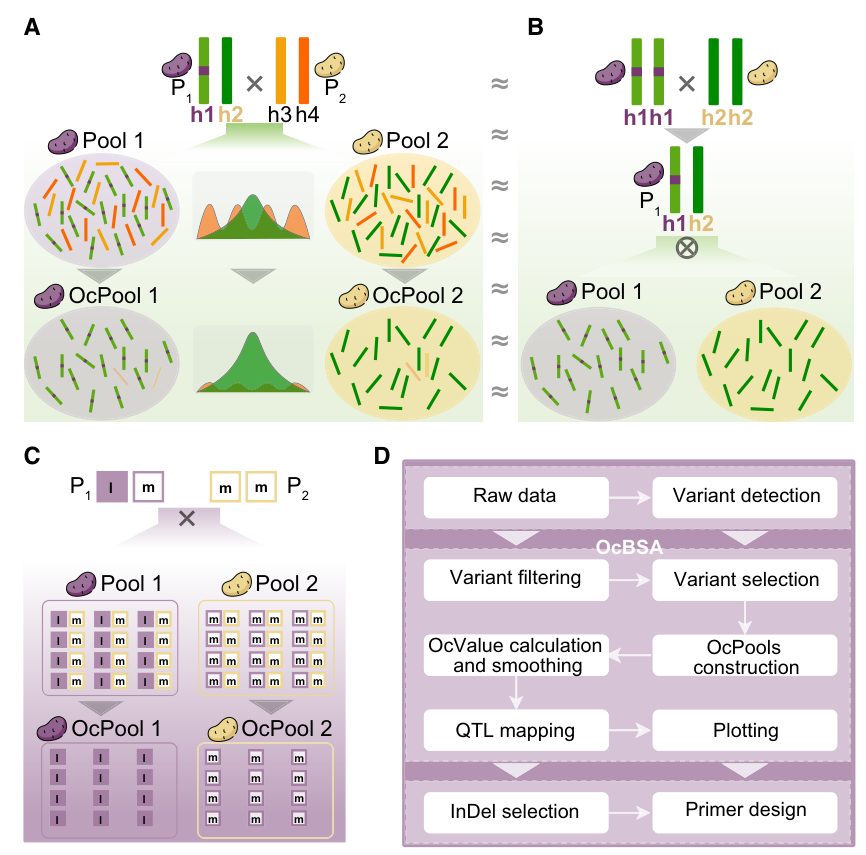
总结 作者通过利用模拟F1代群体数据,将OcBSA与其他BSA-seq分析算法和工具的QTL定位结果进行对比,结果显示,OcBSA的表现显著优于其他分析算法和工具。随后,作者使用OcBSA重新分析了已报道的不同物种的F1代数据集,得到了与原报道一致或更优的结果。最后,作者将OcBSA应用于马铃薯花色QTL定位的研究中,成功定位到前人报道的相关区间及基因。这些结果表明,OcBSA在F1代异交群体的QTL定位研究中具有高度的准确性和有效性,为后续的育种工作提供了重要的支持和帮助。
文章解读完了,大家是否有所启发?让我们回顾一下开篇提到的情况:显性突变,BCF1代,分离比为1:1。BCF1代,即F1代,由于显性突变的存在,携带目标性状的亲本(P1)必定是杂合的,另一亲本(P2)原则上是纯合的。然而,在SNP-index方法未能得到理想结果的情况下,我们有理由怀疑P2的背景也可能不是完全纯合的。如果两个亲本均为杂合,并且为F1代,这就完全符合OcBSA的适用条件。 在实际应用中,对于这种情况,OcBSA往往能带来令人惊喜的结果。所以,大家不妨亲自试一试! 参考文献 1.Zhang, L., et al., OcBSA: An NGS-based bulk segregant analysis tool for outcross populations. Mol Plant, 2024. 17(4): p. 648-657. 如需进一步讨论,欢迎发邮件或者致电我们哟(邮箱地址:genome_support@personalbio.cn,联系电话:021-80118168-6611)!