
2024-09-09

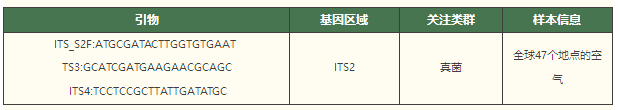
eDNA(environmenta DNA)是指从环境样品(水体、土壤、沉积物、空气、混合物等)中提取物种的DNA,区别于传统从单物种样品中提取的DNA[1,2],是各种生物的DNA混合物[1,2]。而空气样本的收集在生态监测中一直有很大的挑战性,尤其是在检测空气中极微量的DNA时。相比于土壤、水体或沉积物的采集,空气样本难以精确控制其来源和组成,且容易受到外界环境的干扰,如风速、湿度、温度等因素的影响。即便在理想的采样条件下,空气中的DNA通常也极其稀少且易于降解,这给后续的DNA提取和分析带来了困难。 空气滤膜的出现通过使用高效的空气过滤装置,将空气中的颗粒物(包括微生物、花粉、皮屑和其他含有DNA的物质)捕获在滤膜上,为后续的环境DNA分析奠定了基础。空气滤膜eDNA研究的基本原理是通过空气过滤装置捕获空气中的颗粒物,这些颗粒物可能含有来自生物体的DNA片段。eDNA扩增子技术通过从空气滤膜中提取环境DNA,结合高通量测序技术,对这些DNA进行扩增与鉴定分析,从而确定空气中的生物物种组成。eDNA扩增子测序能够对生态群落进行大规模监测和表征,有效的弥补了传统监测方法的不足,为生态研究提供了更加全面和敏捷的手段[3,4]。生物体的监测不再只依赖于物理观察,而是通过其DNA的存在或丰度进行间接监测,这使得可以同时监测成百上千个物种或分类群体,极大提高了监测的广度和效率[3]。这种技术不仅在监测生物多样性方面具有显著优势,还能够识别空气中潜在的病原体,为环境保护与公共卫生提供科学依据。通过定期监测空气样本中的eDNA,可以跟踪物种迁移、季节性变化以及环境压力对生物群落的影响。此外,这种技术在研究气候变化、环境污染和生态修复等领域也展现出巨大的潜力。 真菌是生命中最具多样性和生态重要性的界之一,然而,真菌的分布范围和影响其分布的生态机制在很大程度上仍然是未知的[6,7]。今年(2024)7月份,Abrego等人在Nature主刊上发表了其关于真菌的研究进展[8]。文章从全球47个地点收集了空气中大于1µm的颗粒,揭示了真菌物种丰富度和群落组成在空间和季节上的动态变化过程。 Lynggaard等人在2022年[9]和2024[10]年分别在Current Biology和Mol Ecol Resour上发表了关于动物园和森林中空气eDNA的研究成果,来探索哺乳动物和脊椎动物的多样性。为监测陆地生态系统中的哺乳动物和脊椎动物群落提供了一种高效的工具,这对于理解生物多样性和进行生态系统管理至关重要。这两篇文章用到的是相同的引物,引物详情如下: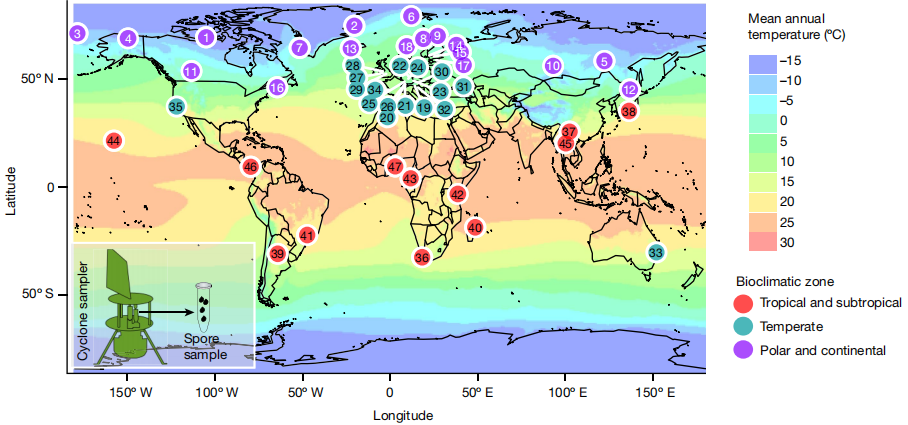
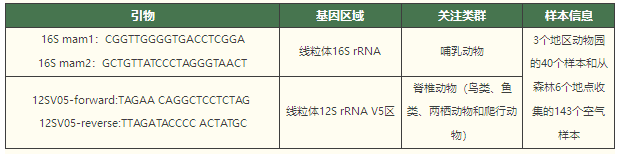
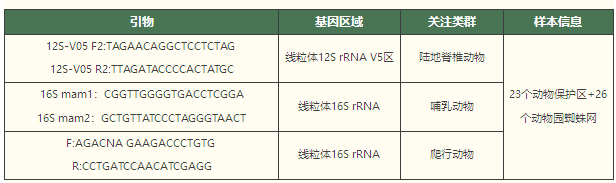
在空气样本的研究中,除了空气滤膜之外,偶尔也会遇到一些有趣的样本。比如Newton等人今年(2024年)在iScience发表了一篇关于蜘蛛网样本的eDNA研究[11],共收集了来自野生动物保护区和动物园的49个蜘蛛网,来探索蜘蛛网能否作为被动生物过滤器来指示环境中的生物多样性。研究结果表明,蜘蛛网能够捕捉到来自周围环境中的多种脊椎动物的eDNA,包括哺乳动物、鸟类、爬行动物和两栖动物。该文用到的测序引物如下:
空气样品采集建议: 1.对于空气样本采集,我们建议使用专门的空气收集仪器,使用时注意对专用采样仪器的进样头、切割头和膜托等关键部位进行清洗。 2.目前主流采样模式大概分为两种,一种是无菌滤膜收集方法,另一种是液体撞击式收集方法。其中无菌滤膜收集方法,可将无菌滤膜置于采样器中(滤膜材质为玻璃纤维,直径80mm),空气流量为100 L/min,采集后用无菌水或PBS缓冲液冲洗滤膜,将滤膜上沉积颗粒物溶于其中,后续对液体12000 r/min离心20min,取沉淀提取DNA即可;另外液体撞击收集方法,建议用无菌水或者PBS缓冲液作为样品吸附液,收集完后,对含有空气微生物的吸附液进行离心或者过滤浓缩的方法提取DNA即可; 3.对于无菌滤膜收集方法,老师可以通过寄送滤膜的形式将收集的空气微生物样品运往公司,运输途中建议干冰寄送; 4.对于液体撞击收集方法,老师可以通过将吸附液,吸附液的体积不能大于500ml;或者将吸附液过滤后的滤膜寄往公司,过滤的步骤可以参照灌溉水样的样本采集流程;样品运输建议干冰寄送;
空气滤膜eDNA研究为生态监测和环境保护提供了一种创新且高效的方法。尽管在样本收集、污染控制和DNA降解等方面仍然面临技术挑战,但我们相信随着分子生物学技术的进步,空气eDNA研究在生态研究和公共卫生领域的发展前景会越来越好!
1.Taberlet P, Coissac E, Hajibabaei M et al. Environmental DNA. Molecular Ecology, 2012, 21(8): 1789-1793 2.What's behind the term? Clarifying the terminology and recom-1/mec.15643.Pawlowski J,Apothéloz-Pemet-Gentil L, Altermatt F. Environmental DNA :mendations for its future use in biomonitoring. Molecular Ecology, 2020, 29(22): 4258-4264. 3.J. B. Mihoub et al., Setting temporal baselines for biodiversity: the limits of available monitoring data for capturing the full impact of anthropogenic pressures. Sci Rep 7, 41591 (2017). 4.K. Bohmann et al., Environmental DNA for wildlife biology and biodiversity monitoring. Trends Ecol Evol 29, 358-367 (2014). 5.L. Estes et al., The spatial and temporal domains of modern ecology. Nat Ecol Evol 2, 819-826 (2018). 6. Peay, K. G., Kennedy, P. G. & Talbot, J. M. Dimensions of biodiversity in the Earth mycobiome. Nat. Rev. Microbiol. 14, 434–447 (2016). 7.Tedersoo, L. et al. The Global Soil Mycobiome consortium dataset for boosting fungal diversity research. Fungal Divers. 111, 573–588 (2021). 8. Abrego, N., Furneaux, B., Hardwick, B. et al. Airborne DNA reveals predictable spatial and seasonal dynamics of fungi. Nature 631, 835–842 (2024). 9. Lynggaard, C., Bertelsen, M. F., & Jensen, C. V., et al. (2022). Airborne environmental DNA for terrestrial vertebrate community monitoring. Current Biology, 32(3), 701-707.e5. 10. Lynggaard, C., Frøslev, T. G., & Johnson, M. S., et al. (2024). Airborne environmental DNA captures terrestrial vertebrate diversity in nature. Molecular Ecology Resources, 24, e13840. 11. Newton, J. P., Nevill, P., & Bateman, P. W., et al. (2024). Spider webs capture environmental DNA from terrestrial vertebrates. iScience, 27(2).