
2024-09-18

当我们做完转录组测序之后,往往需要做qPCR来验证测序结果。这种情况下,基因的选择、样品的情况都是成为影响结果的重要因素。今天就让小派来带大家深挖下这其中的门道吧!
一、验证基因的选择 1、选择自己感兴趣的基因进行验证,即使其表达倍数可能没有到达2倍(比如1.5倍); 2、选择表达量变化幅度较大的基因,比如差异倍数大于两倍(即fc>2或<0.5); 3、选择具有统计学差异的基因,比如pvalue<0.05甚至qvalue<0.05(另外如果样本类型异质性较高且在进行测序时生物学重复数偏低情况下,即使p值大于0.05,在其表达量尚可且变化幅度尚可的情况下也是可以考虑的); 4、选择表达量比较高的基因,在比较的样本组中,至少有一组样本的基因表达丰度(FPKM值)平均值大于10(不是绝对标准); 5、建议选择生物学重复样本间一致性较高的基因(对于异质性较高的样本,基因在生物学重复样本间可能也有明显差异,但差异倍数是实验组和对照组表达量平均值相除,一些异常表达量可能对差异倍数产生影响); 6、尽量选择多一些基因(15-30个)进行验证,看总体差异趋势情况。
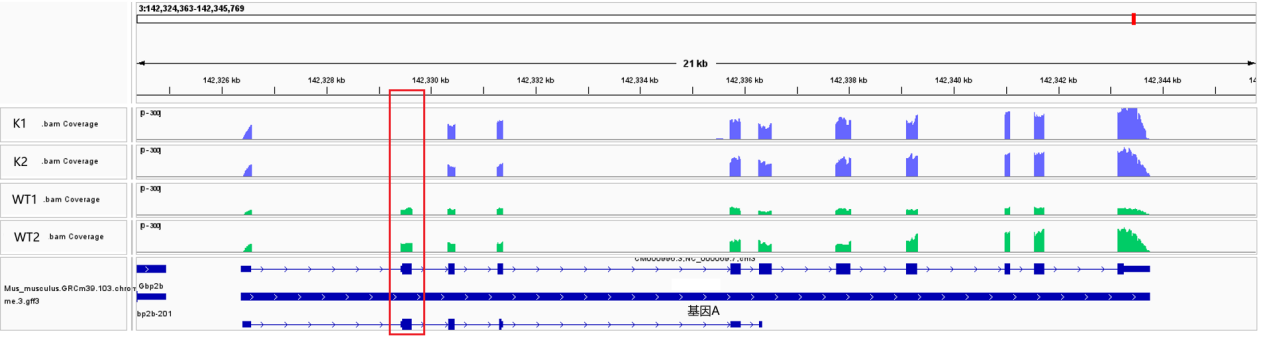
二、转录组和qPCR一致性 首先,我们需要对转录组和qPCR一致性有一个合理预期:这两个实验方法本身存在差异,因此存在一定程度的不一致性(约30-40%)是正常的情况[1]。在Huang J等人的研究中,出现转录组和qPCR结果的不一样(甚至趋势相反的情况),文章也对这种情况进行了探讨[2]。 因此在这里小派总结了几个可能影响转录组和qPCR一致性的原因: 1、样本是否为同一批次: 因为RNA的表达具有时空特异性,不同批次处理的样本之间本身可能存在一定的差异,所以做qPCR验证的时候建议是直接用转录组测序返样的RNA。并且当我们在转录组测序中发现非常明显的离群样本时候,避免这种个体特异性导致结果不准确,可以在做转录组分析和后续验证时候剔除这种离群样本。 2、比较组顺序是否反了: 在转录组分析中,文章中普遍存在两种不同的比较组命名方法,即 对照组 vs 处理组、处理组 vs 对照组,在分析差异的时候都是用 处理组/对照组 的。这两种命名上存在差异,可能是会导致差异的理解存在不同情况,在小派这里转录组项目的命名都是 对照组 vs 处理组。 3、qPCR操作是否规范: qPCR仪器的灵敏度不仅取决于仪器本身的技术规格,还受到实验操作规范性的影响。设置预实验,通过扩增曲线和溶解曲线来判断引物是否合适,是非常重要的步骤;并且在正式实验中,重复的设置也是不可缺少的一环。 4、引物设计是否合理: 在真核生物中,基因可能通过不同的剪切形式产生多条转录本,而各转录本的差异变化趋势也可能是不一致的,因此我们需要明确我们做qPCR是针对基因还是针对特定的转录本去验证。常规转录组分析是针对于整个基因定量的,因此可以针对多个转录本共有的外显子去设计引物,从而验证基因的表达;如果是对特定转录本分析的话,可能需要找到整个转录本特有的外显子部分设计引物验证。另一方面,设计完引物后,可以通过NCBI的Primer blast去看看设计的引物是否在基因组里是特异性扩增的。 5、基因选择是否合适: 验证的基因一般建议选择表达丰度较高的,因为对于表达丰度非常低的基因来说,本身测序和qPCR对其的定量就不一定非常可靠。另外对于基因组相同位置存在多个基因(例如相同位置存在正负2个方向的基因)或在基因组中存在多拷贝的基因,也并不建议选择去验证。选择的内参基因在不同样品里是否稳定表达,是另一个影响qPCR相对定量结果的关键因素。 6、两种技术本身存在差异: 转录组测序是基于检测样品里所有基因整体上的表达情况,qPCR是特异性设计引物对基因的局部区域做相对定量的,两者的技术原理是存在一定不同的,所以转录组和qPCR确实有可能存在并没有非常一致的情况的,一般来说主要看大部分基因的趋势一致就行的。 备注(一种比较特别的情况): 为什么敲除基因A的2号外显子后,K组的表达比WT组还高了? qPCR验证的时候一般是针对敲除部分去检测,所以能很好的验证敲除情况。而常规转录组是对整个基因定量的,虽然敲除了2号外显子,但其它外显子的转录层面可能并没有受到限制,并且由于剂量效应的影响,敲除了2号外显子后,可能刺激了其它外显子的表达,因此从转录组测序的结果来看可能就出来了敲除组的这个基因表达甚至相比对照上升了。 虽然是从转录组测序来看这个表达没有下调,但是敲除后,翻译成蛋白可能受到阻碍,因此结合qPCR的验证和IGV图,是可以充分表明敲除是没问题的哈。 IGV是使用可以参考教程:https://mp.weixin.qq.com/s/7-_l5aQ72p8vvk2Ot4gfZg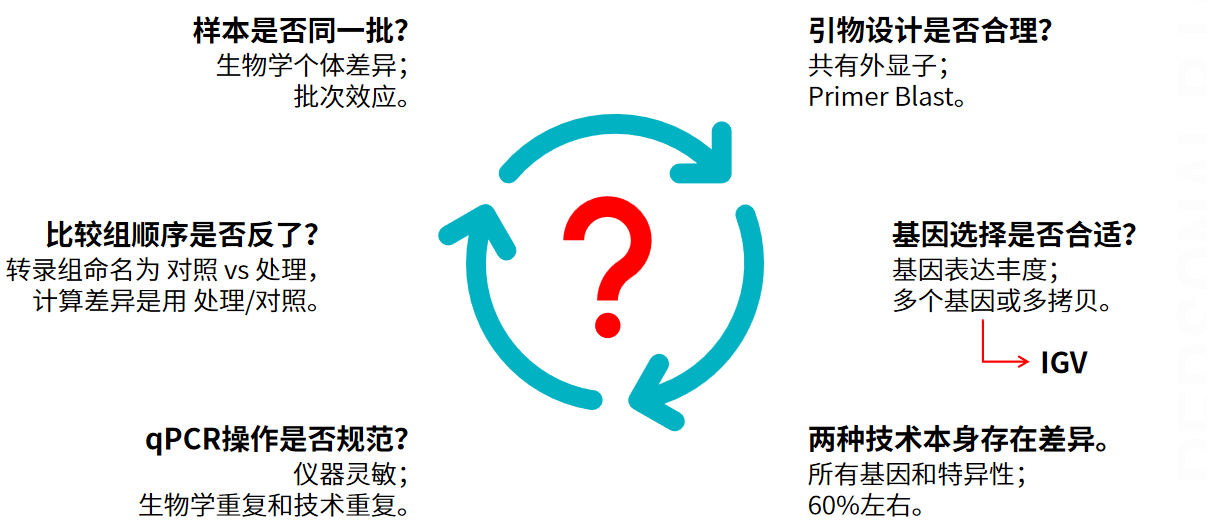
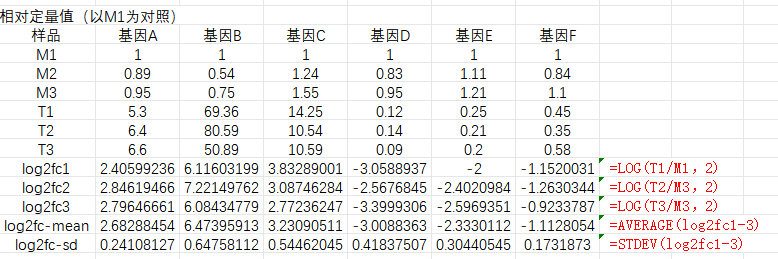
三、转录组和qPCR结果可视化 做完qPCR后,我们会得到基因在样品里的相对定量结果(一般使用2-∆∆Ct),而在转录组中也有这些基因log2fc结果,如下: 针对于这两个数据的展示,通常有以下几种图: 1、柱状图: a.基于qPCR的相对定量结果,引用excel的函数(log、average、stdev)求得log2fc和标准差; b.将qPCR和转录组的数据整理(qPCR和RNA-seq的log2fc、qPCR的标准差)成表; C.基于前3列用excel绘制柱状图,然后添加qPCR的误差线(自定义设置:±标准差)。 2、柱状图+折现图: a.基于1b里整理完的数据,在excel里插入组合图; B.给柱状图增加误差线,调整下,出图。 3、散点图(针对多比较组的结果,展示的更方便): a.基于1a里计算的qPCA的log2fc结果,与转录组的log2fc结果一一对应的整理数据; b.选中2列log2fc结果,用excel绘制散点图,添加趋势线(可以显示公式和R平方)。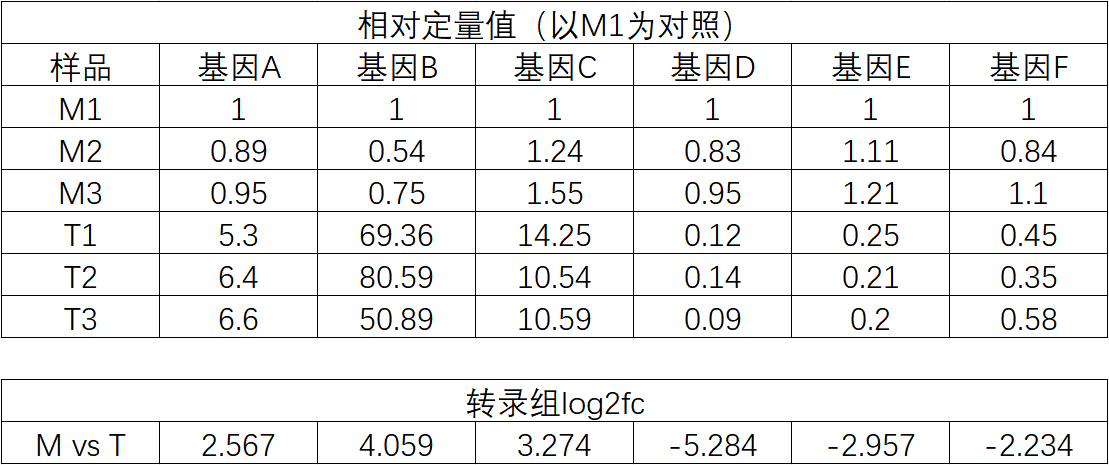
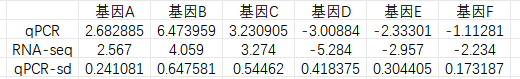
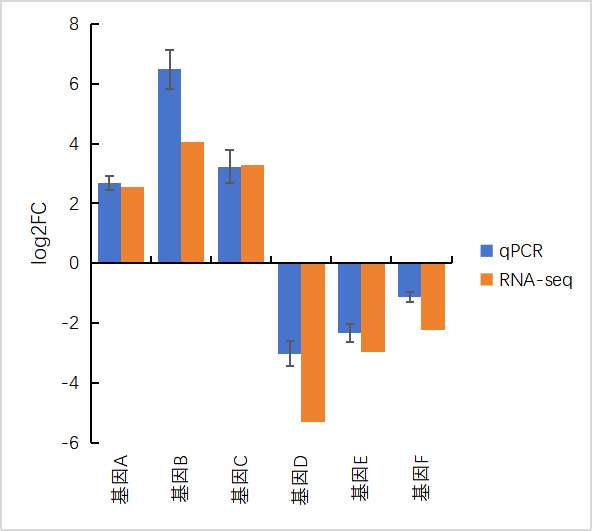
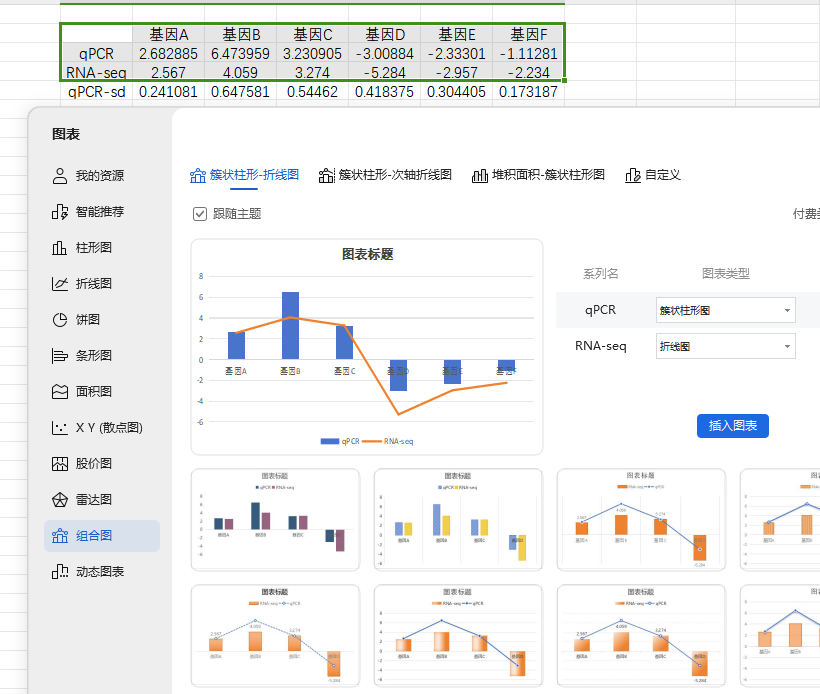
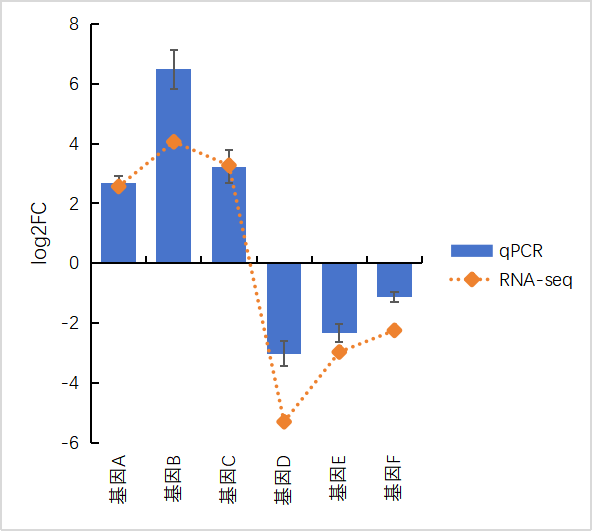
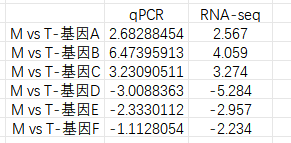
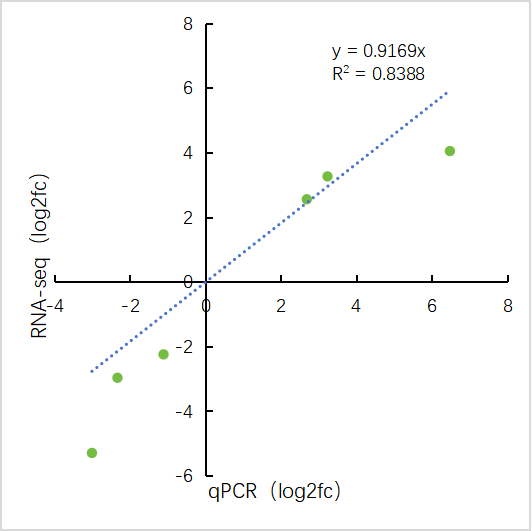
[1]Chen X, Yang X, Xie J, Ding W, Li Y, Yue Y, Wang L. Biochemical and Comparative Transcriptome Analyses Reveal Key Genes Involved in Major Metabolic Regulation Related to Colored Leaf Formation in Osmanthus fragrans 'Yinbi Shuanghui' during Development. Biomolecules. 2020 Apr 4;10(4):549. [2]Huang J, Zhou Q. Gene Biomarkers Related to Th17 Cells in Macular Edema of Diabetic Retinopathy: Cutting-Edge Comprehensive Bioinformatics Analysis and In Vivo Validation. Front Immunol. 2022 May 16;13:858972.