
2024-09-18

DNA复制是一个关键的生物学过程。在人类细胞中,成千上万的复制叉协同进行基因组的复制。这一过程的速率可能受基因组表观遗传状态的影响,并且在不同细胞类型之间,甚至同一细胞类型内部也会有所变化。基因组必须被准确复制,以避免发育缺陷和肿瘤等问题。遗传学和生物化学研究揭示了许多DNA复制因子及其相互作用,这些因子确保了基因组的高保真复制。为全面探测DNA复制过程,DNA测序技术被用于阐明复制的动态特征,如复制时间、复制叉的方向性、起始点以及DNA聚合酶的使用。此外,单分子方法也被广泛应用于监测单个复制叉的行为,包括复制速度和分叉失速现象。这些方法通过显微镜或长读测序进行观察,但由于它们通常从大量细胞中随机取样,因此对单个细胞之间复制动力学的差异较为不敏感。 2024年6月,Hubrecht研究所的Jeroen van den Berg和Alexander van Oudenaarden以共同通讯作者身份在Nature Methods上发表了题为“Quantifying DNA replication speeds in single cells by scEdU-seq”的研究论文,介绍了一种检测DNA复制速度的单细胞测序技术——单细胞5-乙炔基-2 ′-脱氧尿苷测序(scEdU-seq)。
scEdU-seq技术概述及用于揭示S期DNA复制谱
为了测量DNA复制叉动力学的异质性,作者开发了scEdU-seq技术,这是一种在单个细胞中鉴定复制新生DNA的测序方法。scEdU-seq技术利用了核苷酸类似物5-乙基-2'-脱氧尿苷(EdU)的代谢标记和新合成DNA片段的亲和力捕获。此外,scEdU-seq还能同时分析新生DNA和非新生DNA。 具体操作中,首先通过铜(I)催化的叠氮化物-炔环加成点击化学,将生物素部分共价连接到尿嘧啶碱基。点击反应后,将单个细胞分选到384孔板中进行处理。接着,使用限制性内切酶(Nla III)和末端修复酶(如大Klenow片段和多核苷酸激酶)消化单细胞基因组,将生成的片段连接到包含适配器、细胞特异性条形码和独特分子标识符(UMI)的连接体上。细胞池合并后,通过生物素捕获含有EdU的DNA分子,并通过热变性释放非EdU修饰的链。随后,通过Klenow介导的引物延伸和T7依赖性转录进行线性扩增。最后,将线性扩增的RNA通过逆转录(RT)转化为互补DNA,并用聚合酶链反应(PCR)进行扩增,最后便可准备进行Illumina测序。 研究人员在表达人端粒酶逆转录酶(hTERT)的RPE-1细胞中比较了scEdU-seq和Repli-Seq技术,发现两者获得的DNA复制谱有大量重叠(见图1b)。scEdU-seq的一个特点是能够同时分析新生DNA和非新生DNA,并观察到同一样本中,新生DNA和非新生DNA之间存在反相关性。随后,研究人员使用FACS富集S期细胞,并进行15分钟的EdU标记。通过对单个细胞的scEdU-seq信号进行分析,并依据细胞的复制时间对其进行排序,成功重建了S期进展。结果显示,基于scEdU-seq的S期进展能准确反映细胞周期的位置,表明该方法可以用来对细胞进行排序。 为了实现对单细胞S期进程的排序,作者从1,343个RPE-1 hTERT FUCCI细胞中构建了完整的S期DNA复制轨迹(见图1f)。观察到scEdU-seq轨迹的早期S期进展与EdU-seq-HU鉴定的DNA复制起始区位置重叠。研究人员通过拟合每个细胞的隐马尔可夫模型(HMM),识别了由相同复制体产生的复制延伸,发现每个RPE-1细胞中约有4,500个复制叉(见图1g),并且单条染色体在S期的DNA复制叉数遵循相似的趋势。灵敏度比较测试表明,scEdU-seq能检测到scRepli-Seq未能发现或检测较少的DNA复制叉。 图1. ScEdU-seq揭示了整个S期的有序DNA复制谱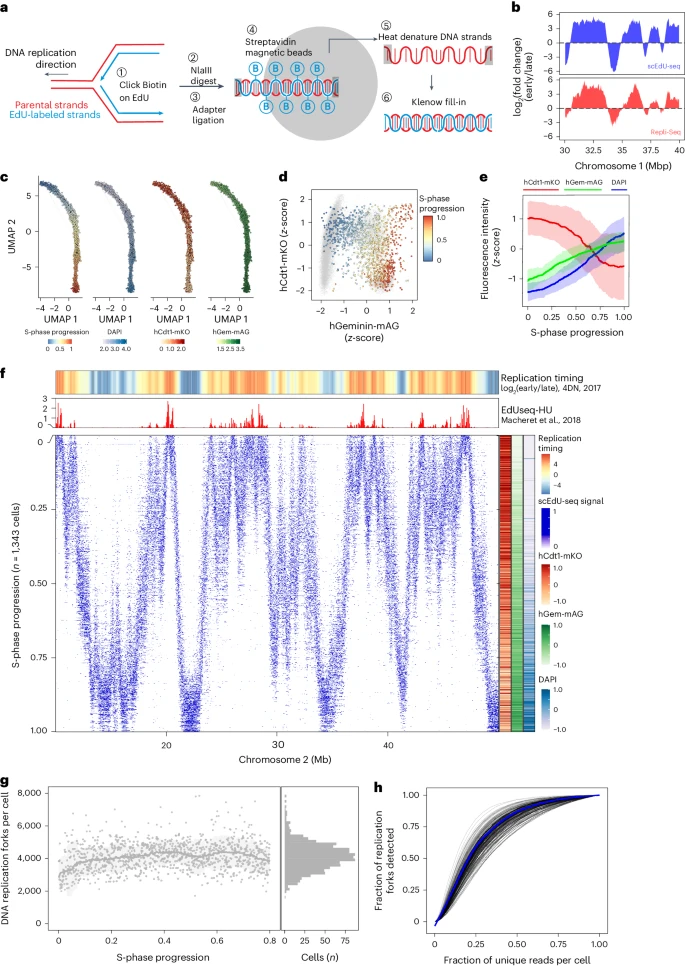
双脉冲scEdU-seq评估DNA复制速度
研究人员采取双脉冲EdU标记策略检测单细胞DNA复制速度。双脉冲EdU标记策略导致单细胞复制速度的准确性增加,并且对每个细胞恢复的唯一读取不太敏感。在接受两个EdU脉冲后,处于S期的单个细胞基因组被间隔一定距离的EdU片段修饰。暴露于单个EdU脉冲的单个细胞在Δx= 0时有最大值(图2b);暴露于双EdU脉冲(Δt = 45, 75或105 min)的细胞显示第二个最大值。随着Δt的增加,第二个最大值会向更大的Δx值偏移(图2b)。研究人员发现单细胞间DNA复制速度差异很大(~1.5倍差异,图2c, d),这种差异极大程度上与单细胞在S期的位置有关。 图2. 双脉冲scEdU-seq允许评估DNA复制速度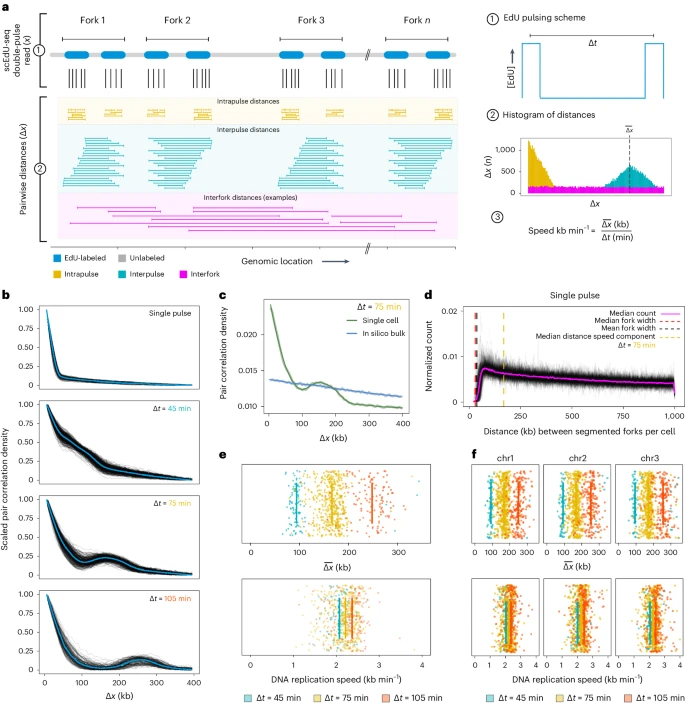
转录限制了早期S期DNA的复制速度
通过双EdU脉冲结合混合模型,研究人员测量了单细胞内DNA的复制速度。结果显示,单个细胞之间的复制速度差异约为1.5倍。总体上,DNA复制速度在S期稳步增加,表明整个S期的复制速度加快(见图3b)。这一观察结果通过对分选的早期和晚期S期RPE-1 hTERT细胞的DNA纤维分析得到确认。同时,类似的S期复制速度增加现象也在诱导的多能干细胞中被观察到。在人类细胞中,尚未观察到整个S期DNA复制速度的加速,而S期早期DNA复制速度降低的机制仍不明确。 先前的研究表明,早期复制的DNA通常靠近活跃的转录区域。为了量化S期的转录水平,研究人员使用了RPE-1细胞的单细胞新生RNA测序数据。结果显示,基因组中与scEdU-seq信号重叠的区域在S期初期的转录水平最高(见图3e、f)。此外,随着DNA复制进入S期,转录区域的数量和转录水平逐渐下降。早期S期的高转录水平与较低的DNA复制速度相关(见图3e、f)。实际上,S期早期的转录区域比非转录区域复制速度更慢(见图3e),这表明转录活动限制了S期早期的DNA复制速度。 图3 早期S期DNA的复制速度受到转录限制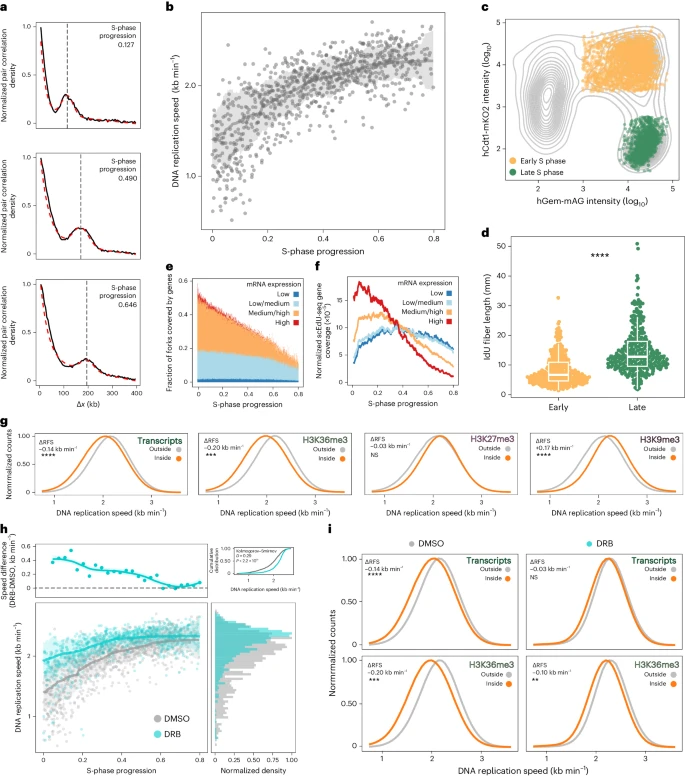
转录偶联损伤降低DNA复制速度
为了评估未激活RNAPII转录对DNA复制速度的影响,研究人员在两个EdU脉冲之间使用转录抑制剂5,6-二氯苯并咪唑-1-β-d-核糖呋喃苷处理细胞,此处理不会改变复制起始区或时间。结果显示,抑制RNAPII的细胞相比于接受二甲基亚砜处理的对照组,DNA复制速度总体上有所增加。进一步的实验通过流式细胞术和PARP-1过表达显示,S期的DNA复制速度加快。研究发现,RNAPII转录及其引发的DNA损伤在S期早期限制了复制速度,而PARP活性在调节响应转录相关DNA损伤的DNA复制速度方面起着关键作用。 图4 转录偶联损伤降低DNA复制速度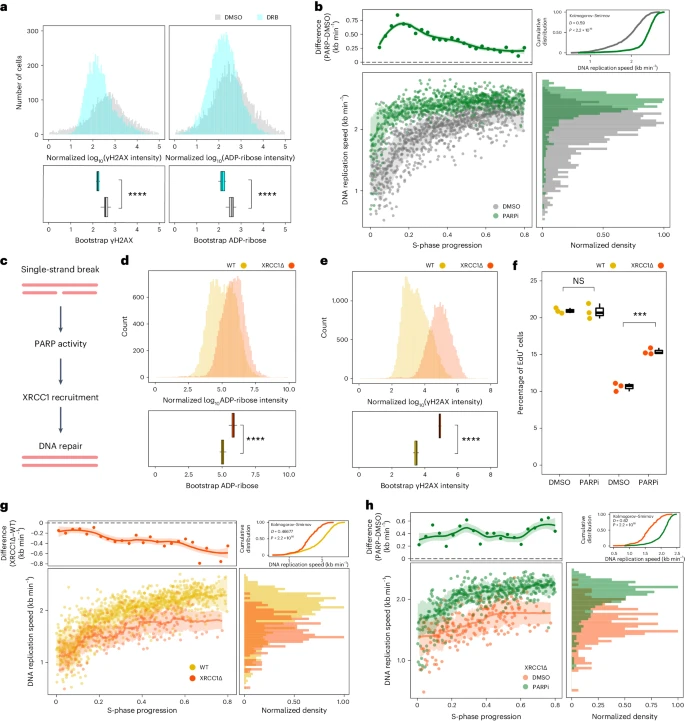
总结
综上所述,研究团队开发了一种单细胞测序技术——scEdU-seq用于分析单细胞DNA复制速度。基于该技术揭示了S期DNA复制速度的加速现象,并发现转录相关的DNA损伤通过增加PARP活性来降低DNA复制速度。该技术不仅具备低成本的优势,同时无需进行拷贝校正。尽管存在一些技术限制,scEdU-seq有望在多种生物系统中有效研究单细胞DNA的复制动力学和复制速度。