
2024-11-11

空间转录组学正在彻底改变我们对发育生物学、肿瘤学和疾病病理学的理解,它能够在原生组织环境中绘制复杂的基因表达模式。然而,目前该领域的研究主要集中在信使RNA(mRNA)表达的分析上。真核细胞的转录组包含各种动态和多样的RNA分子,不仅包括编码蛋白质合成的成熟mRNA,还包括RNA可变剪接体、小RNA和其他具有调控功能的非编码RNA。因此,对不同种类的RNA分子在其生命周期中的空间分析对于解析复杂组织中的RNA生物学全貌至关重要。
此外,在临床实践中,福尔马林固定石蜡包埋(Formalin-fixed Paraffin-embedded,FFPE)组织是病理学诊断的基石。全世界的临床病理科积累了大量FFPE组织块,这些材料与详细的临床数据相结合,构成了尚未充分利用的人体生物学和转化研究的宝贵资源。然而,FFPE样本也带来了特定挑战。在石蜡包埋过程中,样本中的RNA高度片段化,并且在室温储存条件下进一步降解。因此,对临床级FFPE样本中的RNA分子进行空间分析极为困难。
2024年9月30日,来自耶鲁大学樊荣团队的研究人员在Cell发表了题为“Spatially exploring RNA biology in archival formalin-fixed paraffin-embedded tissues”的研究论文。该平台巧妙地利用FFPE样本中自然发生的RNA片段化,向广泛多样的RNA分子原位添加poly(A)尾,结合微流控条形码标记和算法创新,成功实现了对临床存档FFPE组织的全覆盖、逐碱基的空间全转录组测序。Patho-DBiT通过对完整mRNA、缺失poly(A)尾的片段化mRNA、各类大/小非编码RNA、剪接异构体以及携带单核苷酸变异(SNV)的前体RNA进行空间条形码标记,精确解码了FFPE复杂组织中丰富的RNA生物学信息,包括单细胞级mRNA图谱、非编码RNA表达、可变剪接、遗传变异、microRNA调控及RNA动态变化等。
技术原理
Patho-DBIT技术利用FFPE样本中自然发生的RNA片段化过程。在组织切片制备过程中,样本中的RNA经历不同程度的片段化。通过向这些片段化的RNA分子原位添加poly(A)尾,增强了这些RNA分子的标记和稳定性,使其能够在后续步骤中进行有效的测序。通过微流控技术,Patho-DBIT能够在组织样本中以极高的分辨率(可达100×100的网格)进行条形码标记。每个标记的RNA分子都被赋予一个独特的条形码,用于后续的空间定位和数据分析(图1)。结合创新的算法,Patho-DBIT能够精确解码FFPE组织样本中丰富的RNA生物学信息,包括完整的mRNA、片段化mRNA、非编码RNA、剪接异构体以及携带单核苷酸变异(SNV)的前体RNA,从而精准解码FFPE复杂组织中的丰富RNA生物学信息,包括单细胞级mRNA表达谱、非编码RNA表达、可变剪接、遗传变异、microRNA调控以及RNA动态变化等。
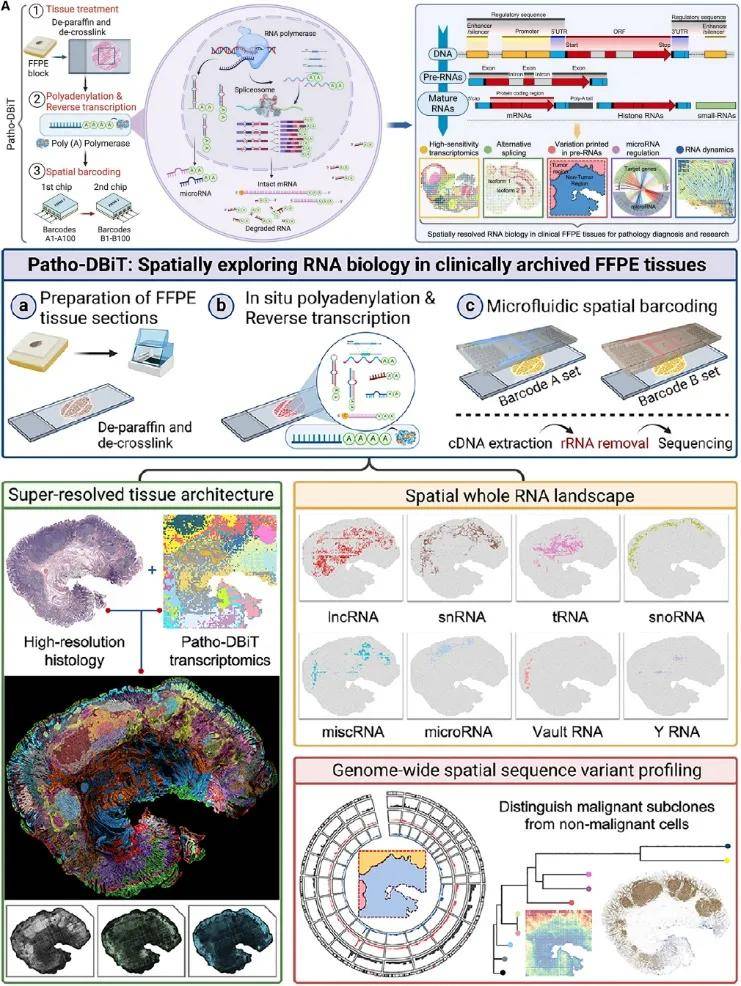
图1 Patho-DBiT技术流程及全场景空间分析能力
研究内容
1、小鼠胚胎Patho-DBiT设计及空间全转录组定位
研究中使用50 mm像素大小的装置将Patho-DBiT应用于E13小鼠胚胎FFPE切片。无监督聚类显示了20个转录组簇,空间UMAP与H&E染色的相邻切片的组织学紧密一致。Patho-DBiT平均每像素检测到5,480个基因和15,381个独特的分子标识符(UMIs),全基因组泛mRNA和UMI图谱显示出与组织形态和密度的强烈一致性。在相邻切片上进行的重复重复性非常高。
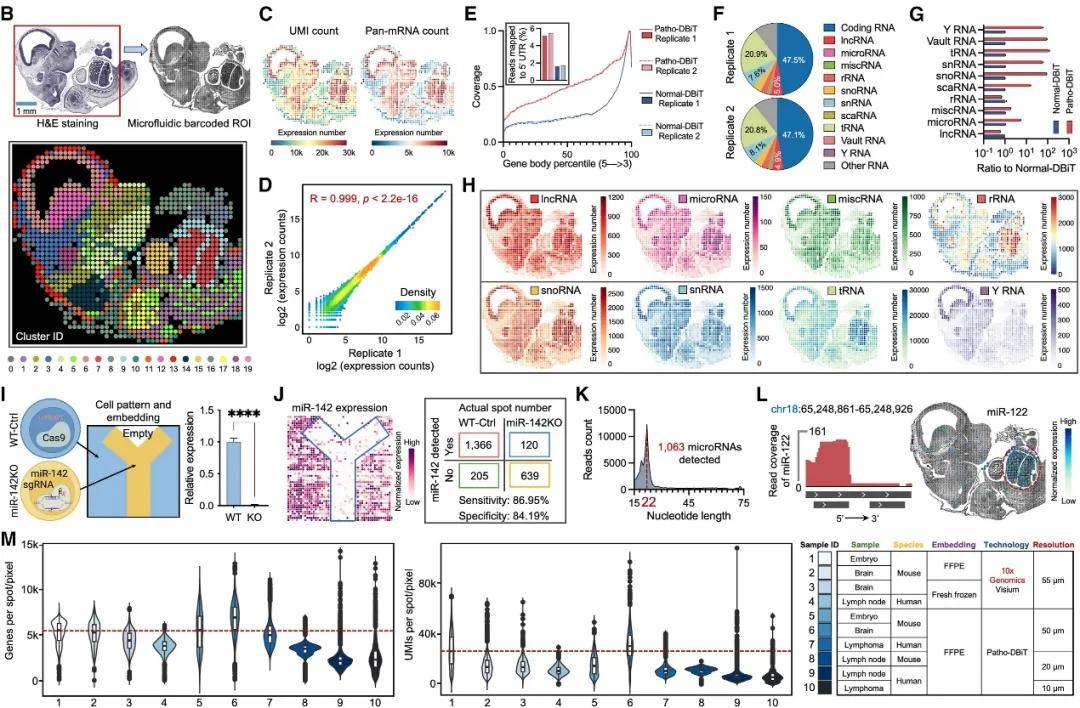
图2 Patho-DBiT在E13小鼠胚胎切片上的表现
2、小鼠大脑中区域特异性替代剪接和A-to-I RNA编辑的空间共谱分析
研究团队在不同组织类型和空间分辨率下对Patho-DBiT进行了充分验证。在小鼠脑冠状切片的分析中,Patho-DBiT在50 μm的空间像素中平均检测到6786个基因和31063个独特分子标记(UMI)。聚类分析精确重构了大脑组织的解剖结构,其空间排列与Allen小鼠大脑图谱上相似切片的区域注释高度一致。此外,Patho-DBiT捕获了调控大脑重要功能的基因及其区域性剪接异构体的表达,以至少需要2个剪接连接跨越读数(splice-junction-spanning read counts)为标准,研究人员在2,368个基因中共检测到3,879个不同的可变剪接事件,包括外显子跳跃(SE)、内含子保留(RI)、3'选择性剪接位点(A3SS)、5'选择性剪接位点(A5SS)和外显子互斥(MXE)等,其空间分布与一套独立的、通过长读长纳米孔测序获得的新鲜冷冻脑样本数据相吻合。
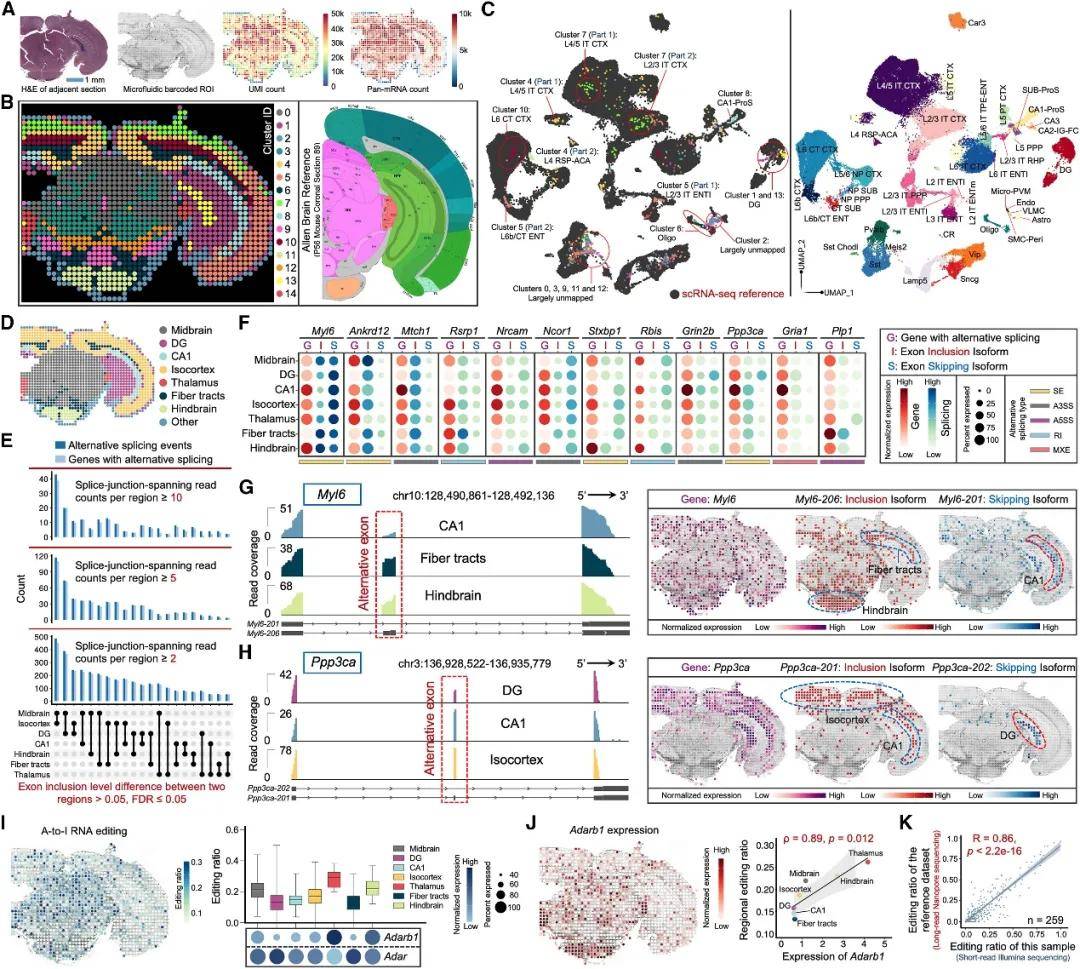
图3 小鼠脑基因表达和RNA加工的空间共定位
3、Patho-DBiT概述了临床档案AITL样本的淋巴瘤发生生物学
接下来,作者将空间分析扩展到临床存档的FFPE 组织。无监督聚类揭示了10个空间组织簇与组织学结构对齐,UMAP平均包含5364个基因,每像素11989个UMAP,描述了标准标记定义的不同细胞类型。为了评估这种长期储存的FFPE切片的捕获准确性,研究团队使用索引共检测(CODEX)法对相邻组织切片进行了高复杂性空间细胞分型。
接下来,作者将空间分析扩展到临床存档的FFPE组织。使用Patho-DBiT对一名诊断为血管免疫母细胞T细胞淋巴瘤(AITL)的患者皮下结节获得的组织切片进行条形码(图4)。这个嵌块在进行实验之前,已经在室温下保存了5年多。无监督聚类显示了10个空间组织的聚类与组织学结构对齐。UMAP平均每像素有5364个基因和11989个UMIs,描述了由典型标记定义的不同细胞类型。为了评估这个长期存储的FFPE切片的捕获准确性,我们使用索引(CODEX)共检测对相邻组织切片进行了高倍空间细胞分型。增殖标志物MKI67和PDCD1经常在恶性AITL细胞上表达,在本切片中呈致密的结节状分布,与CODEX鉴定一致。

图4 储存5年的AITL样本的高灵敏度空间转录组学
4、通过整合扩展Patho-DBiT与高分辨率组织学的超分辨率组织结构
研究团队使用了100×100微流控设备,以20 μm的像素分辨率对10,000个点进行条形码标记,成功对一例诊断为黏膜相关淋巴组织边缘区淋巴瘤(MALT)的临床肿瘤活检样本进行了空间解析。该样本已在室温下存放超过三年。通过Patho-DBiT平台,团队在一次实验中获得了该样本的多重信息。对基因表达矩阵进行无监督聚类分析后,识别出了20个亚群,这些亚群的空间分布与组织学结构高度一致。即使是数量较少的特定细胞类型,Patho-DBiT也能够精确检测,并通过免疫荧光染色进一步验证了结果的可靠性。
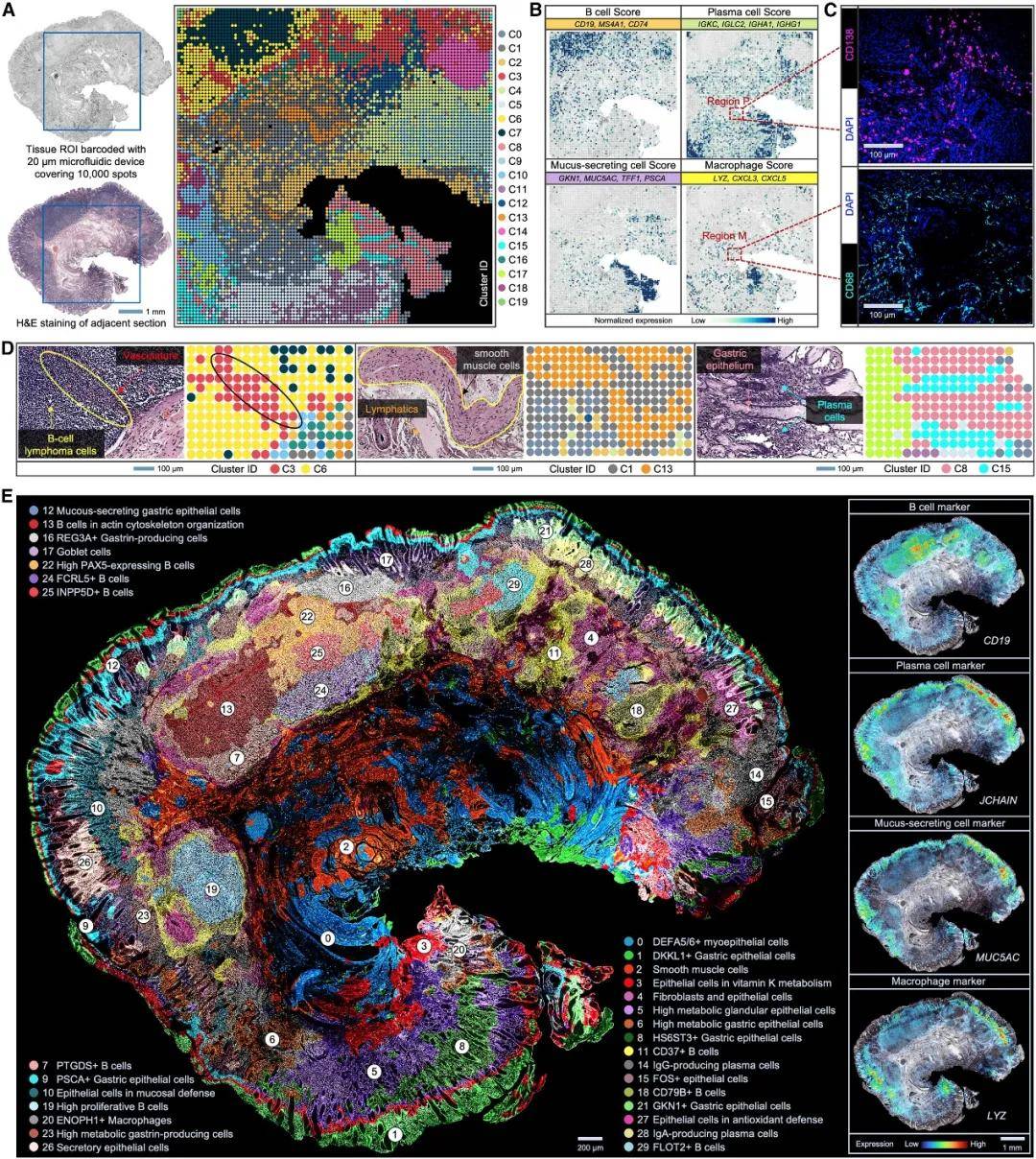
图5 通过将病理DBiT与组织学相结合,解决了高分辨率的组织结构问题
5、用于肿瘤鉴别的基因组尺度空间序列变异分析
序列变异在RNA转录本中很常见,在很大程度上反映了潜在的基因突变或RNA编辑。研究团队进一步使用最新开发的计算平台iStar,将分层组织学图像特征提取与转录组学数据相融合,成功实现了单细胞水平的空间基因表达映射,大幅提升了空间细胞异质性的解析能力(图6)。其次,Patho-DBiT能够捕获全基因组范围内的序列变异,并生成空间SNV表达矩阵。它无需依赖已知标记物,通过无监督聚类即可区分肿瘤亚克隆与非恶性细胞,并预测克隆进化关系。此外,该MALT组织的测序数据中有35%的序列映射到了非编码RNA,Patho-DBiT捕捉到了从数十到数千种不同非编码RNA分子的空间分布及其与肿瘤发生的关系。
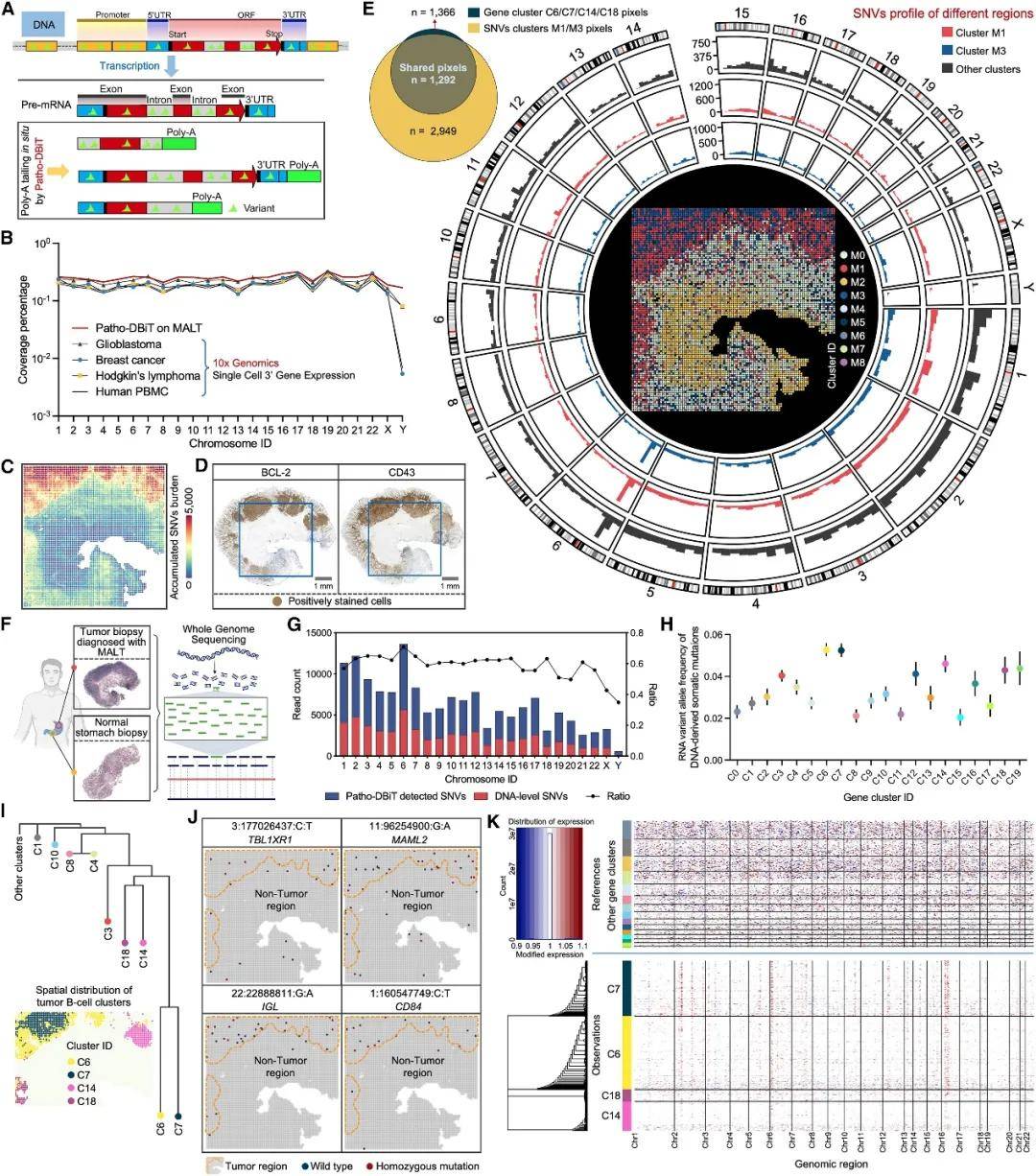
图6 用于区分恶性亚克隆的全基因组空间变异分析
总 结
Patho-DBiT技术具有广泛应用性,不仅可用于小RNA和miRNA的空间表达谱,还可用于探索RNA后转录修饰与空间基因表达模式。它展现了从冷冻组织到陈旧FFPE样本的兼容性。此外,在FFPE样本上具有巨大潜力,该技术在FFPE样本上展示了极高的空间分辨率,为过去的病理样本提供了重要的分析机会。例如,在分析5年储存的AITL样本和3年储存的MALT组织时,该技术展示了对基因表达、基因组突变、小RNA网络和剪接动态的多层次分析。未来在临床的应用中,Patho-DBiT可深入了解不同肿瘤细胞在不同微环境中的存活机制,帮助识别病变进展和耐药性的特定癌细胞基因型或克隆型。这可能帮助个体化治疗方案的制定,如探索特定药物(如ROCK1抑制剂)对特定患者的潜在疗效。