
2024-12-21

RNA Velocity(RNA速率分析)是一种能够重塑RNA转录、剪接与降解过程的动力学分析方法,通过比较未剪接的前体mRNA和剪接后成熟mRNA的相对含量,来分析mRNA的剪接和降解速率。根据剪接前后mRNA丰度的动态变化,可以预测单细胞转录状态的变化趋势和细胞命运。目前常用的转录组分析方法是基于Oligo-dT引物捕获Poly(A)尾的mRNA,主要检测和分析的是剪接之后的RNA,而忽略了细胞中未剪接RNA的信息。因此,同时分析单细胞中剪接与未剪接的RNA能够更精准地重塑单细胞中的RNA动力学过程。
2024年7月,美国贝勒医学院的Chenghang Zong在Nature Communications杂志在线发表题为“Single-cell total-RNA profiling unveils regulatory hubs of transcription factors”研究论文。本研究提出了一种基于多重退火循环扩增引物的单细胞SnapTotal-seq测序方法,具有同时检测单细胞中前体RNA与剪接后RNA的能力。
技术原理
文中开发了SnapTotal-seq方法,可以进行单细胞的全转录组测序。如图1a,SnapTotal-seq使用了多重退火循环扩增的引物,在低温下可实现引物与单细胞中RNA的随机结合,逆转录后引物序列离开RNA,实现模板转换,并进行后续的文库构建,使用了Cell Barcodes与UMI来进行细胞和分子的区分与计数。在1 M reads的深度下,SnapTotal-seq基于外显子与内含子的reads都能够检测到一万多种基因,说明此方法可高效捕获到剪接与未剪接的RNA(如图1b)。利用RNA Velocity分析,SnapTotal-seq成功重构了293T细胞的细胞周期动态(G2/M→G1→S→G2/M)。与其他单细胞转录组技术相比,SnapTotal-seq在细胞周期动态转化的重构上优于基于Poly(A)尾RNA捕获的CEL-seq2和Smart-seq3,而与VASA-seq相当,后者也能较为精确地重构细胞周期动态。
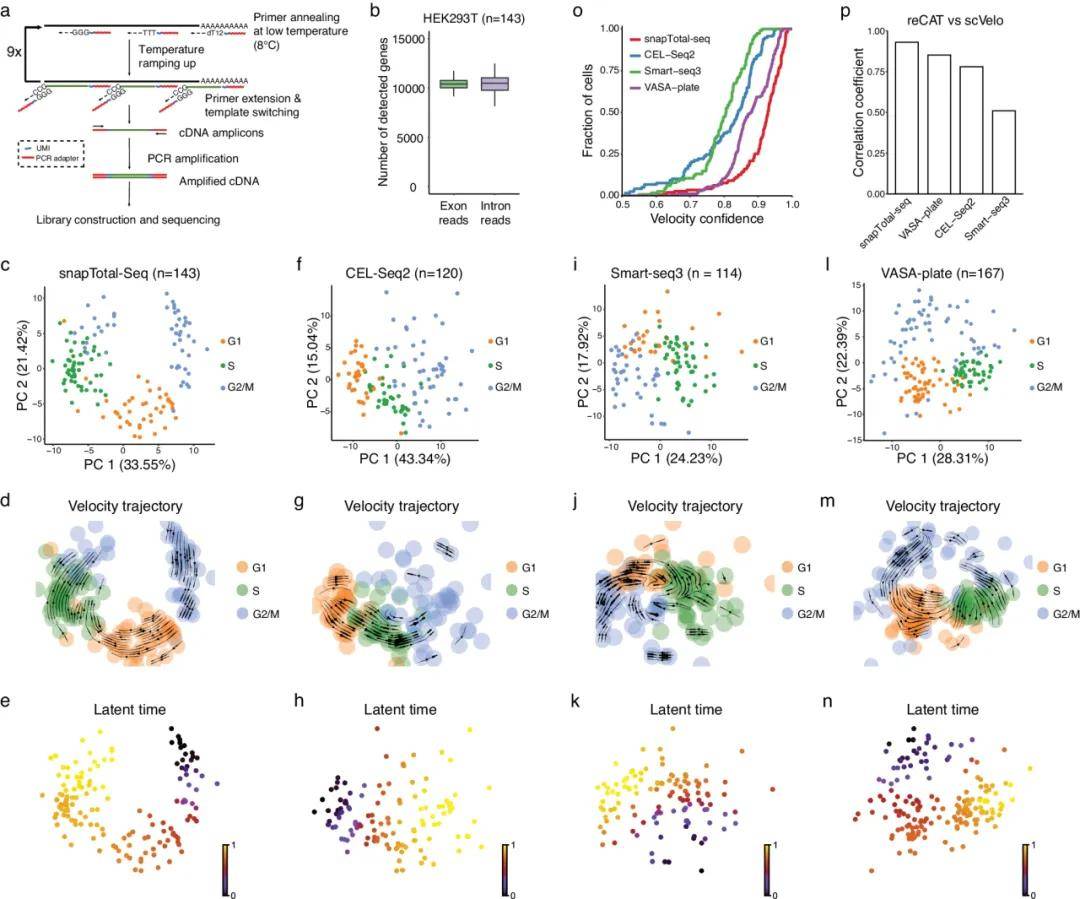 图1 SnapTotal-seq方法及其效果验证
图1 SnapTotal-seq方法及其效果验证
实验结果
1、确定细胞周期进程中的差异表达基因
研究根据细胞周期中基因表达的变化,将差异基因分为三类:Type I、Type II和Type III。Type I基因在外显子和内含子的reads丰度上都有显著变化,可能由转录过程调控;Type II基因只有剪接后的RNA存在显著变化;Type III基因则仅未剪接的RNA有显著变化,可能由转录后的RNA剪接或修饰过程调控。进一步分析发现,Type I基因在不同细胞状态中的表达变化分为五种模式,剪接和未剪接RNA的变化具有较强的相关性,且两者变化存在微小的延迟。为验证差异表达基因与细胞周期的关系,研究人员通过qRT-PCR检测了G1、S/G2、G2/M时期细胞中的8个Type I基因,结果表明这些基因的表达差异与细胞周期密切相关。研究还发现,超过50%的Type I基因是尚未被报道的细胞周期相关基因,这些基因主要参与有机氮化合物的合成和碳水化合物代谢等细胞功能通路。
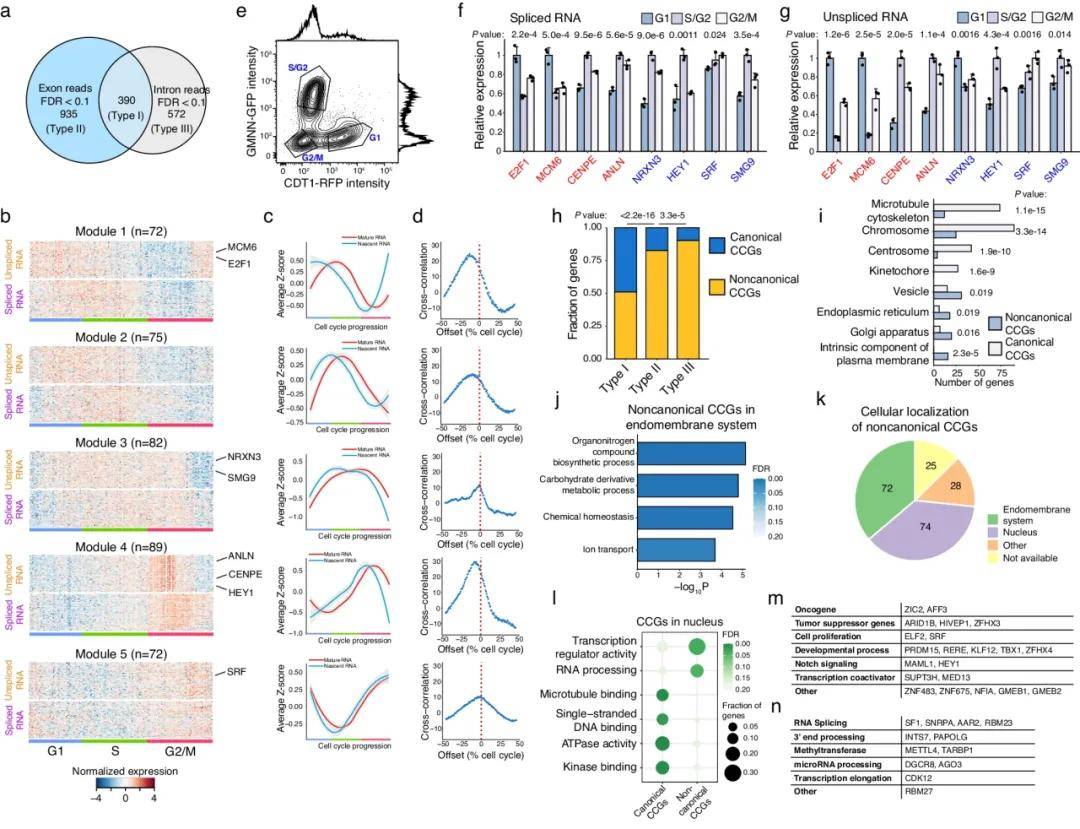 图2 I型细胞周期基因(CCGs)的功能分析
图2 I型细胞周期基因(CCGs)的功能分析
2、鉴定细胞周期进程中的关键转录因子
Type Ⅰ的基因随着细胞周期表达的变化很可能是由于DNA到RNA的转录过程影响的,而调控转录过程的一个重要因素是转录因子(Transcriptional Factor, TF)——一种通过与DNA相互作用在转录水平参与基因表达调控的蛋白质。为了筛选出调节细胞周期进程的关键TF,利用回归算法研究TF的剪接后RNA表达水平与靶基因未剪接RNA转录水平的相关性,检测出Type I中的23个TF的潜在下游的靶基因,根据靶基因的转录活性分成4个Hub(图3a-d)。使用LASSO 回归分析鉴定23个TF之间的互相调控作用(图3e)。
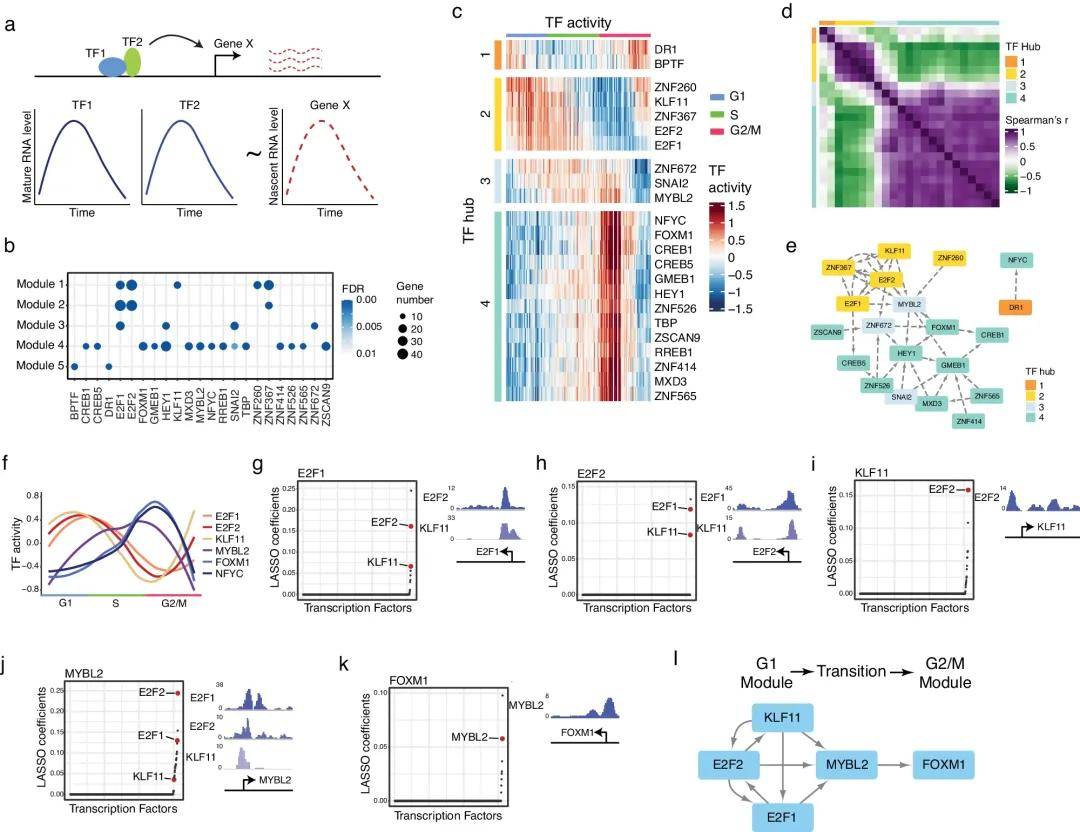 图3 重建细胞周期转录因子(TF)调控网络
图3 重建细胞周期转录因子(TF)调控网络
3、鉴定细胞周期进程中的关键RNA结合蛋白
Type II基因的表达主要受转录后调节的控制,表现为RNA丰度差异。根据这些基因的表达活性变化,作者将其分为五种动力学表达模式,并对每种模式进行了GO富集分析。研究发现,RNA结合蛋白(RBPs)在调控Type II基因表达中起着重要作用,特别是有8种RBPs与Type II基因的表达相关。进一步分析发现,增强RNA稳定性的BCLAF1和G3BP1与其靶基因表达呈正相关,而促进RNA降解的PUM1与其靶基因表达呈负相关。由于PUM1和G3BP1在细胞周期中的作用,这两个基因的敲除会导致细胞存活率显著下降。
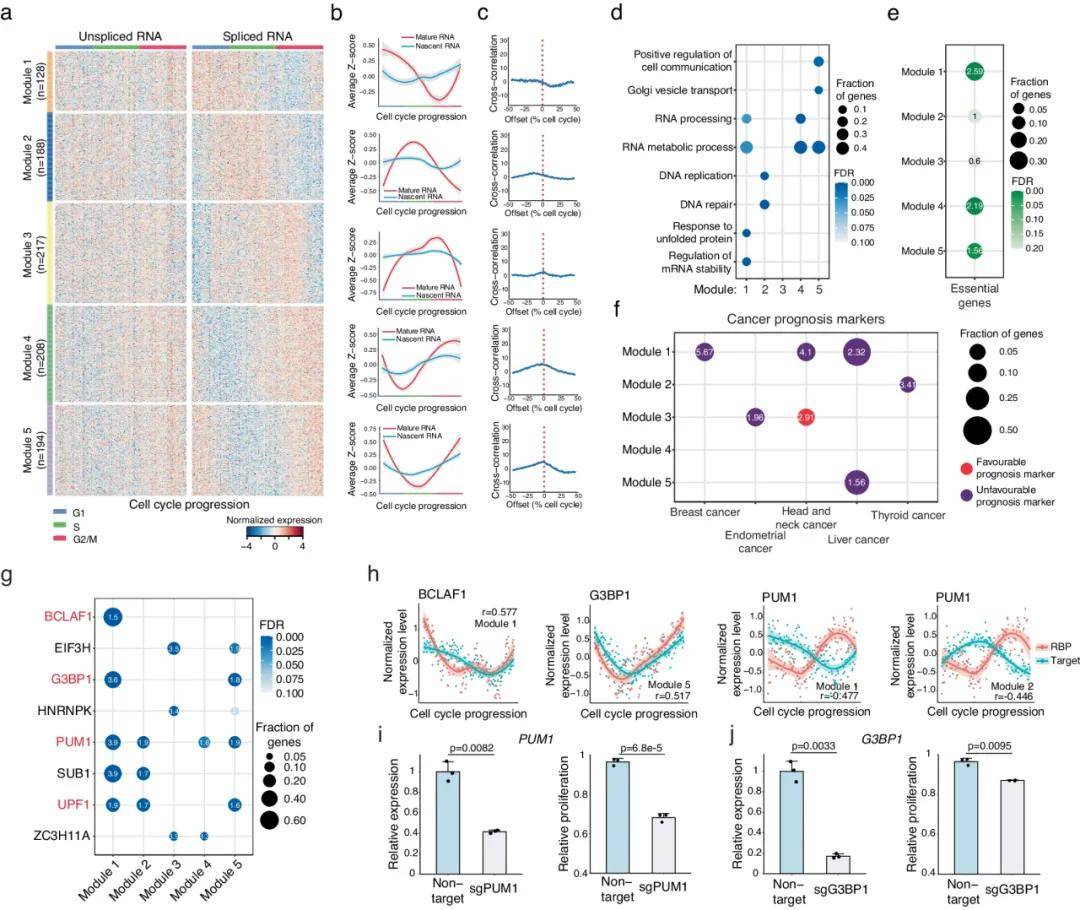 图4 II型细胞周期基因(CCGs)的转录后调控
图4 II型细胞周期基因(CCGs)的转录后调控
4、确定OIS进程中的差异表达基因与转录因子调控网络
接下来,作者将SnapTotal-seq应用于对肿瘤基因诱导的细胞衰老死亡过程(Oncogene-Induced Senescence, OIS)的研究中(图5a)。通过诱导胰腺癌Oncogene KRASG12D的表达,并收集0、1、2、3、5天的人胰腺导管细胞进行SnapTotal-seq分析。由细胞的分群结果可知,在KRASG12D基因表达之后细胞逐渐进入G0期(图5b-d)。与前文的思路类似,作者依照剪接与未剪接RNA丰度变化将差异基因分成三种类型,并详细研究其中Type Ⅰ基因的表达丰度变化(图5e-g),选出了112个可能的TF,并根据TF的活性分为3个Hub。其中Hub2与其他Hub具有更多的上下游调控关系,说明Hub2在细胞的状态转变中可能起到比较关键的作用(图5h-j)。
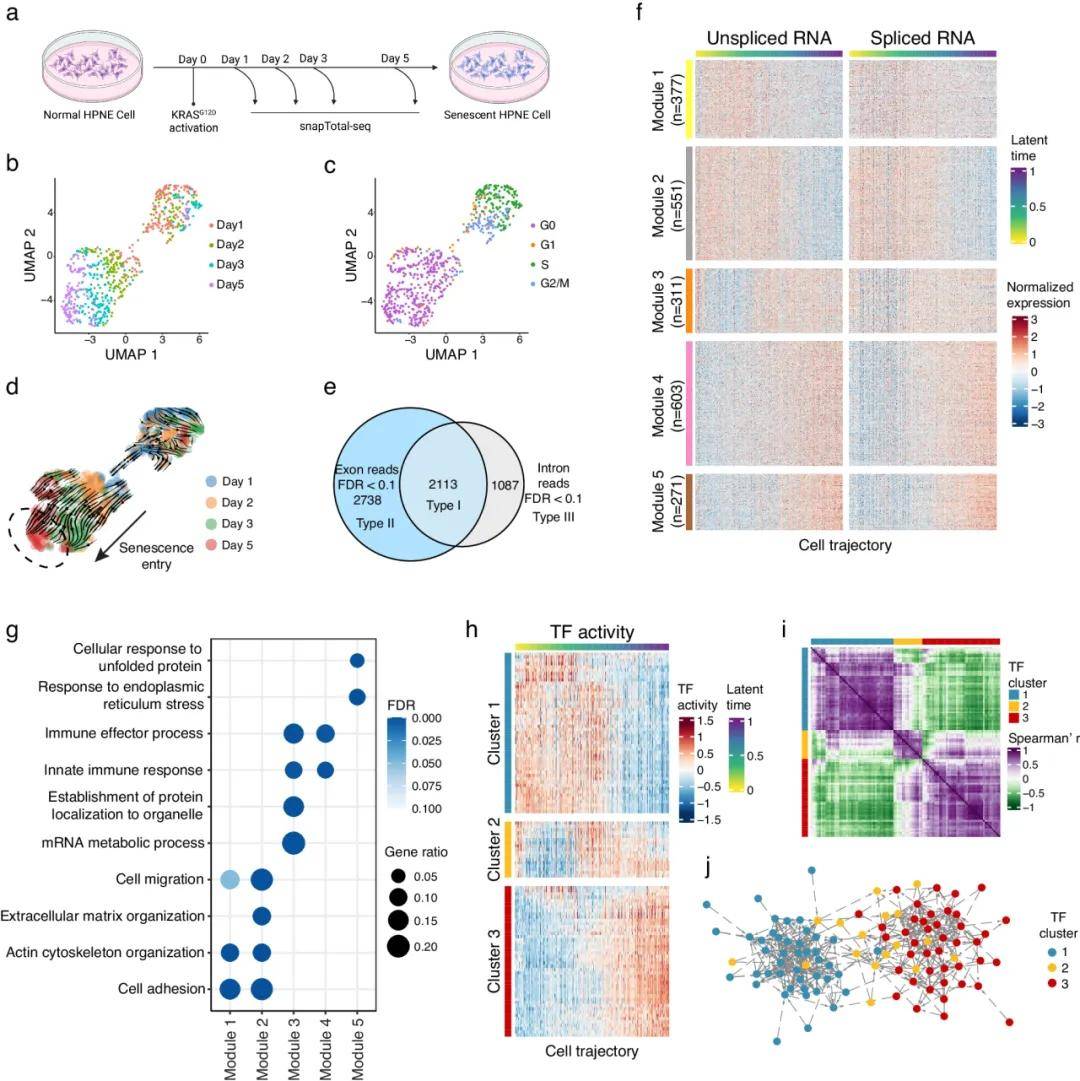 图5 癌基因诱导衰老过程中的转录动力学
图5 癌基因诱导衰老过程中的转录动力学
5、鉴定OIS进程中的关键转录因子
为了得到更有说服力的结果,文章参考了ChIP-seq数据库,发现112个TF中有25个TF的下游靶基因也在ChIP-seq中被显著富集(图6a)。其中,随着细胞进入G0期,Hub3的靶基因表达越来越活跃,根据靶基因表达表达的活跃程度,Hub3的TF进一步被分为两个亚类,两个亚类的靶基因分别作用于免疫反应和应激反应的调节,说明了不同TF在肿瘤基因诱导的衰老死亡中具有独特的功能。通过TF调控网络的构建:发现Hub3.1调控网络与Hub2中的TF有关;而Hub3.2的调控网络中,ATF4是上游TF,级联的调控网络说明了ATF4在细胞的应激反应通路中具有核心作用(图6b-e)。
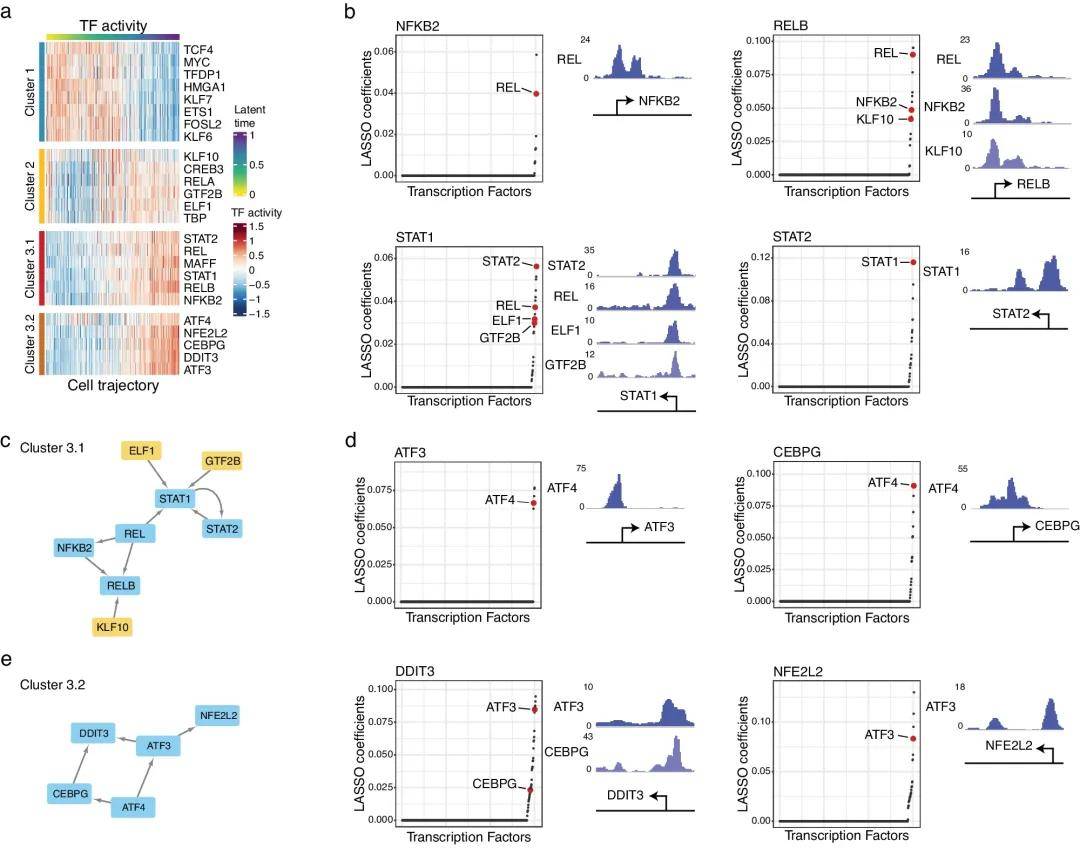 图6 识别推动OIS的关键TF监管网络
图6 识别推动OIS的关键TF监管网络
总 结
研究中通过将多重退火化学和模板切换化学结合到一步反应中,开发了一种高灵敏度的单细胞总RNA-seq方法(snapTotal-seq)。与基于寡核苷酸的方法相比,通过有效检测未剪接RNA和剪接RNA,snapTotal-seq在重建细胞周期进展的潜在动态过程方面显示出显著的优势。该技术可以提升RNA速率的分析效果,较为精确地重构细胞周期的动态变化。snapTotal-seq的开发和应用为基因调控研究提供了新的工具和方法,其在细胞周期和抗癌研究中的成功应用展示了其巨大的潜力。