文章题目:Transfer of antibiotic resistance genes from soil to rice in paddy field
期刊:Environment International
发表时间:2024年8月
研究背景
抗生素抗性基因(ARGs)作为新兴环境污染物,因超级病菌出现已成全球性问题。由于自然发展及人类活动,土壤被认为是ARGs和抗生素抗性菌(ARB)的储藏库。编码结构和功能特征的基因通常与土壤微生物的内在抗性有关,人为因素如灌溉、施肥、耕作、大气沉降等可能将ARGs和ARB引入耕作土壤,且土壤能塑造微生物群落和抗生素抗性谱,是研究抗生素耐药基因的关键基质。
抗生素抗性基因(ARGs)的全球传播和分布备受关注,然而,关于ARGs从一个基质向另一个基质传播的资料仍然不足。种植作物的土壤关乎人类健康,农田土壤中ARGs和ARB向作物潜在转移会威胁食品安全和人类健康。水稻是全球重要粮食作物,目前,为了获得更高的产量,会过度使用化学和有机肥料,施肥会增加稻田土壤中ARGs和MGEs的多样性和丰度。因此,采集了生长季分别施用猪粪堆肥、化肥及未施肥情况下的土壤和水稻样本,采用高通量实时定量PCR技术,以探究ARGs在土壤及水稻不同部位的变化、传播途径以及施肥对水稻食用部分ARGs谱系的影响。
研究思路
研究地点的选择及处理
选择作物、施肥和管理方式一直保持不变的试验田。试验田包含三种处理:化肥组(CF)、堆肥组(CM)和对照组(CK)。每种处理使用三块地(30平方米),并使用水泥墙防止交叉污染。灌溉用水来自靠近稻田的一条河流。
采样和DNA提取
收集第一个作物生长季的土壤和水稻样本。
土壤样本:在每个试验田中随机设置了五个采样点,并收集了0~15cm的表层土壤,然后将其均匀混合;
植物样本:从每个试验田中收集了五株完整的水稻,将这些水稻秧苗被移植到不同的施肥地块(堆肥猪粪、化肥和无肥)。
分别采集第0天、第7天、第30天和第79天的土壤和植物样本,每个样本都有三个重复样本。还收集了堆肥猪粪和灌溉水的样本,分别提取DNA。
ARGs和MGEs的高通量QPCR,并计算相对丰度和基因拷贝数
16S rRNA基因的高通量测序
数据分析
研究结果展示
土壤和水稻植株中ARG的多样性
在针对猪粪堆肥、土壤以及植物样本所开展的研究中,共计观测到了76种抗生素抗性基因(ARGs)亚型与9种MGEs亚型。
不同样本中的抗生素抗性基因类型:
猪粪堆肥:对氨基糖苷类、四环素类以及磺胺类等耐药基因较为常见(见图S1)。
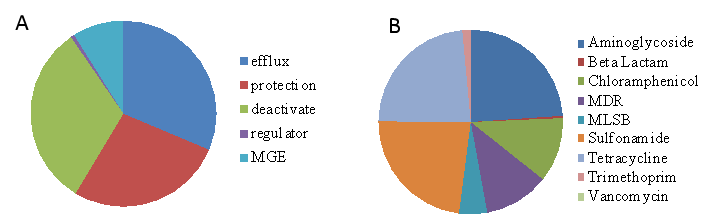 图S1该稻田使用的猪堆肥中ARG的主要机制(A)和靶向抗生素(B)。
图S1该稻田使用的猪堆肥中ARG的主要机制(A)和靶向抗生素(B)。
土壤:无论是否施肥,多药耐药基因(主要是 mexF)数量最多(见图1A),表明施用堆肥未影响稻田土壤中主要的抗生素抗性类型。
不同处理下地下部分样本的ARGs多样性:
在CM处理下,ARGs的Shannon多样性指数稳定;但是CK和CF处理时,该指数随水稻生长下降。施用堆肥猪粪可使水稻地下部分的ARGs多样性增加,这可能表明,由于肥料、土壤以及植物微生物群落之间相互融合,可能存在ARGs从堆肥粪肥向水稻植株转移的可能性。CF和CK处理中,第79天与第0天对比,水稻地下部分和地上部分的ARGs的Shannon多样性指数显著下降,预示稻田轮作中水稻植株内部ARGs多样性可能明显降低。
不同处理下地上部分样本的ARGs多样性:
在CM、CF以及CK处理下,ARGs的Shannon多样性指数无明显差异,可能不同施肥方式对内生ARGs影响小。
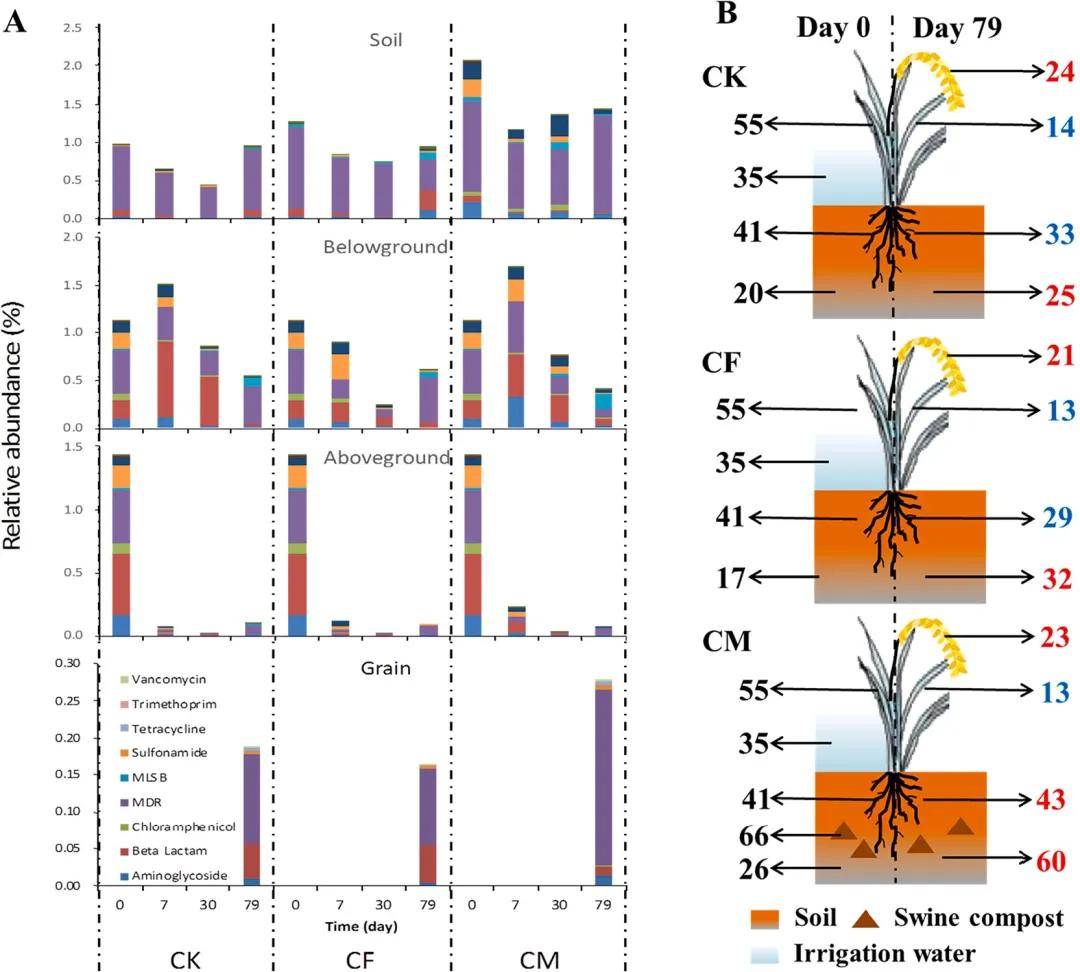
图1. ARGs在土壤、水稻植株和稻米中的主要靶向抗生素(A)。ARG的初始来源和ARG在土壤-植物(水稻)系统中的最终分布(B)。黑色数字代表土壤-植物(水稻)系统各部分ARG的初始检测数量。红色和蓝色数字分别表示ARG在相应部分的检测数量增加和减少。CK、CF和CM代表了稻田中不施肥、化肥和猪粪堆肥的不同处理。
植物中ARGs分布特征:
植物(包括地下和地上部分)中ARGs的分布特征与土壤截然不同(图2A)。一方面,植物不同生长阶段、器官和组织携带不同内生菌和ARGs,且植物过滤作用使某些ARGs和intI1自下而上传播时相对丰度降低;另一方面,稻田中影响ARGs转移的外部因素比室内盆栽试验更复杂。
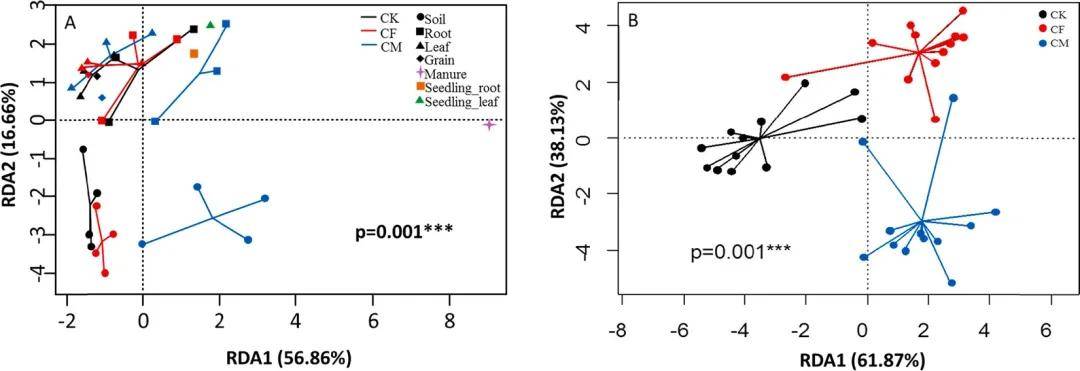
图2. 不同施肥方式及猪粪堆肥处理下水稻植株不同部位和土壤抗生素抗性基因(ARGs)的分布(A)。不同施肥方式下土壤中细菌属水平的分布(B)。CK、CF和CM分别代表未施肥、施用化肥和施用猪粪堆肥的水稻田中的不同处理。
土壤和水稻中抗生素抗性基因(ARGs)的动态
猪粪堆肥对土壤中ARGs的影响
前期土壤微观实验表明,施用猪粪堆肥后第一个月内土壤中ARGs丰度持续下降(见图3),粪肥中携带的ARGs数量减少。本研究中,施用化肥和猪粪堆肥的土壤在第79天ARGs总基因拷贝数显著增加,但总相对丰度未上升,可能是微生物量增加导致ARGs富集。这与长期施用化肥促进ARGs减少的报告不一致,表明长期和短期施用化肥对水稻土中ARGs影响不同。
不同处理的土壤中ARGs的变化
未施用肥料的土壤中,ARGs总基因拷贝数在第一周迅速减少,后缓慢减少至第30天再呈上升趋势至收获期结束(见图3)。表明淹水和作物种植影响内源ARB和ARGs分布,主要是影响微生物群落结构变化,这表明微生物结构在土壤抗生素抗性组中起着重要作用。
施用猪粪堆肥在第0天显著增加土壤中ARGs总基因拷贝数,是施用化肥和对照土壤的1.5倍和3倍(图3),由于施用化肥可能会显著改变微生物群落,因此可以合理地推断,营养可能会影响施用化肥的土壤中ARGs的丰度。
水稻幼苗中ARGs的变化
水稻幼苗地下和地上部分主要携带抵抗β-内酰胺(KPC、cphA)、多重药物(mexF)和磺胺(sul1、sul2)的ARGs(图1A)。随着作物生长,地下部分MLSB抗性基因(pncA)数量增加,尤其是在施用粪肥的地块中(图1A),可能是畜牧业中抗生素迁移所致。基因pncA从土壤转移到水稻根再到叶片。收获时水稻地下或地上部分ARGs总相对丰度和总基因拷贝数相比第0天降低,这表明由于生物量的增加而产生了稀释效应。
推测育苗期间使用猪粪堆肥导致幼苗中ARGs大量积累,水稻幼苗可能是稻田中ARGs主要来源之一,在种植水稻幼苗时控制外源ARGs对减少稻田中ARGs分布至关重要。
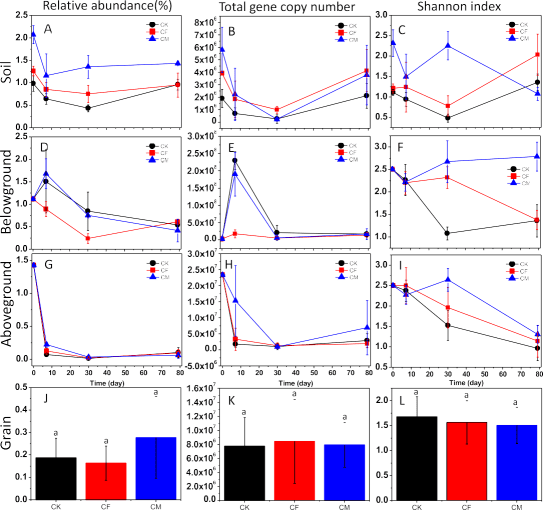
图3. 施用无肥(对照,CK)、化肥(CF)和猪粪堆肥(CM)的土壤中ARGs的总相对丰度(A)、总基因拷贝数(B)和Shannon指数(C)的动态变化;施用无肥(对照,CK)、化肥(CF)和猪粪堆肥(CM)的水稻地下部分中ARGs的总相对丰度(D)、总基因拷贝数(E)和Shannon指数(F)的动态变化;施用无肥(对照,CK)、化肥(CF)和猪粪堆肥(CM)的水稻地上部分中ARGs的总相对丰度(G)、总基因拷贝数(H)和Shannon指数(I)的动态变化;施用无肥(对照,CK)、化肥(CF)和猪粪堆肥(CM)的水稻籽粒中ARGs的总相对丰度(J)、总基因拷贝数(K)和Shannon指数(L)的动态变化。在这三种不同的施肥处理中,水稻籽粒中ARGs的相对丰度、总基因拷贝数和Shannon指数没有显著差异(p > 0.05)。
抗生素抗性基因(ARGs)在土壤-水稻系统中的潜在传播
不同处理下水稻植株和土壤中ARG亚型检测情况
水稻叶片:第79天,CM、CF和CK处理下,从水稻叶片中检测到的ARG亚型数量分别为60、56和54,相同的有50种亚型(见图S2)。施用猪粪堆肥可将新的ARG亚型((如aadE、cphA、erm36、tetW等))引入水稻植株(见图S3)。
水稻根:CM、CF和CK处理下,从水稻根中检测到的ARG亚型数量分别为的68、56和59,相同的有50种亚型(见图S2)。
土壤:CM、CF和CK处理下,土壤中检测到的ARG亚型数量分别为60、32和25,相同的有24种亚型(见图S2)。猪粪堆肥施用对土壤中ARG亚型检测数量的影响比对水稻植株大。
水稻谷粒:CM处理的水稻谷粒中检测到20种ARG,其中13种与CF和CK处理的水稻谷粒相同(见图S2)。TetK、tetM和strB仅在CM水稻谷粒中观察到(见图S3),施用猪粪堆肥对土壤中ARG谱的影响比对水稻植株更大,未显著影响水稻谷粒中的ARG,但丰富了水稻幼苗中的ARG。
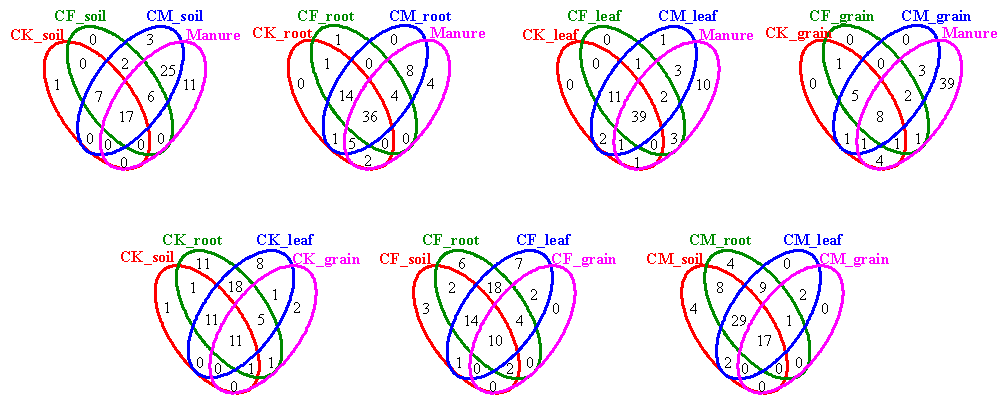
图S2 第79天不同组相同的ARG和MGE数量。CK、CF和CM代表了稻田中不施肥、化肥和猪堆肥的不同处理。根指水稻的地下部分,叶指水稻的地上部分,肥料指这片稻田中使用的猪堆肥。
水平基因转移可能性及ARGs来源
整合酶和转座酶经常被报道为水平基因转移(HGT)的指示基因,研究中几乎所有样本中都发现了intI1和tnpA基因(见图S3),支撑了ARGs转移的可能性。ARGs的来源包括土壤、灌溉水、降水以及当前稻田中使用的水稻秧苗。CM处理因使用猪粪堆肥含有额外的ARGs。CK和CF处理的水稻植株中ARGs和MGEs的检测频率降低,而土壤中的检测频率升高,表明ARGs可能从灌溉水和水稻秧苗转移到稻田土壤中(图1B)。土壤中的固有ARGs可能迁移到水稻植株中。所有三种处理的水稻籽粒中都检测到了ARGs,支撑了ARGs从水稻根和叶转移到水稻籽粒中的可能,其具体来源需进一步探索。大气干沉降和湿沉降对ARGs的影响也需在未来研究中检验。
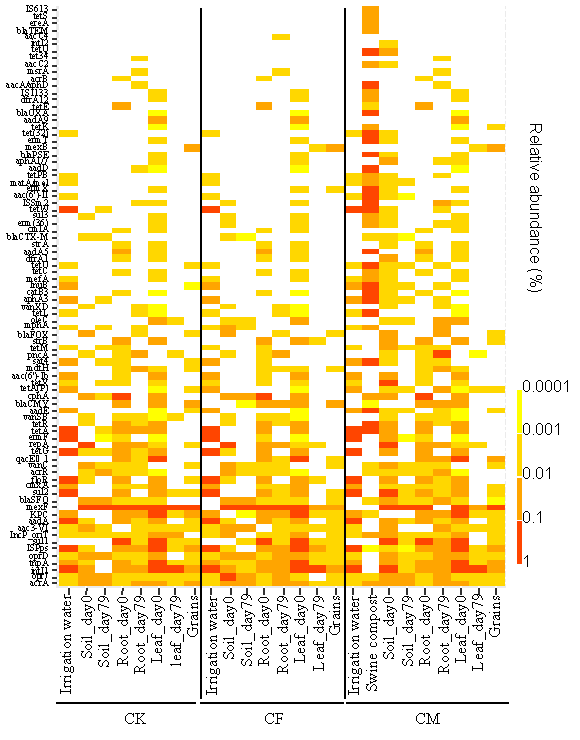
图S3分别在无肥料(CK)、化肥(CF)和猪堆肥(CM)的稻田中,灌溉水、猪堆肥、土壤、水稻和稻谷中ARG和MGE在第0天和第79天的相对丰度热图。
不同处理下水稻地上部分ARGs情况及原因分析
结果:从三个地点收集的水稻地上部分(包括稻谷)中,在不同处理方式下,ARGs的总相对丰度、总基因拷贝数和Shannon指数没有显著差异。这表明施肥对水稻叶片和籽粒中ARGs的分布影响不大。
原因分析:
--淹水条件对土壤中ARGs的消散起到积极作用,减少土壤颗粒在叶面上的潜在附着,抑制ARGs的水平基因转移。
--稻田强降雨对水稻叶片的冲刷和清洁产生强烈影响。
--本研究中使用的水稻秧苗携带大量ARGs并且并未播种,可能掩盖了施肥对水稻移栽后ARGs的影响(见图1A)。
土壤中微生物群落的变化
猪粪堆肥中厚壁菌门是主要微生物门(占比75.72%),其次是放线菌门(占比12.15%)。土壤里变形菌门(占比19.47%-38.09%)、蓝细菌门(占比17.50%-34.86%)和酸杆菌门(占比16.11%-25.03%)是主要微生物门(图4A),施用猪粪堆肥未显著影响水稻土门级微生物结构。属水平上,不同处理间土壤微生物分布差异显著(图2B),水稻生长期间其细菌属级多样性显著降低,施化肥和有机肥的土壤微生物组多样性高于未施肥土壤(图4B)。
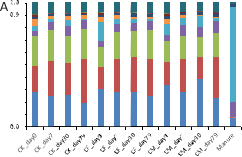
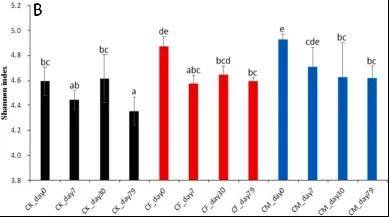
图4. 每个采样点不同施肥方式下土壤中细菌在门水平上的组成,收获时间为第79天(A)。每个采样点不同处理方式下土壤中细菌在属水平上的Shannon指数动态,收获时间为第79天(B)。CK、CF和CM分别代表无肥料、化肥和猪粪堆肥的水稻田中的不同处理方式。
抗生素抗性基因(ARGs)与微生物的共存
对不同处理土壤中的ARGs、MGEs和微生物属进行相关性分析(图5)。对照组有40个节点与47条连接线,CF处理组有66个节点和71条连接线,CM处理组有118个节点和164条连接线。施用化肥和猪粪堆肥显著增加水稻土中核心ARGs与微生物属,猪粪堆肥效果更显著,核心基因和关键细菌因处理而异。在施猪粪堆肥的土壤中,intI1和tnpA为核心基因,与多个细菌属相关,表明ARGs流动性更高。堆肥处理没有显著改变粪便中tetG相对丰度,tetG易成施猪粪堆肥土壤核心基因之一,会随相关细菌属进入土壤。
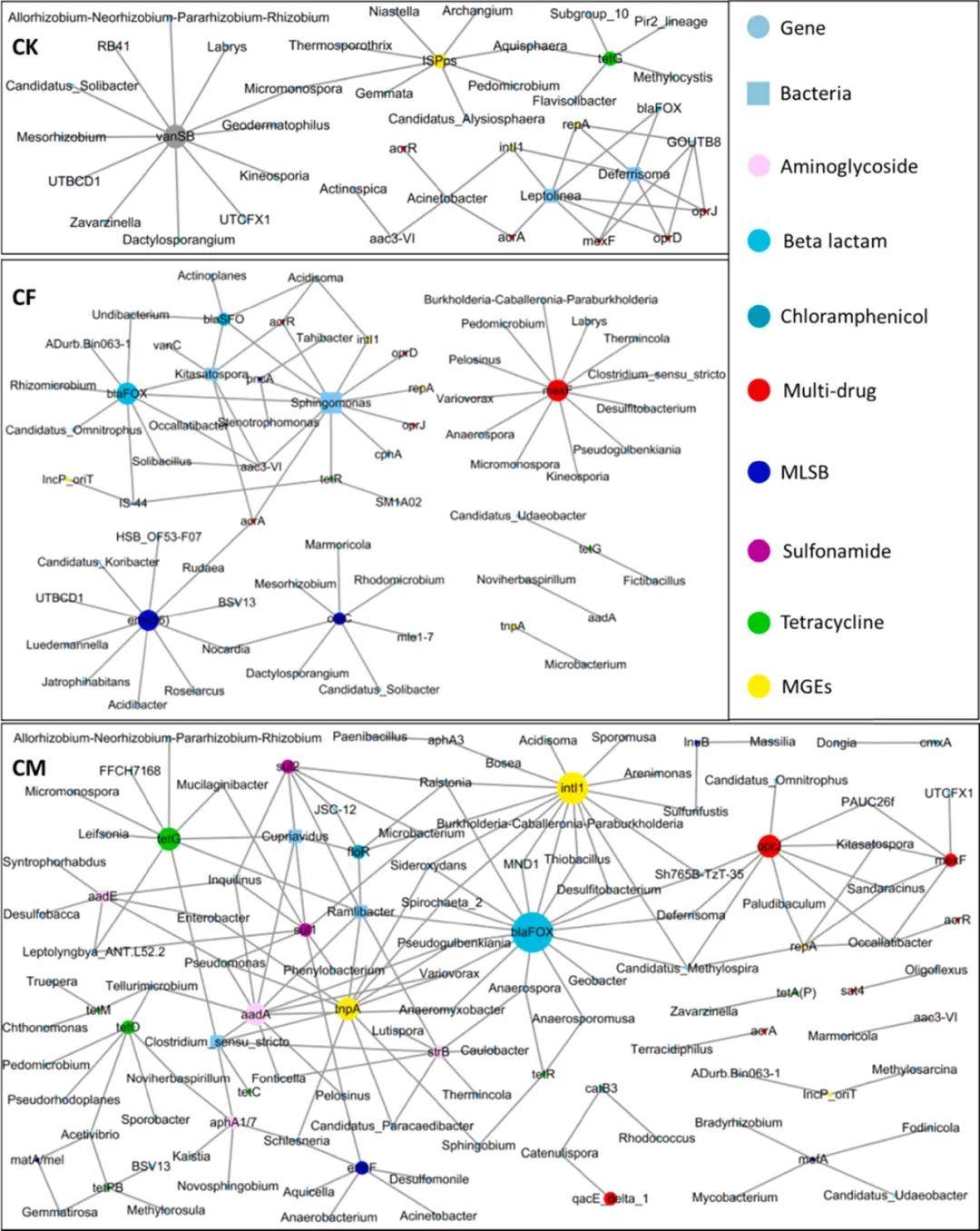
图5. 不同处理下ARGs、MGEs和微生物属级分类群之间的共存网络分析,相关系数(Spearman’s ρ)> 0.7,p < 0.05,N=36,n>=6。方形代表属级细菌。圆形代表ARGs和MGEs。节点颜色代表主要目标抗生素,节点大小与相应基因和微生物属级分类群的频率相关。CK、CF和CM分别代表无肥料、化学肥料和猪粪堆肥的水稻田中的不同处理。
总结
水稻幼苗在分蘖期积累了大量的ARGs,这可能是控制稻田中ARGs积累和扩散的关键步骤。施用粪肥显著增加了土壤和水稻地下部分的ARGs多样性和丰度,但对稻米中的ARGs影响不大。稻米中ARGs的来源仍需进一步探索。目前的研究发现,粪肥携带的ARGs从土壤迁移到水稻植株,以及ARGs从水稻幼苗到稻米和土壤的存在潜在双向转移。这些发现为评估稻田中ARGs的风险提供了基础信息





















