
2025-01-06

前言
基因组测序(Genome Sequencing):是在全基因组层面对DNA进行测序的一项关键技术,能够全面揭示生物体的遗传信息。通过这一技术,能够深入了解个体基因组的构成、变异及遗传特征。基因组测序不仅推动了生命科学领域的基础研究,还为医学、农业等多个领域带来了革命性的变化。随着技术的不断进步和研究的深入,基因组学研究的应用前景也更加广阔。
回望过去一年,基因组项目研究的浪潮汹涌澎湃。在这承上启下的时刻,小派特地为大家呈上基因组项目文章的精彩盘点。接下来,小派将从动植物基因组和微生物基因组两大类,精选2024年的一些标杆性文章,带大家领略基因组项目文章的精妙思路,快来与小派一同重温这些科研盛宴吧!
一、 动植物基因组
1.中国樱桃的逆袭之路! De novo+比较基因组+转录组深入解析助力高分密钥

发表期刊:Horticulture Research
测序平台:Illumina、PacBio、Hi-C、ONT
分析内容:单倍型基因组组装、比较基因组分析、TEM、转录组测序、亚细胞定位、转基因等
结果展示:

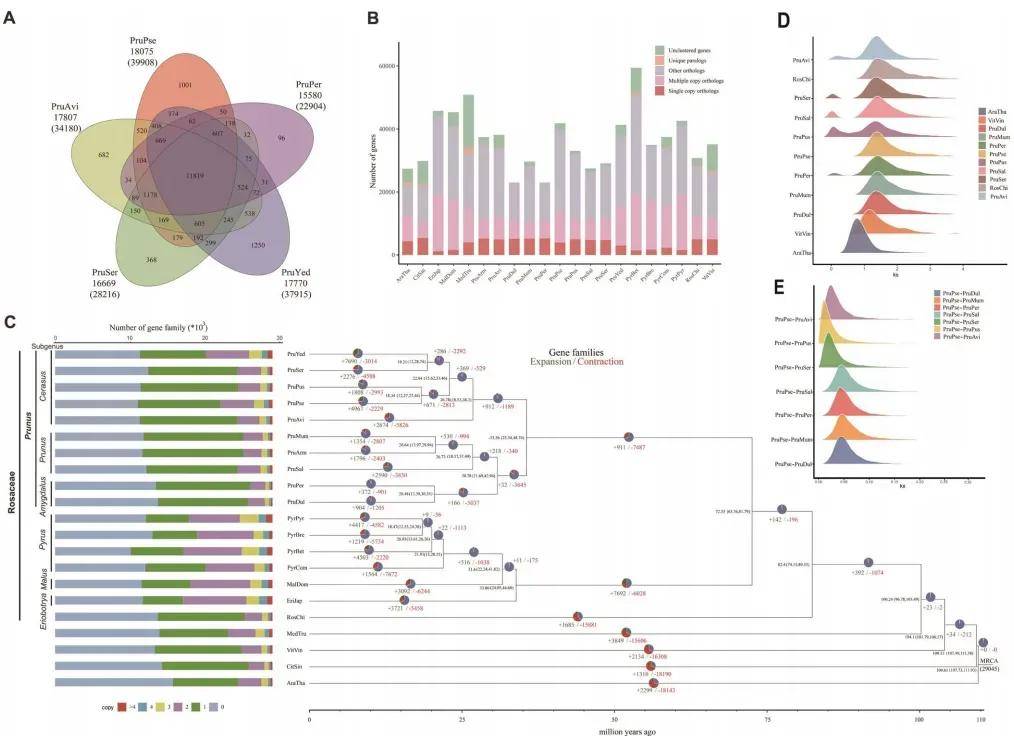
中国樱桃,又称酸樱桃或山樱桃,是一种重要的果树作物,广泛种植于中国及亚洲其他地区。本研究利用单体型基因组组装技术,成功组装了高质量的四倍体中国樱桃基因组。鉴定了多个与果实硬度相关的基因,特别是细胞壁修饰酶基因。通过表达谱分析,揭示了这些基因在果实发育过程中的动态变化。这些成果不仅为理解果实硬度的遗传机制提供了重要线索,还为未来的樱桃育种和改良工作带来了巨大的潜力。具体应用前景包括:分子标记辅助育种、基因编辑技术、精准农业。
2.BSR测序解锁黄油生菜理想株型的秘密

发表期刊:Horticulture Research
测序平台:Illumina
分析内容:BSR测序分析、CRISPR/Cas9敲除、转基因互补、RNA-seq、电镜、RT-PCR等
结果展示:
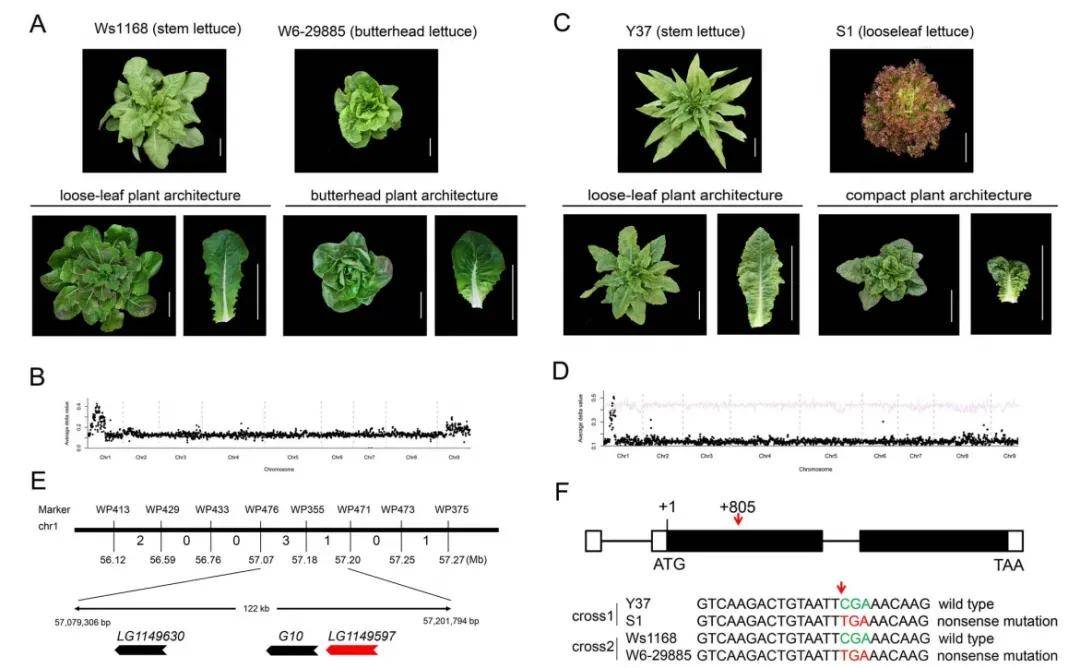
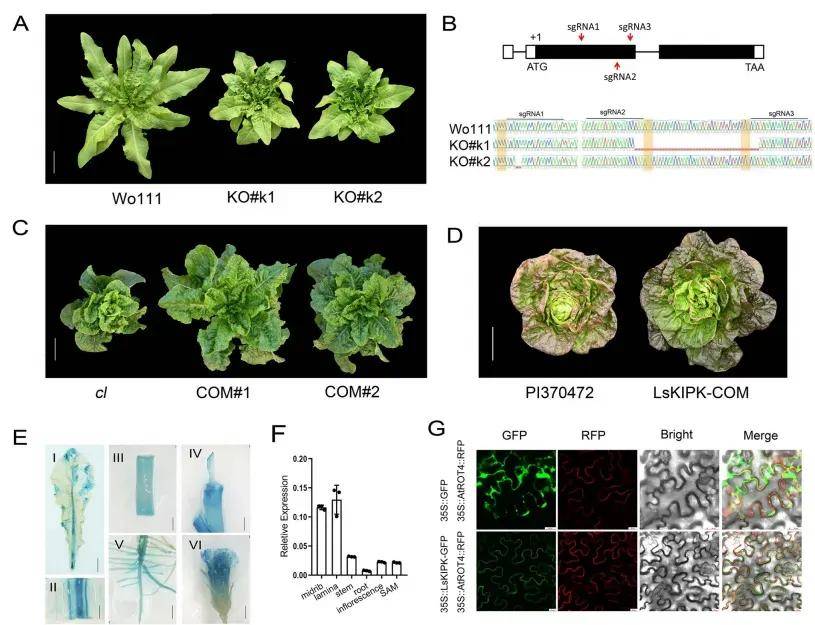
本研究将一个黄油生菜品种(同样携带Lskipk突变)与一个结球生菜品种杂交,并进行了BSR遗传定位分析,鉴定出一个新的编码ATP酶的基因,该基因同样对黄油生菜的株型有所贡献。进一步通过敲除与互补实验验证了LsATPase功能的缺失也是形成黄油生菜株型所必需的。本研究结果不仅增进了对控制生菜等蔬菜作物特定性状遗传基础的理解,也为实现作物品种的精准设计和高效培育奠定了理论与实践基础,有望在未来的农业可持续发展中发挥重要作用。
3.基因组+转录组测序揭示藏羊高原适应的遗传多样性及机制

发表期刊:Journal of Animal Science and Biotechnology
测序平台:Illumina
分析内容:全基因组测序、群体遗传结构分析、遗传多样性分析、选择性清除分析、转录组测序分析等
结果展示:
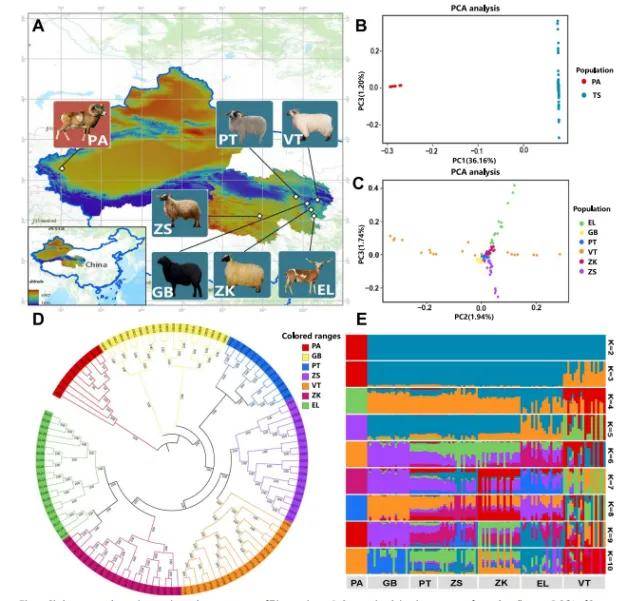
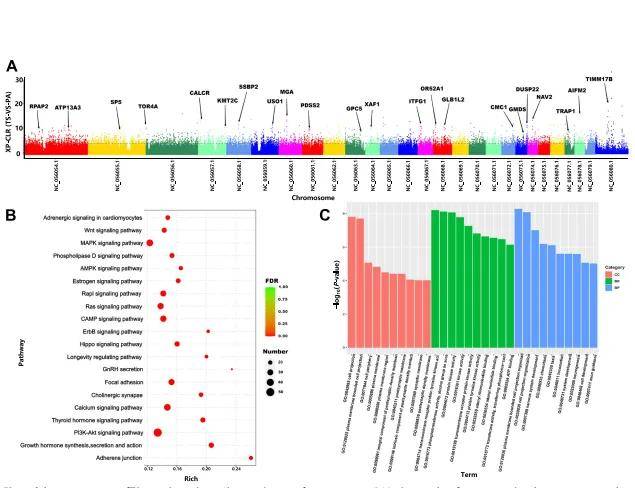
本研究对6个藏羊品种进行了全基因组重测序,利用其遗传结构和群体多样性进行分析。结果显示,6个藏羊品种在系统树上表现出明显的分离;但各品种间差异较小,存在广泛的基因流现象。聚类分析将这6个品种分为3种不同的生态类型:高原型、谷地型和欧陆型。阿尔泰野山羊与藏羊的特有单核苷酸多态性(SNP)及选择性扫荡分析表明,藏羊驯化主要与感官信号传导、营养吸收代谢以及生长繁殖特性相关。最后,综合分析选择性扫荡和转录组数据发现,在青藏高原不同海拔生活的藏羊通过增强心肺功能、调节体液平衡并通过肾回吸收来适应环境,并且通过改变消化道中营养物质的消化和吸收途径以提高耐受力。该研究为极端环境条件下藏羊进化过程提供了重要启示,并将有助于藏羊遗传多样性的保护以及青藏高原动物环境适应机制的研究。
4.群体遗传学分析揭示了玉米胚性愈伤组织诱导能力的关键遗传变异和驱动因素

发表期刊:Journal of Integrative Agriculture
测序平台:Illumina
分析内容:全基因组测序、遗传多样性分析、群体遗传结构分析、选择性清除分析等
结果展示:
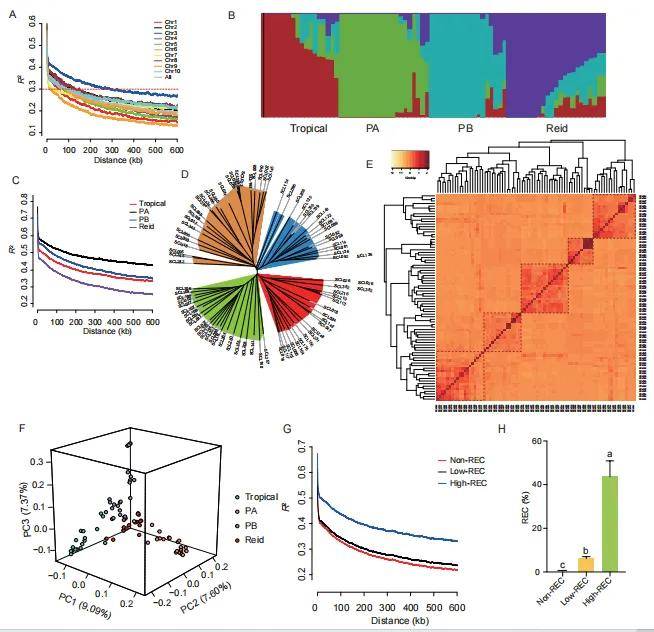
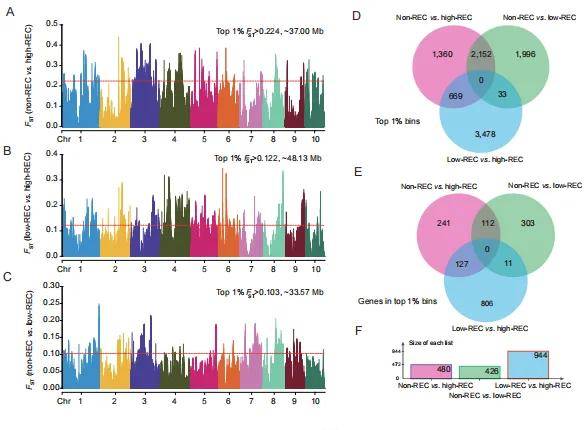
本研究对80份核心种质进行了全基因组测序,并利用我们开发的新一代高密度变异图谱构建管道(MQ2Gpipe),建立了基于诱导率(REC)的分类体系。低REC种质具有更丰富的遗传多样性;通过整合全基因组选择性标记筛选和区域关联分析,揭示了95.23Mb的选择性区域以及43个REC相关位点。这些位点的表型方差解释值在21.46%到49.46%之间。总共鉴定出103个候选基因位于这些REC相关位点的连锁不平衡区域内。这些基因主要参与细胞周期调控、细胞分裂调控和其他功能,在其中MYB15和EMB2745位于先前报道过的EC诱导QTL内。许多与叶面积相关的大型效应变异紧密地连接到多个REC相关位点上,暗示现代玉米育种中可能存在着REC和叶片大小协同选择的现象。这项研究不仅增加了我们对玉米胚胎培养诱导能力遗传基础的理解,也为培育具有高REC能力的玉米品种提供了理论和实践指导。
二、 微生物基因组
1、细菌基因组
(1)细菌基因组完成图+比较基因组助力ST1049-KL5肺炎克雷伯菌的双重耐药机制探索

发表期刊:communications biology
测序平台:Illumina+Pacbio
分析内容:细菌基因组完成图测序、比较基因组圈图、easyfig遗传环境分析、cgMLST分析、质粒复制子分析、系统进化树构建等
结果展示:
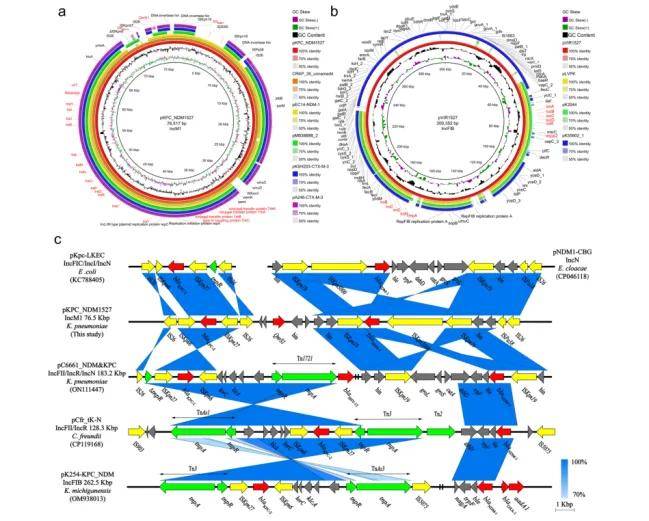
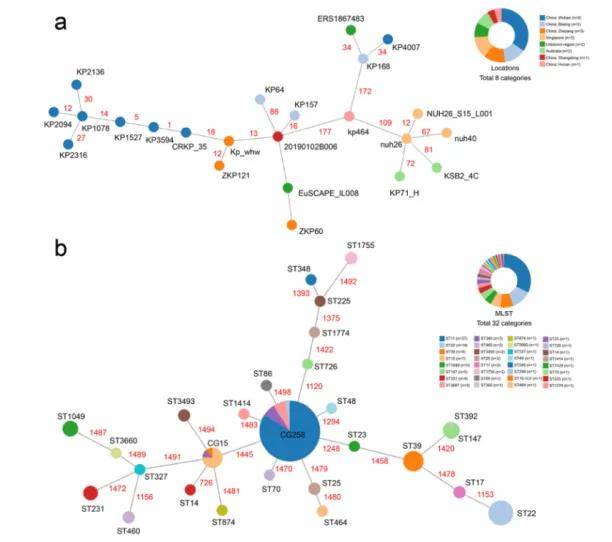
本研究在4个ST1049-KL5肺炎克雷伯菌分离株中鉴定了一个结合的IncM1质粒pKPC_NDM,它共同携带blaKPC-2和blaNDM-1基因。该质粒表现出较高的种内和种间可转移性,并增强了碳青霉烯类耐药性,ST1049-KL5型CRKP的出现及克隆传播对公共卫生构成了严峻挑战。本研究凸显了ST1049型菌株作为高毒性及多重耐药质粒携带者的潜力,强调了持续监测和实施有效感染控制策略的重要性。这些发现不仅增进对复杂耐药机制和病原体传播动态的理解,而且为未来针对性的治疗干预和防控措施提供了科学依据。
(2)细菌完成图+转录组+代谢组学揭秘高效菲降解菌的超能力

发表期刊:Journal of Hazardous Materials
测序平台:Illumina+PacBio
分析内容:细菌完成图、降解效率分析、HPLC、LC-MS、GC-MS、基因表达分析等
结果展示:
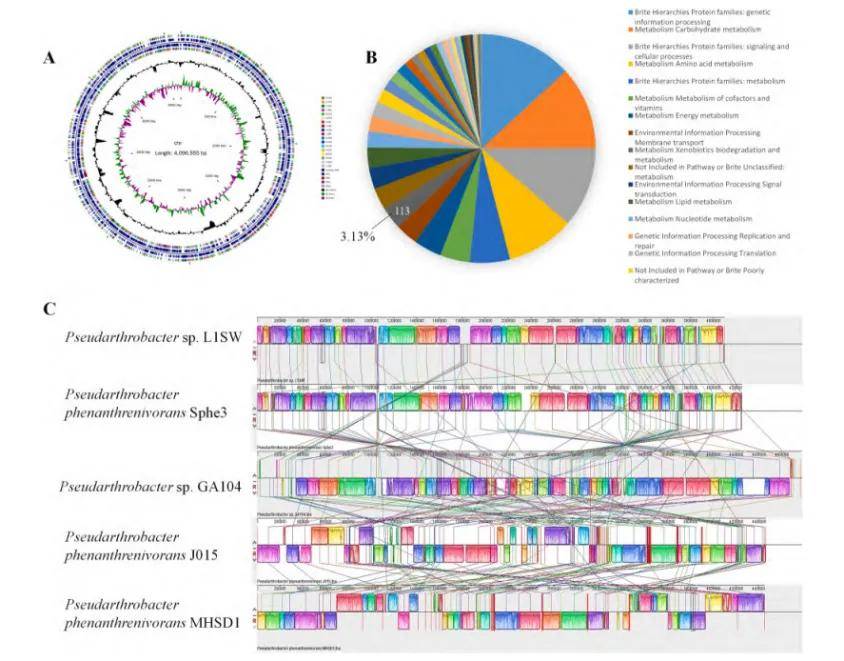
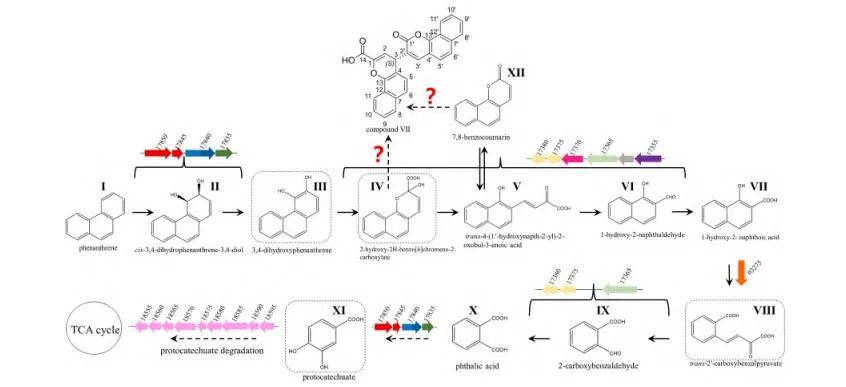
本研究中,分离并鉴定了一株具有高效降解菲能力的菌株,即Pseudarthrobacter sp. L1SW。该菌株展现出在多种环境条件下稳定降解菲的能力,包括不同的盐度和碱度、重金属暴露以及表面活性剂存在的情况。它主要通过邻苯二甲酸途径降解菲,并形成了一个新颖的中间代谢产物。在L1SW中鉴定了三个涉及菲降解的基因簇,并据此提出了菲的代谢途径。此项工作加深了我们对革兰氏阳性菌及植物生长促进根际细菌(PGPR)中菲降解机制的理解,为利用微生物进行多环芳烃(PAHs)生物修复提供了更多潜在菌株资源。
(3)肥胖儿童中培养的肠球菌B6通过生物活性代谢物酪胺促进非酒精性脂肪肝病的发展

发表期刊:Gut Microbes
测序平台:Illumina+Nanopore
分析内容:细菌基因组完成图、16S多样性分析、代谢组和转录组测序分析、小鼠实验等
分析结果:
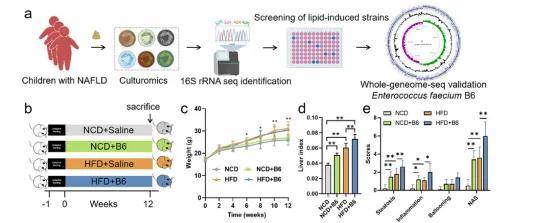
本研究鉴定了Enterococcus faecium B6作为一种潜在的新型致病菌株,它可能促进儿童非酒精性脂肪肝病(NAFLD)的发展。E. faecium B6衍生的生物活性代谢物——酪胺,可能对肝脏脂肪变性、炎症和纤维化进展产生影响,这些因素促进了NAFLD的发展。这些结果表明E. faecium B6和酪胺与NAFLD之间存在因果关联。这项研究强调了宿主-微生物的相互作用,并为NAFLD或相关代谢性疾病的肠道微生物群管理提供了新的见解。
(4)通过碳离子束辐照诱导的高产纳豆激酶活性突变体——枯草芽孢杆菌的表型和基因组洞察

发表期刊:International Journal of Biological Macromolecules
测序平台:Illumina
分析内容:细菌基因组完成图、重测序等
结果展示:
在本研究中,我们首次使用12C6+重离子束诱变技术获得了一种高产NK活性的突变菌株(枯草芽孢杆菌ZT-S1)。重离子束还改变了枯草芽孢杆菌的表面形态,这是由于功能团的变化所致。此外,枯草芽孢杆菌ZT-S1需要更多的碳和氮源,并且达到稳定期的时间更晚。比较基因组分析揭示,大多数与突变相关的基因(oppA、appA、kinA、spoIIP)与孢子形成有关。受影响的rpoA基因与编码NK的基因aprE的合成有关。此外,通过诱变获得的枯草芽孢杆菌ZT-S1具有良好的遗传稳定性。本研究进一步探讨了影响NK活性的因素,并为商业应用中NK的生产提供了有前景的微生物资源。
2、真菌基因组
(1)多组学分析揭示野生型酿酒酵母WY319高效苯乙醇转化机制

发表期刊:Biotechnology Journal
测序平台:Illumina+Nanopore
分析内容:真菌重测序、转录组测序、比较基因组分析等
结果展示:
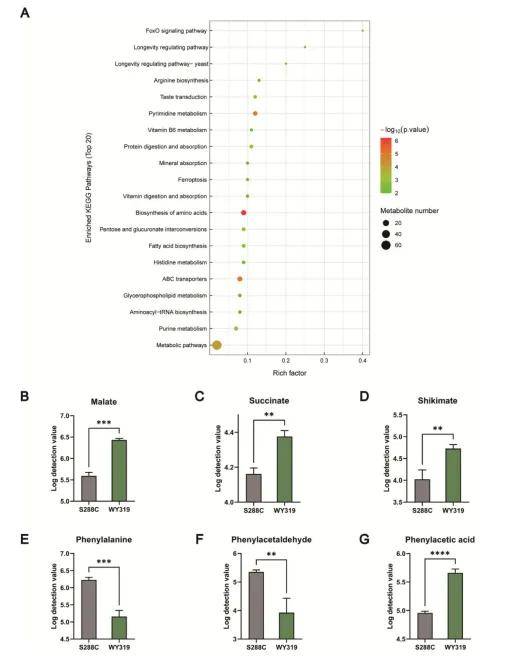
本研究对高、低产2-PE菌株进行了系统性的代谢特征比较分析,结果表明高产菌株可能得益于呼吸代谢增强及耐受性提高而具有更高的产量潜力;同时,通过对关键生长速率下的转录组进行分析发现,线粒体功能基因簇的整体上调在2-PE的合成过程中发挥着更为突出的作用。此外,无靶向代谢组学的结果显示,在提高呼吸代谢的同时降低Crabtree效应,可促进内源氨基酸和嘌呤等抗逆物质的积累,从而为2-PE的合成提供更丰富的前体和辅因子来源。综上所述,该策略为后续针对相关合成途径的强化提供了研究基础,并且也为S. cerevisiae非酒精产品的合成提供了思路。
(2)IF=8.4!探索耐药真菌的进化之路:FKS1突变在棘白菌素耐药耳念珠菌中的进化积累

发表期刊:Emerging Microbes & Infections
测序平台:Illumina
分析内容:药敏试验、真菌重测序、系统发育分析、蛋白-蛋白互作网络分析等
结果展示:
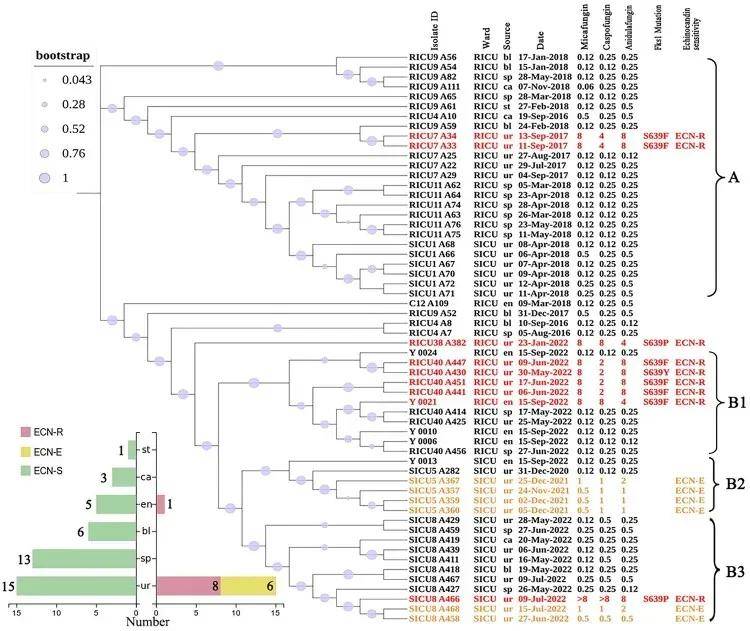
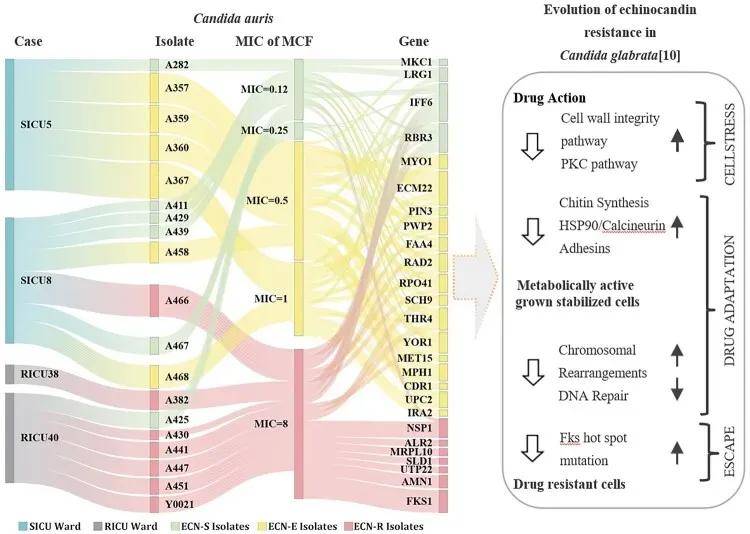
本研究收集并分析了4例临床病例和29株与棘白菌素耐药性逐步发展相关的分离株,通过抗真菌药物敏感性测试和基因组测序来评估棘白菌素耐药性的演变。结果发现从接受棘白菌素治疗的患者的泌尿系统中分离出6株棘白菌素最低抑菌浓度(MIC)升高的耳念珠菌菌株和7株耐药菌株。同时,系统发育分析表明,这些棘白菌素耐药菌株与同一患者中的其他菌株密切相关。基因组数据显示,这些棘白菌素耐药菌株具有FKS1突变。此外,耳念珠菌中的三类非同义突变SNP基因(如RBR3、IFF6、MKC1、MPH1、RAD2和MYO1)似乎与光滑念珠菌棘白菌素耐药性的三阶段进化模型相关:细胞壁应激、药物适应和遗传逃逸(FKS突变)。本研究通过分析耳念珠菌分离株的全基因组微进化特征,获得对棘白菌素耐药性机制的深入理解。
3、小基因组
(1)多功能药用植物金盏花的叶绿体基因组分析与进化研究

发表期刊:scientific report
测序平台:Illumina
分析内容:叶绿体基因组denovo测序、系统发育分析、分化时间分析、IR边界区分析等
结果展示:
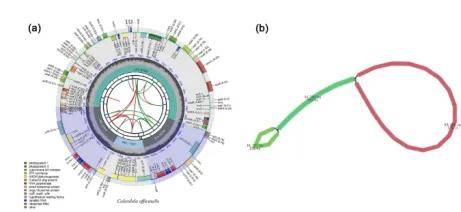
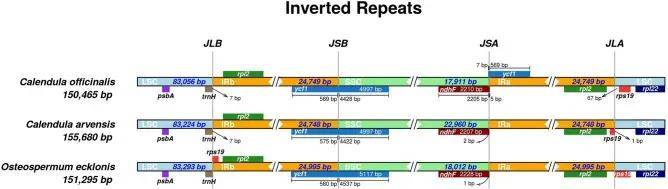
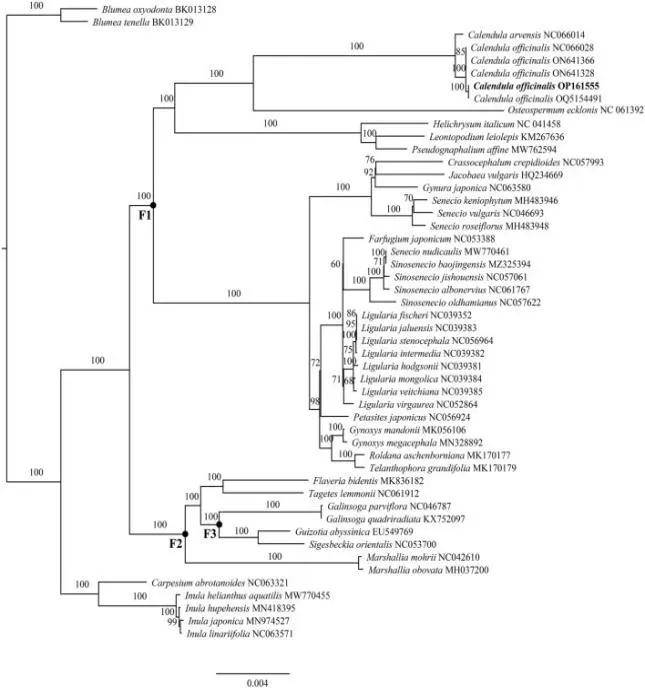
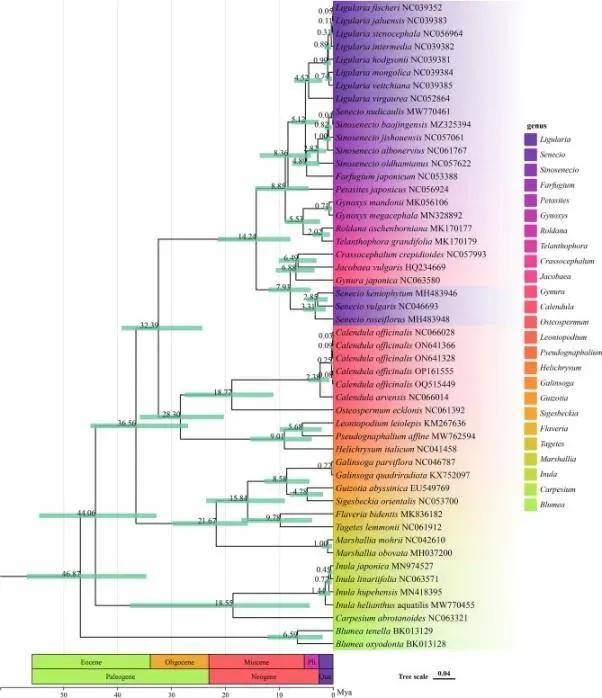
本研究对金盏花的叶绿体基因组、系统发育关系、密码子偏好性和分化时间进行全面分析,结果表明,这些金盏花、欧洲金盏花和南非万寿菊之间存在密切的进化关系,并且它们在共同环境选择压力的作用下发生了相似的基因表达模式和遗传变异。此外,该研究还发现了某些特定基因的进化特点,这可能与金盏花的药用价值有关。本研究不仅提高了对金盏花基因组的认识,还为探索其遗传多样性和挖掘其巨大的药用潜力奠定了基础。
(2)线粒体基因组比较基因组分析揭秘瘿螨高级分类群的演化关系

发表期刊:BMC Biology
测序平台:Illumina NovaSeq
分析内容:线粒体基因组测序、系统发育分析、分化时间估算、基因排列等
结果展示:
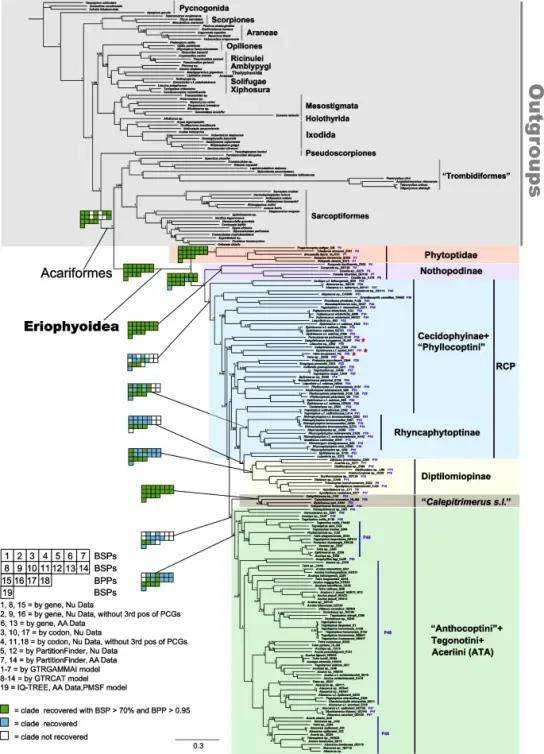
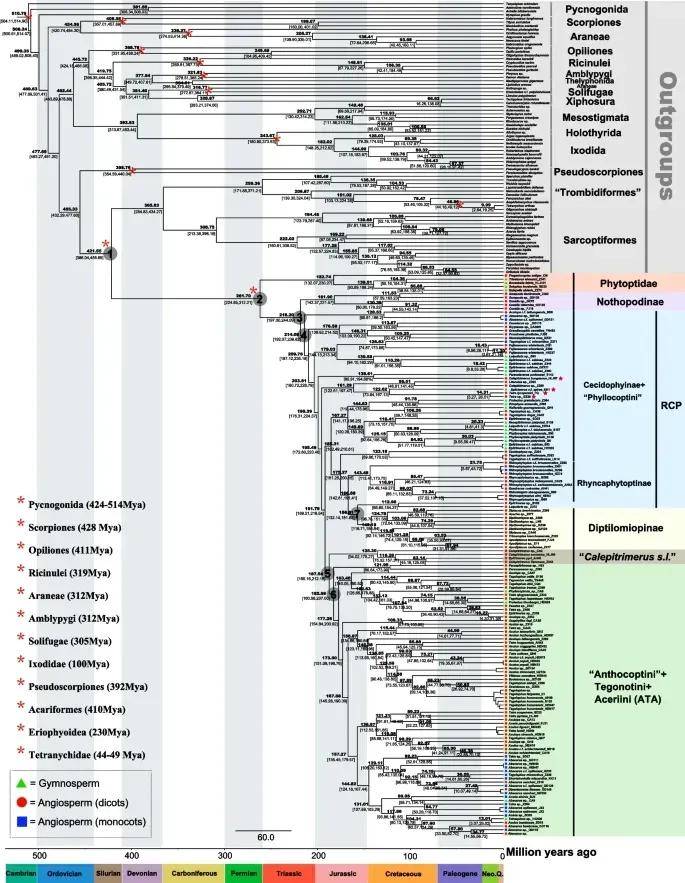
本研究对153种瘿螨的线粒体基因组进行了测序和比较,研究结果首次证明线粒体基因组变化的基因可以解决瘿螨总科内部高级阶元的分类系统。同时发现蛛形纲中多样化的线粒体基因排列模式与各个类群的进化速率呈正相关。本研究进一步推测瘿螨总科起源于三叠纪,多样性形成于白垩纪,与被子植物多样性的形成相吻合。该研究为利用线粒体共享基因簇解决存在争议的节肢动物分类系统提供了直接证据,为理解多样化的线粒体排列模式的诱因提供了崭新的思路。
(3)短尾噬菌体A1432的基因组分析揭示Mesyanzhinovviridae新属

发表期刊:Frontiers in Microbiology
测序平台:Illumina NovaSeq
分析内容:噬菌体基因组测序、系统发育分析、比较基因组分析等
结果展示:
本研究对一株感染S. maltophilia YCR3A-1的溶原性噬菌体A1432进行了表征。透射电镜显示噬菌体A1432具有二十面体头和较短的尾部结构。BLASTn分析显示,噬菌体A1432基因组与NCBI数据库中的Xanthomonas phage Xoo-sp2共享最高相似度(81.46%),但覆盖率为37%。该噬菌体含有双链DNA,基因组长度为61,660 bp,GC含量为61.92%,预测包含79个开放阅读框和一个tRNA,没有毒力或抗药性基因。系统发育分析表明,噬菌体A1432属于Bradleyvirinae亚家族,但其进化分支与其他成员明显不同。进一步使用平均核苷酸同源性、蛋白质谱系统发育分析以及基因组网络分析比较了已知分离的S. maltophilia噬菌体,结果表明这些噬菌体之间存在显著的遗传多样性。本研究将有助于进一步了解它们的形态学和遗传多样性的研究,并阐明导致它们进化的演化机制。
文章索引:
1.Jiu, ST, et al. Haplotype-resolved genome assembly for tetraploid Chinese cherry (Prunus pseudocerasus) offers insights into fruit firmness.Hortic Res.2024 Jul 8;11(7):uhae142.
2.Xie, Sai, et al. Lskipk Lsatpase double mutants are necessary and sufficient for the compact plant architecture of butterhead lettuce.Horticulture Research, 2024, 11: uhad280.
3.Li, X., Han, B., Liu, D. et al. Whole-genome resequencing to investigate the genetic diversity and mechanisms of plateau adaptation in Tibetan sheep. J Animal Sci Biotechnol ,2024, 15: 164 .
4.Liu, P, et al. Population genomic analysis reveals key genetic variations and the driving force for embryonic callus induction capability in maize, Journal of Integrative Agriculture, 2024, 23(7): 2178-2195.
5.Liu, Hongmao, et al. Prevalence of ST1049-KL5 carbapenem-resistant Klebsiella pneumoniae with a bla KPC-2 and bla NDM-1 co-carrying hypertransmissible IncM1 plasmid. Communications Biology 7.1 (2024): 695.
6.Li, JL, et al. Identification of an efficient phenanthrene-degrading Pseudarthrobacter sp. L1SW and characterization of its metabolites and catabolic pathway .J Hazard Mater.2024 Mar 5:465:133138.
7.Wei J, Luo J, Yang F, et al. Cultivated Enterococcus faecium B6 from children with obesity promotes nonalcoholic fatty liver disease by the bioactive metabolite tyramine. Gut Microbes. 2024, 16(1):2351620.
8.Sheng Y, Zhang S, Li X, et al. Phenotypic and genomic insights into mutant with high nattokinase-producing activity induced by carbon ion beam irradiation of Bacillus subtilis. Int J Biol Macromol. 2024, 271:132398.
9.Yang, C.H. et al., Unveiling the mechanism of efficient β-phenylethyl alcohol conversion in wild-type Saccharomyces cerevisiae WY319 through multi-omics analysis. Biotechnology Journal, 2024.
10.Tian S, Wu Y, Li H, Rong C, Wu N, Chu Y, Jiang N, Zhang J, Shang H. Evolutionary accumulation of FKS1 mutations from clinical echinocandin-resistant Candida auris. Emerg Microbes Infect. 2024 Dec;13(1):2377584.
11.Zhang N, Huang K, Xie P, Deng A, Tang X, Jiang M, Mo P, Yin H, Huang R, Liang J, He F, Liu Y, Hu H, Wang Y. Chloroplast genome analysis and evolutionary insights in the versatile medicinal plant Calendula officinalis L. Sci Rep. 2024 Apr 26;14(1):9662.
12.Zhang Q, Lu YW, Liu XY, Li Y, Gao WN, Sun JT, Hong XY, Shao R, Xue XF. Phylogenomics resolves the higher-level phylogeny of herbivorous eriophyoid mites (Acariformes: Eriophyoidea). BMC Biol. 2024 Mar 22;22(1):70.
13.Li S, et al., Characterization and genomic analysis of a lytic Stenotrophomonas maltophilia short-tailed phage A1432 revealed a new genus of the family Mesyanzhinovviridae. Front. Microbiol, 2024, 15.