2025-01-10
1、背 景
非负矩阵分解(NMF)是一种分析高维数据的方法,它通过将高维的基因表达矩阵分解为低维的特征矩阵和系数矩阵,提取出隐藏在数据中的主要特征和模式。目前常用于单细胞数据中识别基因表达的潜在模式和多个样本中稳健的基因程序(MP),尤其适合分析异质性极高的的肿瘤细胞中的基因程序。软件由瑞士洛桑大学的Santiago J. Carmona团队开发,目前发布在https://github.com/carmonalab/GeneNMF。
2、分析方法与流程
GeneNMF 分析主要包括三个步骤:
A.不同样本的非负矩阵分解
B.基因模块聚类和分群
C.确定基因模块功能
流程示例如下:
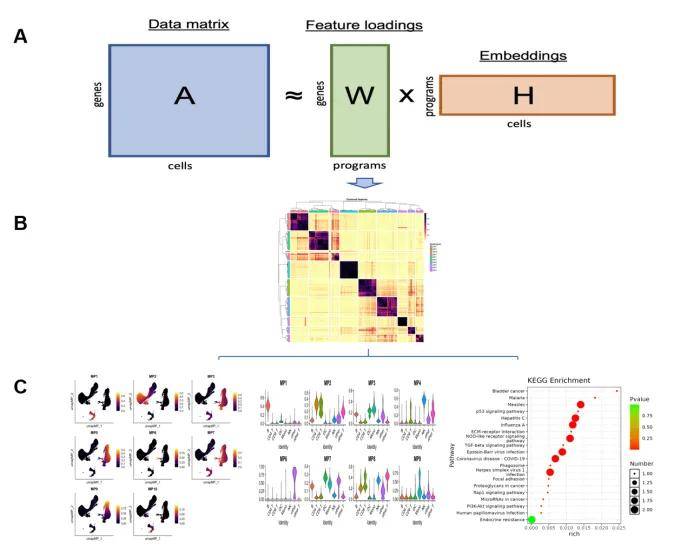 图1 GeneNMF 分析流程
图1 GeneNMF 分析流程
在实际项目分析中,我们会根据单细胞scRNA分析得到的RDS文件,提取过滤后的data信息来进行每个分组(推荐sample)的NMF分析;然后根据每个基因模块之间的成对相似性对基因程序聚类和可视化;最后对每个基因程序的基因进行富集分析和Ucell打分,评估不同基因程序的生物学意义。
3、结果示例
3.1基因程序聚类热图
软件首先会根据数据分组信息,对每个样本在多个K值(分解基数量,也可以是预期细胞类型数量等)水平上分别进行NMF分析,以保证识别到基因模块的稳健性。然后根据基因模块间的余弦相似性聚类得到基因程序,这里可以自定义聚类结果的程序数。
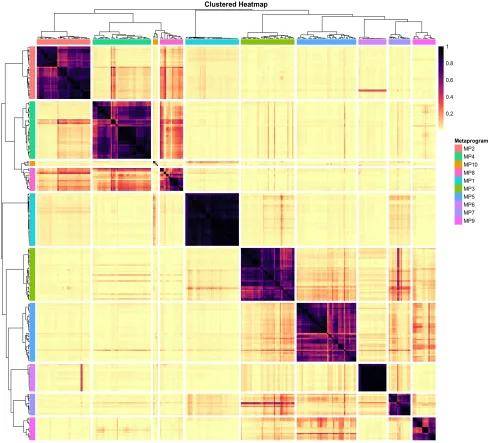 图2 基因程序聚类热图
图2 基因程序聚类热图
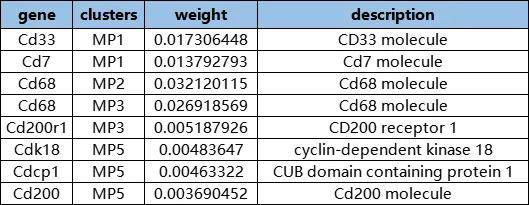
我们同时也会输出每个基因程序的基因和权重的gene2weight_anno.xls表格,方便初步确定基因程序基因的功能。
表1 基因程序注释表格
3.2基因程序的特征打分
得到基因程序后,我们可以将每个基因程序作为一个基因集进行Ucell打分,查看不同分组下某些基因程序是否在单个样本中富集,或者不同的基因程序在某些分组中有较高得分。结合不同样本或细胞类型的临床数据,可以探究基因程序与不同性状间的关联。
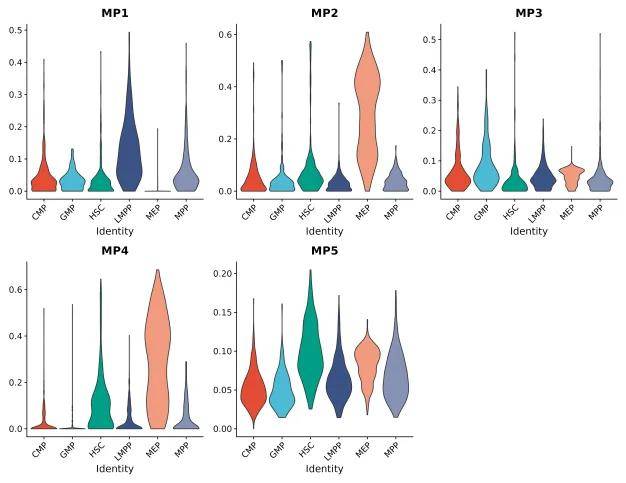
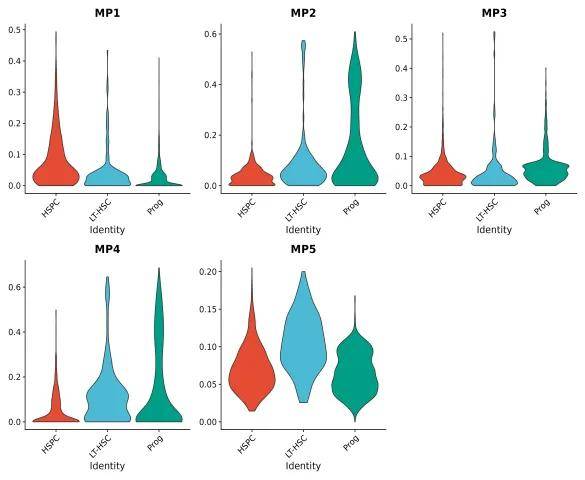 图3 基因程序不同分组小提琴图
图3 基因程序不同分组小提琴图
3.3打分聚类图展示
为了方便查看不同分组的打分情况,软件会根据Ucell打分进行降维聚类。因为这里的基因程序是在多个样本中发现的,因此重新降维聚类可能是减轻样本间批次效应的一种有效方法。展示结果如下图所示,通过对比不同分组的打分高低,进一步确认不同类型的细胞和基因程序的关联。

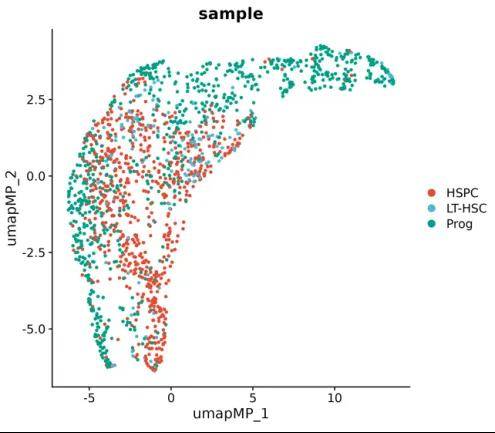 图5 Ucell打分Feature图
图5 Ucell打分Feature图
3.4基因程序的富集分析
最后,我们会对每个基因程序的基因与GO/KEGG数据库进行富集分析,以确定不同基因程序参与和影响哪些生物学过程。这里也可以选择物种的marker基因集,辅助判定不同的细胞类型。
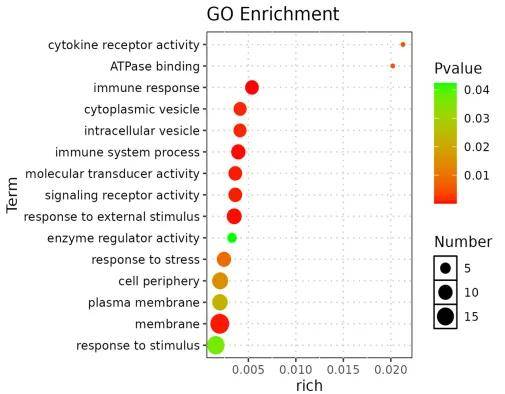
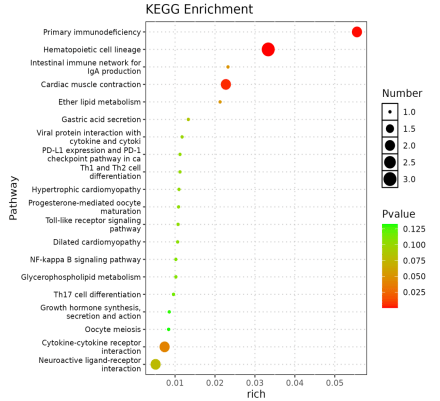 图4 每个基因程序的基因富集分析
图4 每个基因程序的基因富集分析
通过GeneNMF分析,我们可以更好地理解细胞的异质性和功能多样性,目前该算法已经广泛应用于单细胞的细胞亚群识别、基因模块发现以及多样本数据整合等分析。
参考文献
[1]https://www.biorxiv.org/content/10.1101/2024.05.31.596823v1
[2]Barkley, D., Moncada, R., Pour, M.et al.Cancer cell states recur across tumor types and form specific interactions with the tumor microenvironment.Nat Genet 54, 1192–1201(2022).
[3]Gavish, A., Tyler, M., Greenwald, A.C.et al.Hallmarks of transcriptional intratumour heterogeneity across a thousand tumours.Nature 618, 598–606 (2023)


