2025-01-13

拿到转录组的数据,辛辛苦苦数据挖掘后,该如何获取目的基因的CDS信息去做qPCR验证呢?今天小派就以项目为例,给大家介绍如何快速获取参考基因组CDS信息。
章节目录
1、转录组项目参考基因组信息获取
2、NCBI来源基因组CDS信息获取
3、ensembl来源基因组CDS信息获取方式
4、近期转录组项目CDS信息获取
一.转录组项目参考基因组信息获取
在接收到我们的项目分析结果后,根据不同项目类型,分析结果分别在线上(云平台)和线下进行释放。云平台和线下(网页版报告或PDF版report)获取基因组信息如下:
云平台参考基因组信息获取:
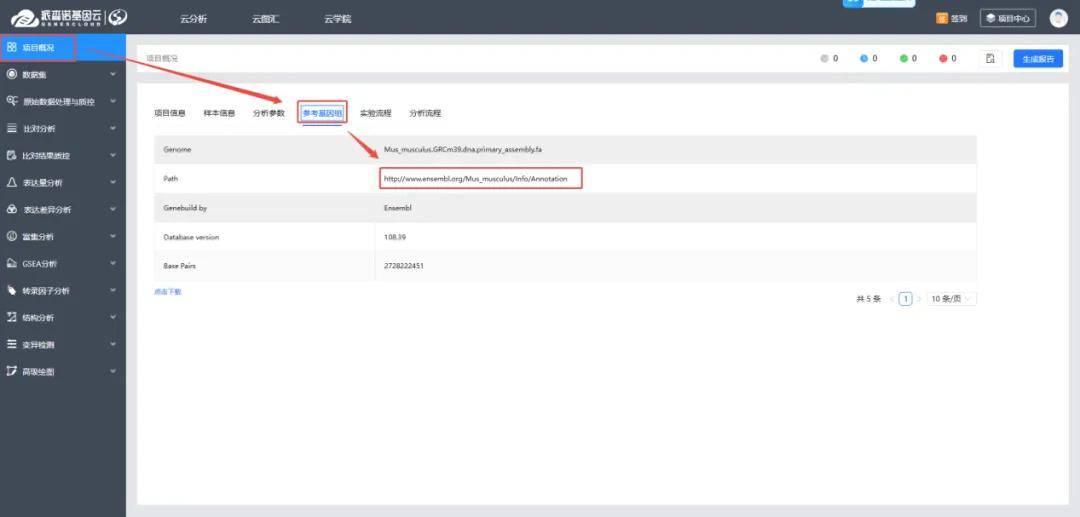
线下报告参考基因组信息获取:

二.NCBI来源基因组CDS信息获取方式
1.在获取参考基因组信息后,可以直接复制该网址在浏览器中打开。
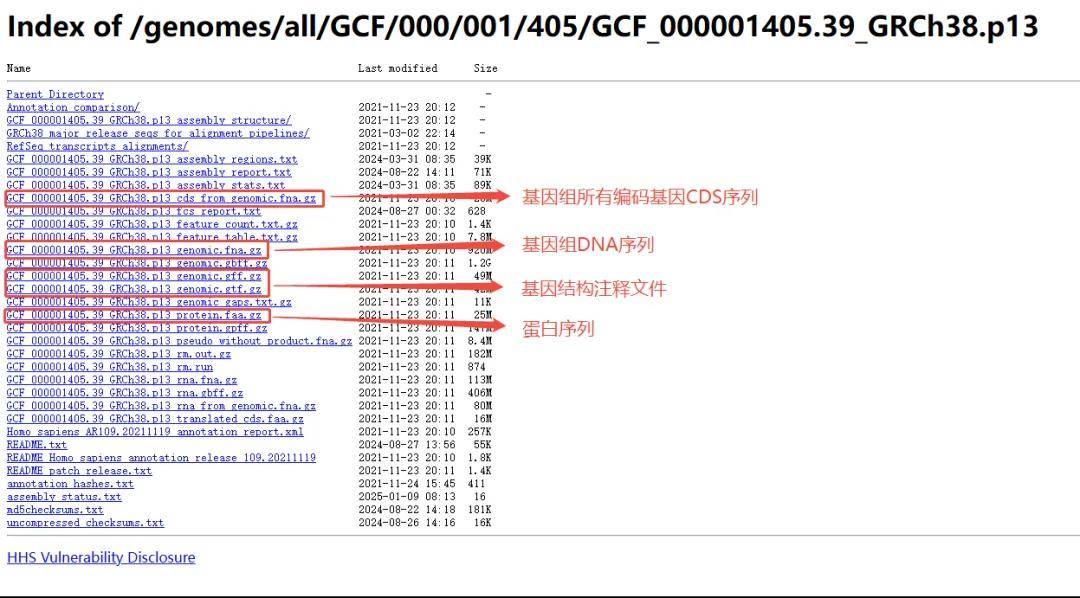
2.例如NCBI来源的基因组打开对应链接后直接点击下载“cds from genomic.fna”。
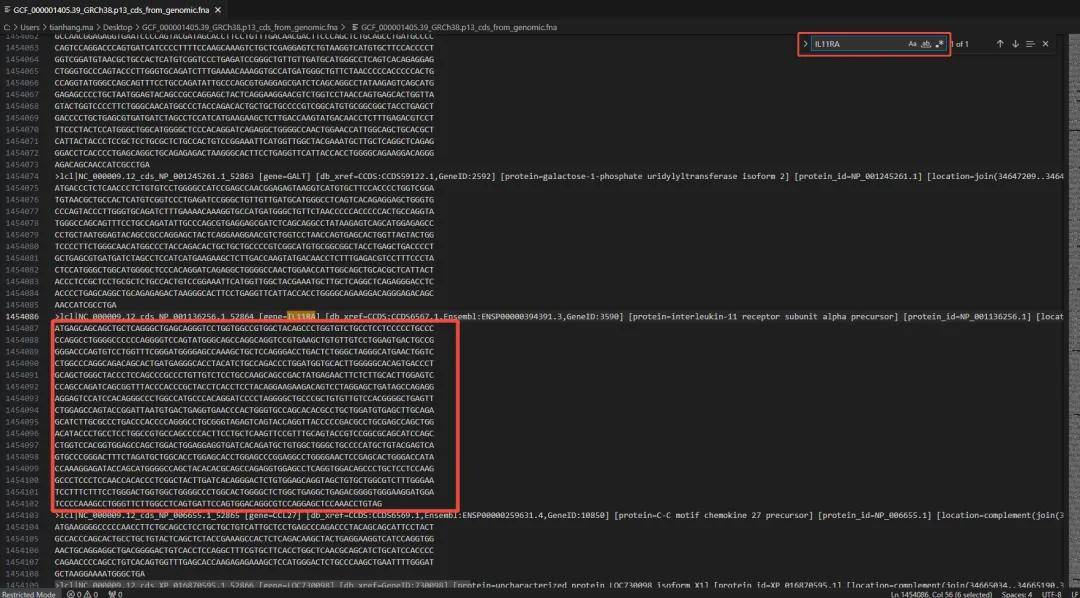
3.解压后,用Visual Studio Code或是 Editseq等软件打开序列文件(实在没有对应软件,浏览器也可以打开),根据gene ID进行检索,即可获取基因对应的CDS信息。
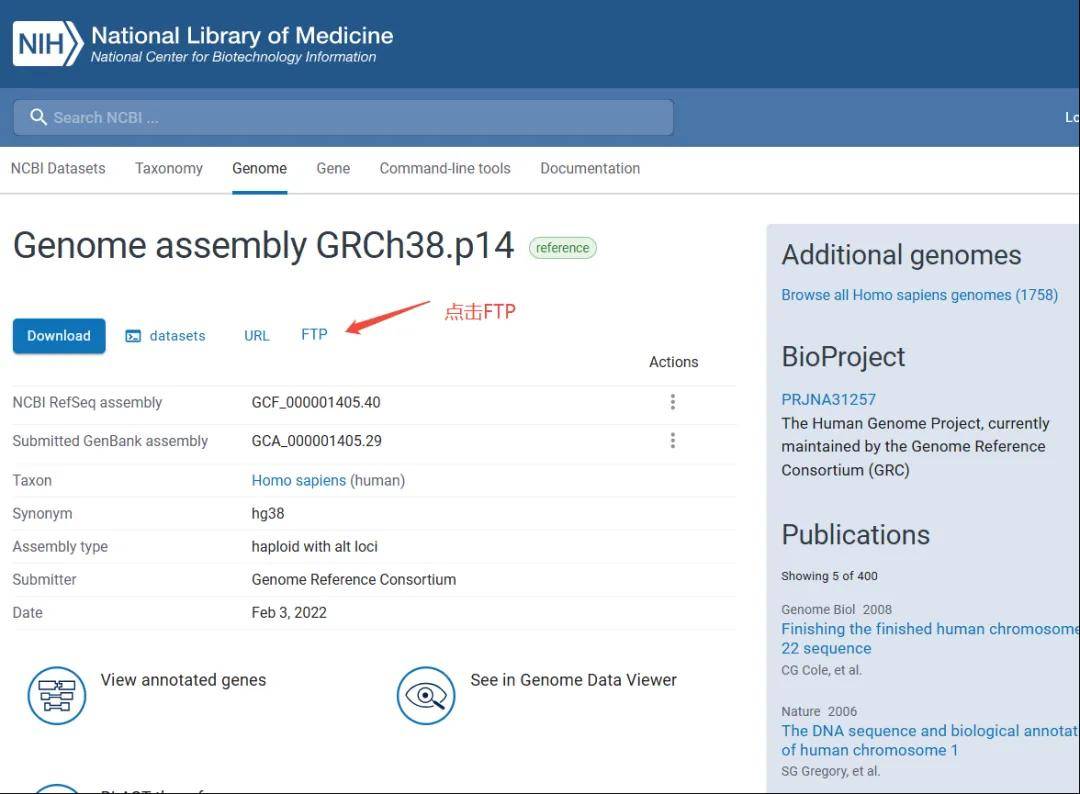
4.此外如果基因组链接不是链接上图页面,也可以在NCBI直接检索GCF号/GCA号后,点击FTP进入页面。

三.Ensembl来源基因组CDS信息获取方式
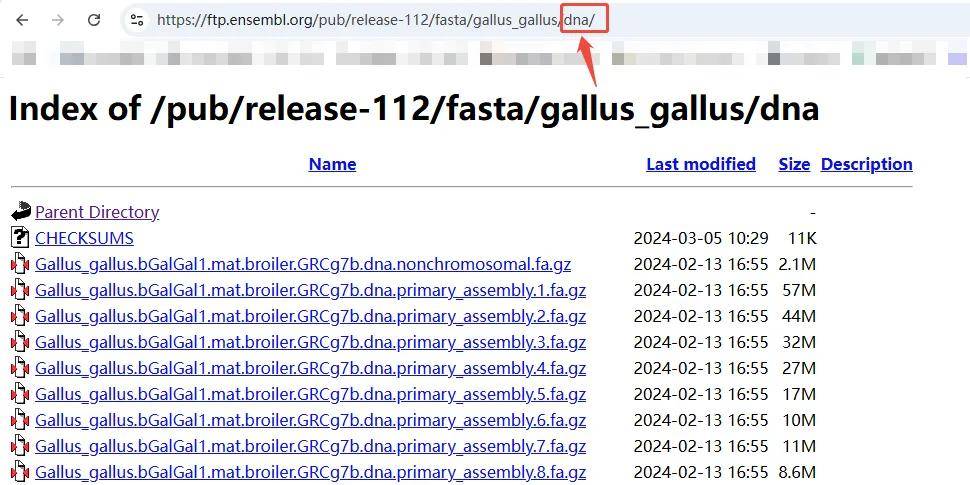
1.参考基因组是ensembl数据库来源,我们同样只需复制该网址在浏览器中打开。
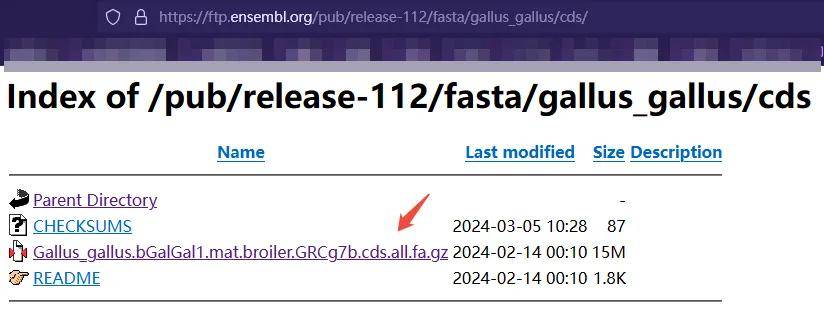
2.打开链接后删除dna返回上一级,点击进入cds文件夹下载CDS序列即可。
3.后续同上述NCBI来源基因组一样,打开序列文件后,根据gene ID检索基因获取信息。

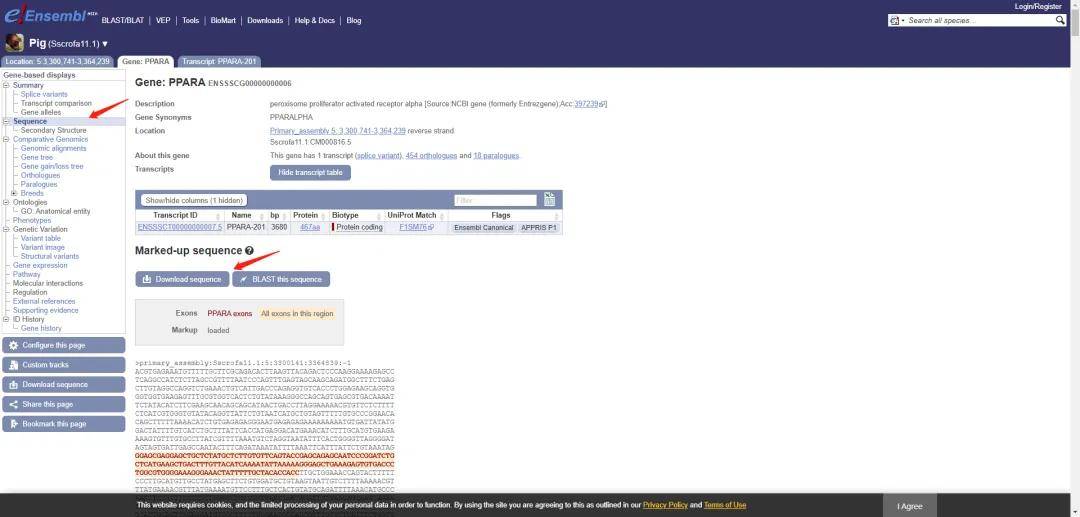
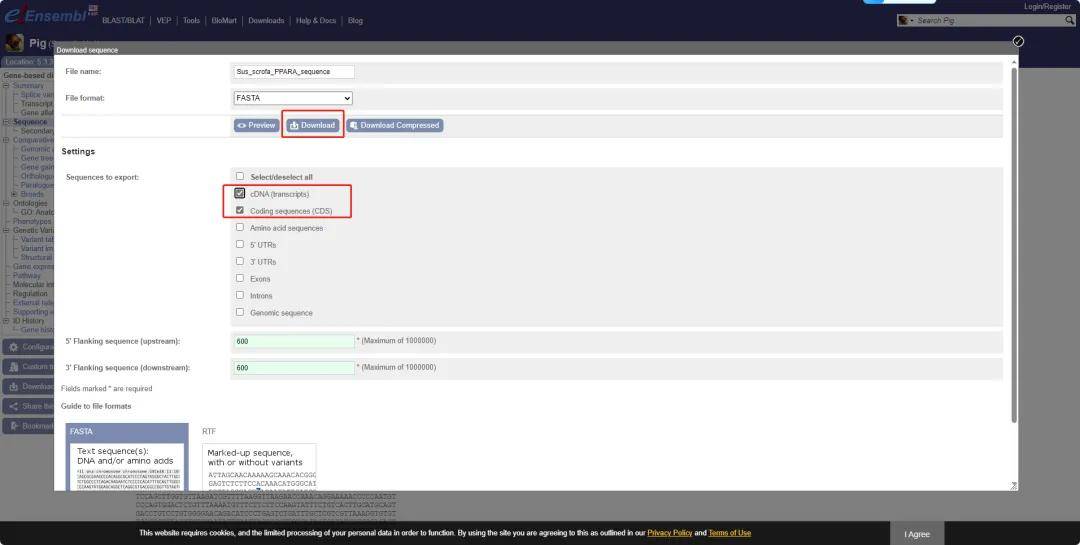
此外,ensembl数据库可以直接根据gene ID检索获取CDS信息,如图所示,可以直接在ensembl网址检索框检索gene ID(注意gene ID信息是唯一的,不建议用gene name检索),然后点击Sequence后,接着选择Download sequence,除了可以下载CDS外,转录本cDNA,3’UTR,5’UTR等信息也是可供下载的。
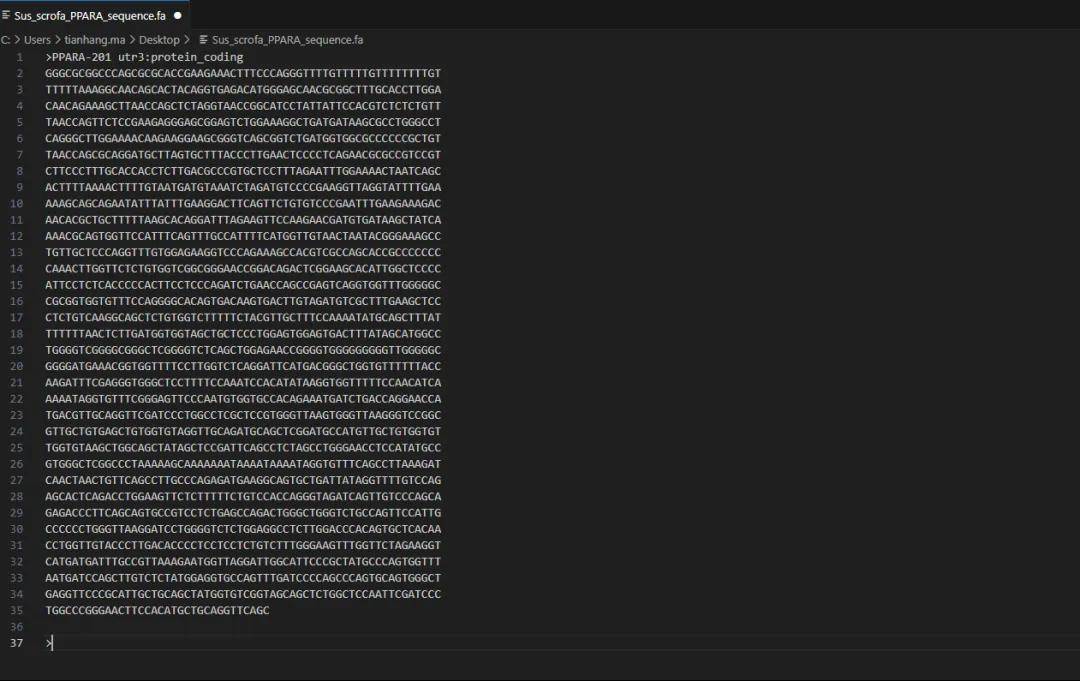
四.近期转录组项目快速获取CDS
12月交付的转录组项目中,线下文件除了交付Rawdata,Map文件夹外等信息,我们也直接整理了基因组CDS信息放在了annotation文件夹下(原核转录组我们额外提供了基因组序列信息fasta以及基因结构注释信息gff)。

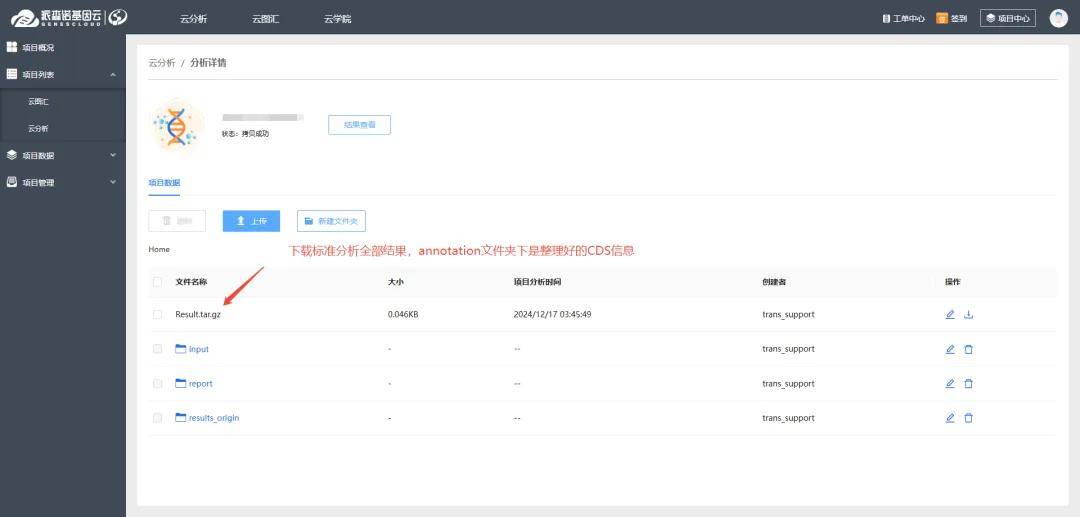
此外咱们云平台近期项目在云平台也可以直接查看CDS啦,老师们只需在云平台下载“Result.tar.gz”,解压后里面的annotaion文件夹下是整理好的CDS,妈妈再也不用担心我找不到了。
最后,如果大家对qPCR该如何挑选基因有选择困难症,可以看看我们往期“RNA-seq与qPCR,你不得不知道的那些事!!!”(点击查看)


