
2025-01-20

单细胞多组学技术在近年来快速发展,已成为揭示复杂生物学过程的重要工具。过去的研究中,主要采用了单细胞RNA测序(scRNA-seq)来分析单细胞层面的基因表达。然而,这类方法无法同时揭示基因组染色质结构与RNA异构体之间的复杂关系。RNA异构体的多样性决定了基因的可变功能,进而影响细胞的表型复杂性。与此同时,表观遗传修饰显著增强了细胞间的异质性,并且近期研究表明,表观遗传修饰在选择性剪接(AS)事件中发挥着重要作用。然而,目前仍缺乏有效的技术手段,能够在单细胞层面同时分析基因组RNA异构体与表观遗传状态。
2024年9月,中山大学胡友金教授团队在Nature Communications杂志在线发表题为“Simultaneous profiling of RNA isoforms and chromatin accessibility of single cells of human retinal organoids”研究论文。因此作者在他们之前开发的方法scRCAT-seq基础上和单细胞ATAC-seq整合进一步开发了可同时分析同一单细胞内的染色质可及性和全长RNA异构体的方法scRICA-seq。作者利用scRICA-seq绘制了人类视网膜类器官的多组学景观,揭示了染色质可及性和RNA剪接对介导人类视网膜类器官内神经元发育和再生的关键命运决定基因的一致调控。
PART1、技术原理
scRICA-seq技术基于从同一单细胞中并行捕获DNA和RNA,旨在同时分析同一细胞内的染色质可及性、RNA表达及RNA异构体。为实现DNA和RNA的并行捕获与测序,作者采用了两种策略:对于低通量单细胞样本,研究团队利用之前开发的核-细胞质分离方法,从同一细胞中获取细胞核和细胞质RNA;而对于高通量单细胞群体,研究团队则采用微流控系统,可以同时分析单个细胞核中的染色质可及性与转录组(图1a)。
为了对全长RNA异构体的单细胞转录组进行分析,作者进一步优化了先前的方法,开发了新型的scRCAT-seq2技术。图1b展示了scRCAT-seq2的具体流程,主要包括以下步骤:(1)使用唯一分子标识符(UMI)标记单个RNA/cDNA分子的末端;(2)通过端到端连接构建环状cDNA;(3)进行滚环扩增,以生成多个全长cDNA的拷贝;(4)使用Tn5转座酶在环状全长cDNA上随机插入,将其片段化;(5)构建文库并对片段进行双端测序;(6)整合具有相同UMI的短读段,将这些短读段映射到特定的异构体特征,包括转录起始位点(TSS)、转录终止位点(TES)以及特定外显子。最后,通过UMI标记将每个短读段分配到特定的RNA异构体,并利用测序数据对异构体进行重构。
 图1 scRICA-seq技术的工作流程及其应用示意图
图1 scRICA-seq技术的工作流程及其应用示意图
PART2、主要结果
1、scRCAT-seq2在实验中的表现
scRCAT-seq2的短读长可以有效覆盖全长转录本,包括TSS、TES和外显子(图2a)。作者加入了包含92个ERCC和69个异构体的RNA标准集(SIRV-set 3)作为真实值对照用以评估scRCAT-seq2在识别和量化单细胞内异构体方面的灵敏度和准确性,scRCAT-seq2可以识别61个SIRV转录本,灵敏度为88%,全长特征揭示准确率为100%(图2b,c)。此外,作者观察到单个细胞之间ERCC表达的高度相关性,以及观察到的丰度与预期值的高度一致性(图2d,e)。我们还发现单个细胞之间的基因和异构体存在很强的相关性(图 2f),表明 scRCAT-seq2 数据具有很高的一致性。
作者将scRCAT-seq2的性能与其他能够在单细胞水平异构体检测方法进行了比较,包括基于PacBio长读长测序的ScISOr-seq和HITscISO-seq、基于Nanopore长读长测序的SCAN-seq2和scCOLOR-seq,以及基于短读长测序的scRCAT-seq。观察到,在相同成本的不同测序深度中,scRCAT-seq2在检测单细胞异构体方面具有最高的灵敏度和效率(图2g)。
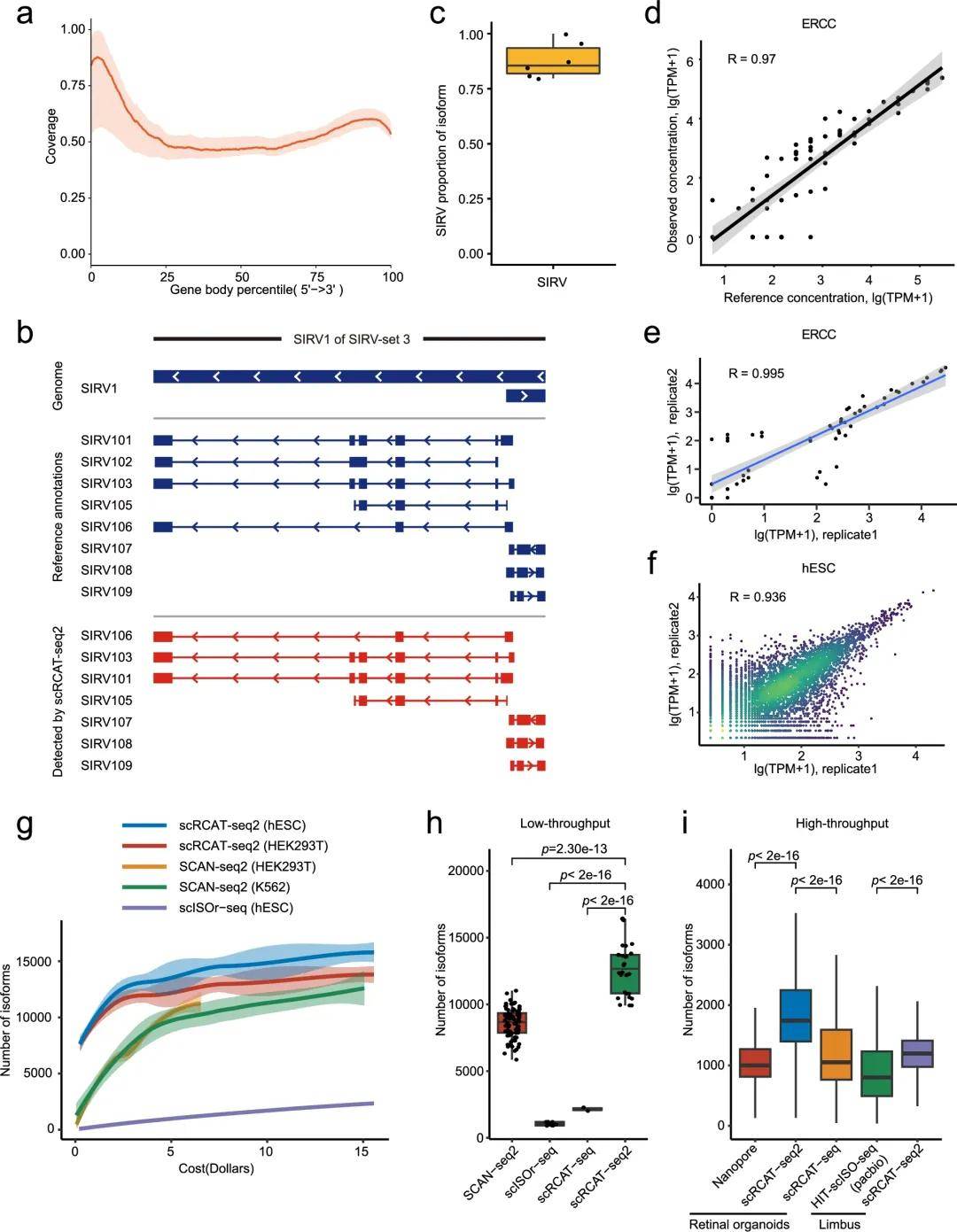 图2 scRCAT-seq2的性能表现
图2 scRCAT-seq2的性能表现
2、构建基于scRICA-seq数据的人类视网膜类器官的多组学单细胞图谱
研究者将scRICA-seq应用于人类视网膜发育过程,构建了人类视网膜类器官的单细胞多组学图谱。基于单细胞异构体的聚类分析,确定了六个细胞簇,对应于六种典型的视网膜早期神经元,包括视网膜祖细胞(RPCs)、视网膜神经节细胞(RGCs)、无长突细胞/水平细胞(AC/HCs)、视锥细胞前体(PR precursors)、神经生成RPCs(neurogenic RPCs)以及视锥细胞(cones),其结果与scRNA-seq技术所得数据高度一致。
 图3 基于scRICA-seq数据的人类视网膜类器官的多组学单细胞图谱
图3 基于scRICA-seq数据的人类视网膜类器官的多组学单细胞图谱
3、染色质可及性与视网膜祖细胞(RPC)分化为视锥细胞过程中RNA转录之间的关系
图4a通过伪时间分析展示了RPC向视锥细胞发育的轨迹,并在图4b中揭示了这一过程中基因表达与染色质可及性的显著变化。图4c至4f则展示了不同基因和异构体在RPC分化为视锥细胞过程中的动态变化。图4g至4i进一步揭示了关键基因(如NRL、CRX和SOX2)在这一过程中染色质开放性与基因表达水平之间的相关性。这些结果清晰地表明,染色质可及性在视网膜细胞命运决定过程中发挥了关键作用。
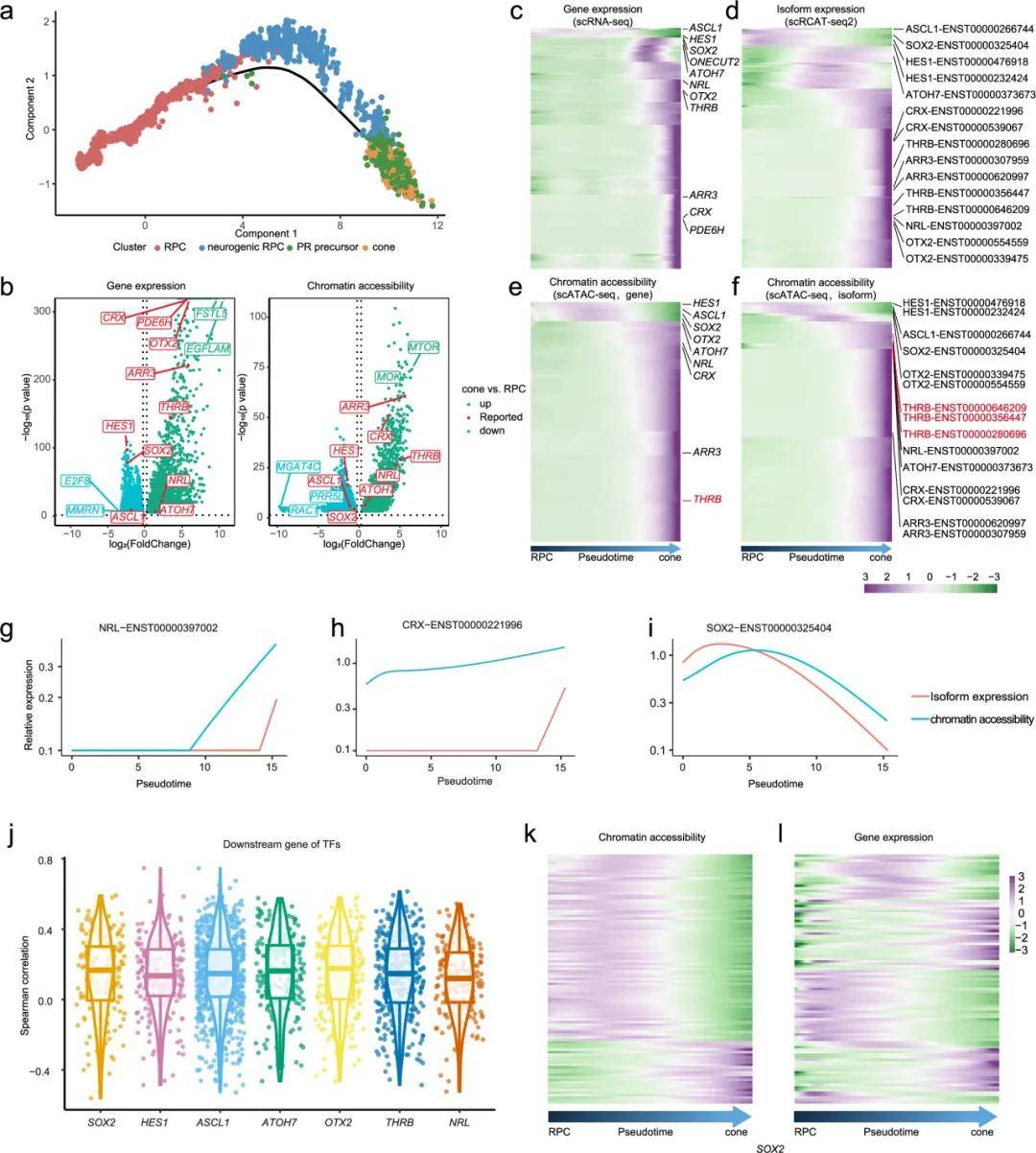 图4 染色质可及性与RPC命运决定因子的RNA转录相关
图4 染色质可及性与RPC命运决定因子的RNA转录相关
4、RNA异构体选择与染色质可及性及转录因子(TF)之间的关系
进一步,作者研究了RPC分化为cone过程中RNA isoform的动态选择模式,发现cone和RPC之间共有2292种差异表达的isoform,其中1143种isoform在cone中高度表达,1149种在RPC中高度表达。通过转录因子motif分析,研究者预测了589个可能与这些动态调节的剪接位点结合的转录因子。在预测的转录因子中包含了11种先前报道的参与调节RNA剪接的转录因子,包括NEUROD1、SP1和KLF4等,同时还包含了已知的细胞命运决定因子,如NEUROD1、ASCL1和THRB等。这提示,剪接位点染色质可及性的动态变化与RNA剪接可能协同调控细胞命运决定
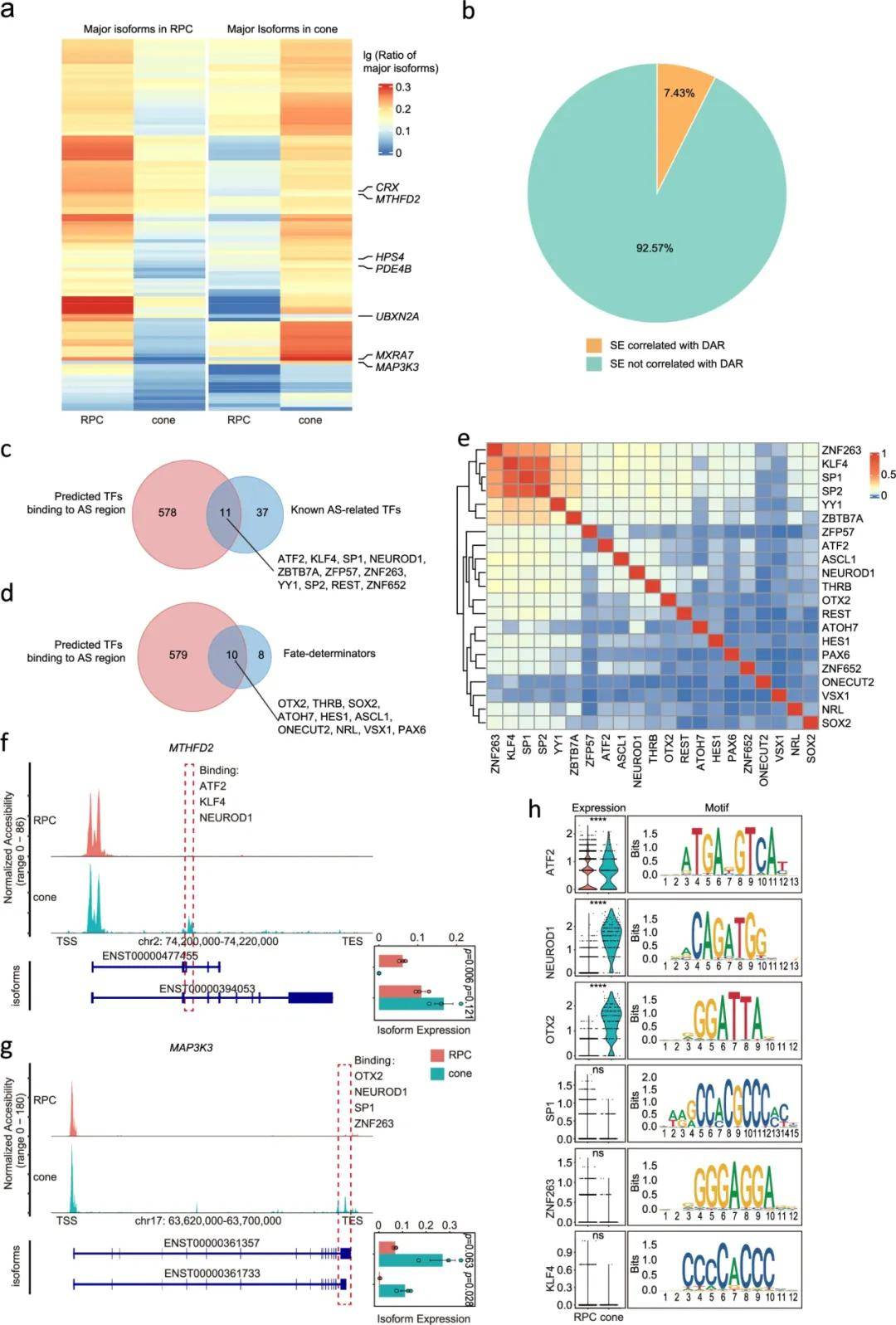 图5 RPC发育过程中RNA异构体选择与染色质可及性和TF的相关性
图5 RPC发育过程中RNA异构体选择与染色质可及性和TF的相关性
PART3、总 结
综上,该研究发展了一种单细胞多组学方法scRICA-seq,在单细胞水平实现了表观遗传学、RNA转录和剪接的整合分析,能够全面分析了染色质可及性、RNA表达和差异异构体选择之间的相关性。利用scRICA-seq,检测到了视网膜祖细胞(RPC)、视网膜神经元、感光细胞前体以及其他视网膜相关细胞类型的特异性基因和异构体表达模式,为进一步理解视网膜的发育机制奠定了基础。