
2025-02-16
前言
在生命科学领域,单细胞测序技术被誉为“打开细胞异质性黑箱的金钥匙”。而单细胞全长转录组测序(Single-cell Full-Length Transcriptome Sequencing),作为该技术的“升级版”,不仅能够解析单个细胞的基因表达全景,还能精准捕捉mRNA的完整结构信息(如可变剪切、融合基因、新转录本等)。这一技术正在推动发育生物学、肿瘤微环境、免疫治疗等领域的研究走向更深层次的突破。
一、技术原理:为什么需要“全长”测序?
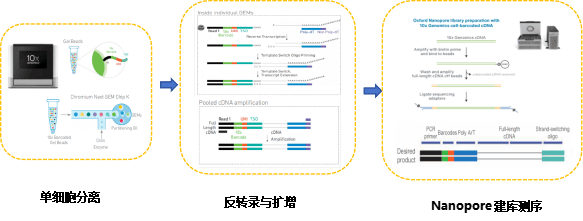
传统单细胞转录组测序(如10x Genomics)通常基于3’或5’端标签捕获,虽能高效获取基因表达量,却丢失了mRNA中间序列的异构体信息。而单细胞全长转录组测序能通过以下关键步骤实现“完整解析”:
1.单细胞分离:通过10x Genomics液滴微流控技术捕获单个细胞;
2.反转录与扩增:在单个油包水结构中反转录合成全长cDNA;
3.建库测序:利用Oxford Nanopore长读长测序平台,直接读取完整mRNA序列。
实现转录本 5’至 3’端全长度覆盖,全面解析基因结构与表达特征。不仅能精确测定基因表达量,还能高效检测转录本异构体(isform)、融合基因及 RNA 层面突变,深度洞察基因结构变异与功能多样性,为揭示疾病发生机制和遗传调控提供详实依据。
核心优势:精准覆盖转录本全长,敏锐捕捉可变剪切、RNA 编辑、基因融合等复杂生物事件,为深入探究基因功能机制提供关键线索,全方位赋能疾病与遗传研究。
二、应用场景:哪些领域正在受益?
单细胞全长转录组测序,可获得完整mRNA序列,随着单细胞研究领域的越来越深入,在癌症肿瘤、神经生物学、发育生物学、免疫学等方向都有广泛应用前景。
1、肿瘤研究:破解耐药性与异质性
肿瘤细胞具有异质性,单细胞全长转录组测序能助我们洞悉其基因表达差异,挖掘肿瘤驱动基因,为精准治疗提供理论支撑。比如,利用 Nanopore 技术,可精准识别肿瘤细胞的融合基因转录本,这些基因与肿瘤发展紧密相连,某些融合基因转录本可能会激活特定的信号通路,促使肿瘤细胞不断增殖和恶化。准确识别这些融合基因转录本,对开发靶向药物意义重大。分析肿瘤单细胞转录组,还能找出药物敏感或耐药的细胞亚群,助力制定更有效的治疗方案。
2、神经科学:追踪疾病的“剪切异常
大脑由数十亿神经元构成,极为复杂。单细胞全长转录组测序可解析不同神经元的分子特征,揭示神经发育基因调控机制和神经系统疾病发病机理。以自闭症、阿尔茨海默病等神经退行性疾病研究为例,运用 Nanopore 单细胞测序,能清晰呈现病变细胞中长链非编码 RNA(lncRNA)转录本的变化,它们可以通过与 DNA、RNA 或蛋白质相互作用,影响基因的表达和细胞的功能。这些 lncRNA 在神经发育和疾病中起重要调控作用,为寻找治疗靶点提供精准线索。
1、发育生物学:解码细胞命运的“异构体开关”
胚胎发育时细胞分化形成不同组织器官。单细胞全长转录组测序可追踪细胞分化轨迹,展现发育中基因表达的动态变化,帮助我们理解生命起源与发展。研究早期胚胎细胞分化为心脏、肝脏等器官细胞时,Nanopore 技术能准确检测细胞分化中的可变剪接事件,同一个基因转录产生的前体 mRNA,通过不同的剪接方式,产生多种不同的成熟 mRNA 转录本,进而翻译出不同的蛋白质异构体。这些异构体在细胞的功能和命运决定中起着关键作用。这对细胞命运决定至关重要,有助于深入探究发育生物学基本规律。
三、单细胞全长转录组测序特有分析
1、isform鉴定分析
通过使用可变的转录起始/终止位点、多聚腺苷酸化位点和/或可变剪接位点,同一个基因可产生不同的mRNA序列(即异构体)-isoform,它们具有不同的蛋白质编码能力。
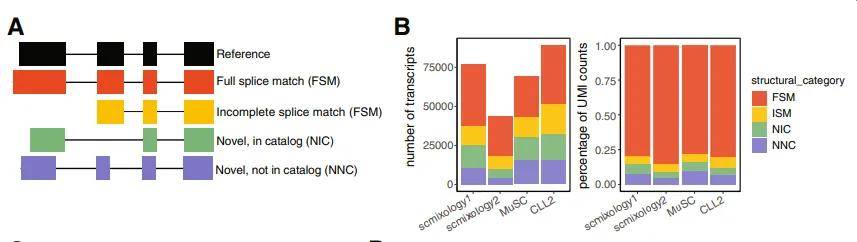 不同全长转录本鉴定分类特征图[1]
不同全长转录本鉴定分类特征图[1]
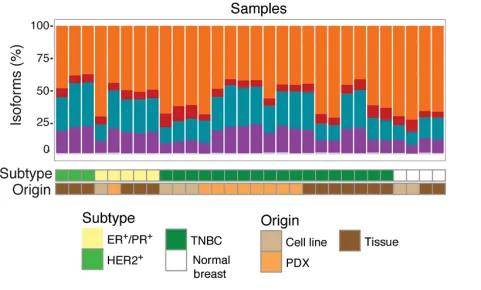 各样本检测到的isoform数量/类别分布图[2]
各样本检测到的isoform数量/类别分布图[2]
2、融合基因检测
可以有效检测出细胞中存在的融合基因,即由染色体易位、倒位等结构变异导致的两个或多个基因融合形成的新基因,对于研究肿瘤等疾病的发生机制、诊断和治疗具有重要意义。
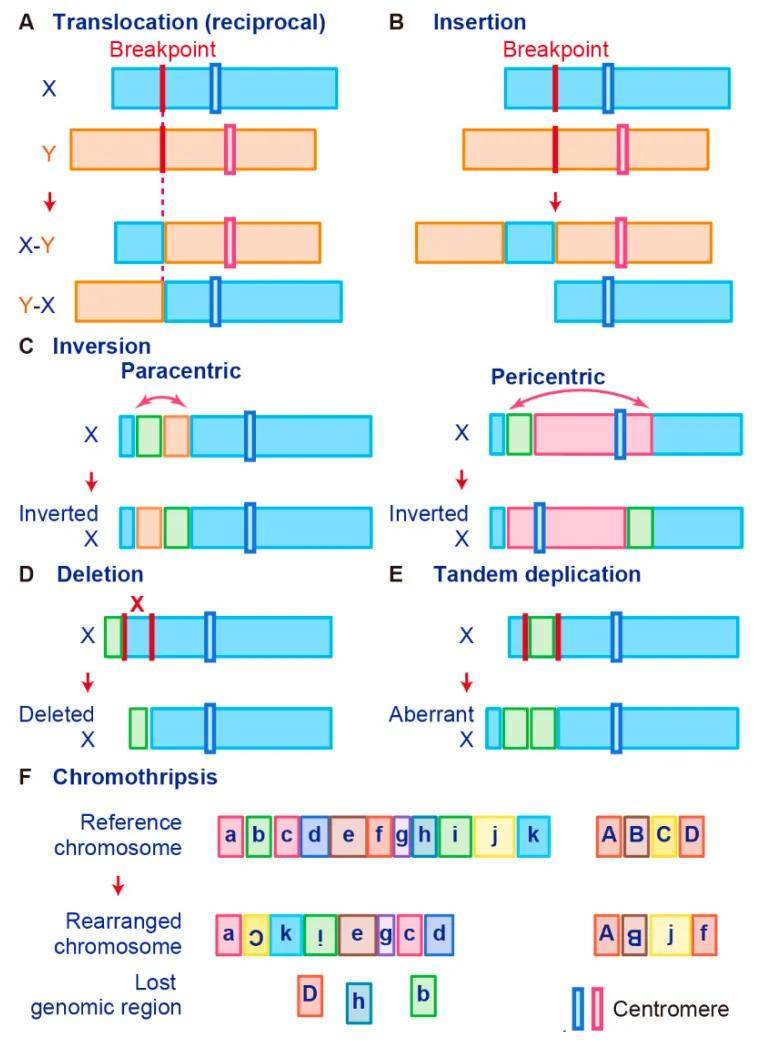 结构性染色体重组形成融合基因[3]
结构性染色体重组形成融合基因[3]
3、非编码 RNA 分析
长链非编码 RNA(lncRNA)分析:可以鉴定和分析细胞中的 lncRNA,包括其表达水平、细胞特异性表达模式以及与其他基因的相互作用,探索 lncRNA 在基因调控、细胞分化和疾病发生中的功能。
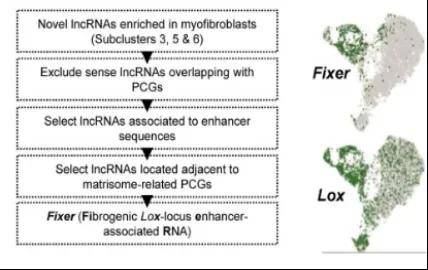 成纤维细胞lncRNA异质分析[4]
成纤维细胞lncRNA异质分析[4]
四、应用案例
案例1:单细胞全长转录组测序揭示卵巢癌的基因融合和细胞转变新图景

组学研究方法:单细胞全长转录组测序、scRNA-seq
组学测序技术平台:10× Genomics、Illumina、PacBio。
研究结果:
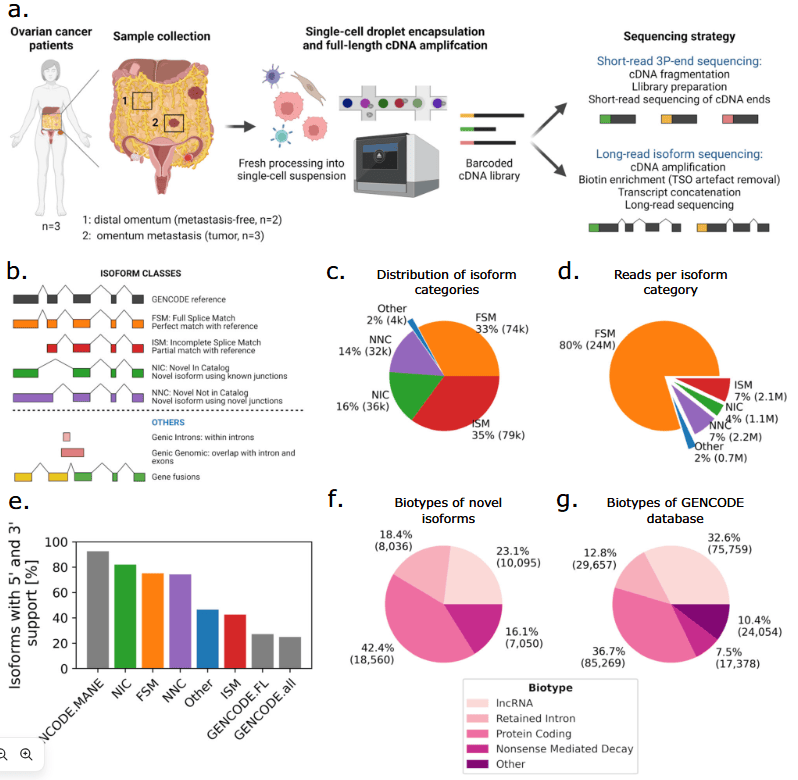
1.构建卵巢癌亚型目录:长读长测序在 2571 个细胞中生成 2.12 亿 HiFi 读长,鉴定出 152,546 种亚型,30% 为新亚型,且新亚型中 42% 为蛋白质编码,验证了长读长测序在鉴定新亚型方面的能力。
2.实现细胞类型鉴定:长读长和短读长测序在细胞类型鉴定上结果相似,细胞聚类的 Jaccard 距离显示二者高度相似,表明长读长测序可独立进行细胞类型鉴定。
3.检测突变和新连接:长读长数据检测到生殖系和体细胞突变,肿瘤细胞中体细胞突变主要集中在 TP53 基因。肿瘤细胞的亚型和转录表达增加,新亚型和新连接比例更高。
4.揭示肿瘤微环境变化:TME 中的间皮细胞和基质成纤维细胞在转移样本中发生细胞状态转变,通过 TGF-β/miR - 29 / 胶原蛋白轴,间皮细胞部分转化为癌症相关成纤维细胞(CAFs)。
5.分析癌症中差异亚型使用:HGSOC 细胞与远端细胞相比,960 个基因的亚型使用发生差异,部分基因的最高表达亚型生物型改变,且使用基因水平信息估计蛋白质表达会导致 20% 的高估。
6.捕获基因融合:检测到 34 个融合转录本,其中 IGF2BP2::TESPA1 融合在 Patient 2 的 HGSOC 细胞中高度表达,短读长数据会误将其报告为 TESPA1 过表达。
该研究表明,长读长测序能够更全面地呈现癌症特异性变化,为精准肿瘤学研究和个性化治疗带来新机遇,但仍需进一步提高测序深度以捕获低丰度转录本。
案例2:单细胞全长转录组测序揭示小鼠脑神经元发育成熟不同阶段转录本的多样性
组学研究方法:单细胞全长转录组测序、scRNA-seq
组学测序技术平台:10× Genomics、Illumina、Nanopore
研究结果:
1.鉴定转录本亚型:研究利用单细胞全长转录组系统分析了胚胎小鼠大脑中的转录异构体多样性,总共发现33002个已经注释的转录本和4388种新的异构体。
2.研究通过筛选不同簇间转录异构体表达的基因,观察到一些基因存在细胞类型选择性亚型使用现象,如 Clathrin light chain A(Clta)和 Myosin Light Chain 6(Myl6)在神经元成熟过程中会发生明显的亚型转换。
研究结果表明单细胞全长转录组可用于揭示不同转录本在不同神经元成熟过程中不同阶段的表达情况。
案例3:单细胞全长转录组测序揭示发育中及成年小鼠和人类大脑中的特定剪接模式

组学研究方法:单细胞全长转录组测序、scRNA-seq
组学测序技术平台:10× Genomics、Illumina、Nanopore
研究结果:
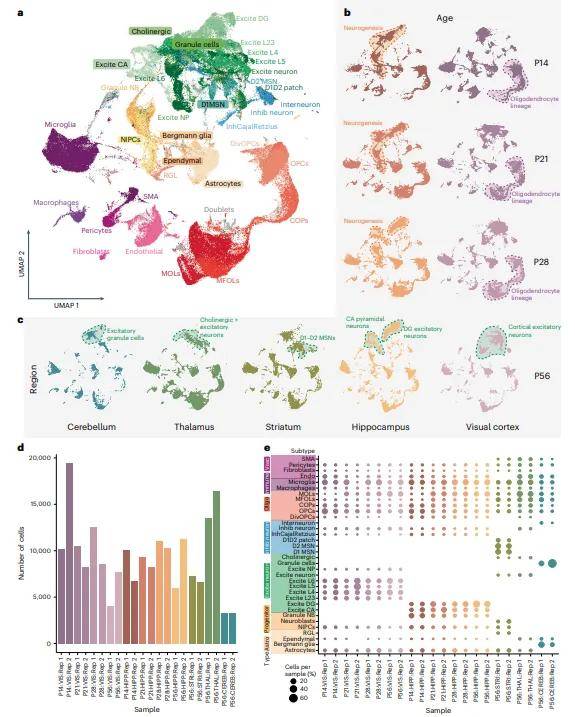
1.细胞类型的异构体变异特征:研究收集了小鼠在出生后不同时间点(P14、P21、P28和P56)以及成年时期(P56)的五个脑区样本(视觉皮层、海马、小脑、纹状体和丘脑),对其进行单细胞RNA测序和单细胞全长转录组测序,共得到4种主要细胞类型(神经元细胞、神经胶质细胞、血管细胞和免疫细胞)。不同细胞类型在异构体变异模式上存在显著差异,神经元的异构体变异高于神经胶质细胞和祖细胞。兴奋性神经元的异构体变异主要体现在细胞亚型间,而抑制性神经元在年龄和脑区之间变异明显。
2.大脑区域特异性剪接:丘脑和小脑的星形胶质细胞表现出较强的转录起始位点(TSS)、聚腺苷酸化(poly (A))位点和外显子调控。神经元在不同脑区的 TSS 和 poly (A) 位点使用差异比星形胶质细胞更高,且 TSS 使用更具区域特异性。
3.可变外显子的特征与功能:鉴定出 4557 个高可变外显子(hVExs)和不同类别的极可变外显子(EVExs),这些外显子与细胞功能和疾病相关。部分 EVExs 在人类和小鼠中保守,且受 RBP 调控,与疾病相关的 sQTLs 有关。
4.发育过程中的异构体调控:青春期(P21 - P28)大脑区域特异性会短暂增加,部分基因的异构体表达在该时期发生显著变化。同时,在发育过程中,剪接和基因表达定义的神经胶质成熟存在不一致性,且外显子变异呈现波动模式。
研究揭示了大脑中 RNA 亚型表达在细胞类型、脑区和发育阶段的广泛差异,明确了不同外显子类别与蛋白质功能的关联,发现小鼠成年细胞类型的变异在人类中大多保守,且发育过程中的异构体波动比成年脑区间的差异更大,强调了研究异构体表达时同步记录基因表达和剪接的必要性。
参考文献:
[1]Tian L, Jabbari JS, Thijssen R, et al. Comprehensive characterization of single-cell full-length isoforms in human and mouse with long-read sequencing. Genome Biol. 2021;22(1):310. Published 2021 Nov 11. doi:10.1186/s13059-021-02525-6.
[2] Veiga DFT, Nesta A, Zhao Y, et al. A comprehensive long-read isoform analysis platform and sequencing resource for breast cancer. Sci Adv. 2022;8(3):eabg6711. doi:10.1126/sciadv.abg6711.
[3] Taniue K, Akimitsu N. Fusion Genes and RNAs in Cancer Development. Noncoding RNA. 2021;7(1):10. Published 2021 Feb 4. doi:10.3390/ncrna7010010
[4] Aghagolzadeh P, Plaisance I, Bernasconi R, et al. Assessment of the Cardiac Noncoding Transcriptome by Single-Cell RNA Sequencing Identifies FIXER, a Conserved Profibrogenic Long Noncoding RNA. Circulation. 2023;148(9):778-797. doi:10.1161/CIRCULATIONAHA.122.062601