
2025-04-25
Highlights
1.精准拆解单细胞注释中使用的小工具,解锁细胞新的生物学意义;
2.单细胞注释的小工具总结与应用;
3.派森诺单细胞产品部细胞注释灵活运用各种小工具注释细胞,解锁细胞新的身份。
在单细胞测序技术飞速发展的今天,如何从海量数据中精准解析每个细胞的身份和功能,成为科研攻关的核心挑战。无论是肿瘤微环境中的恶性细胞的鉴定,还是免疫细胞的异质性解析,细胞注释都扮演着“解码”的角色。前文我们详细介绍了单细胞注释中的遇到的难点,而本文将从四个维度,精准拆解单细胞注释全流程中使用到的小工具,助您掌握解锁细胞身份的终极密码!
一、自动化注释:高效判定细胞身份的双引擎
在单细胞鉴定中,自动化注释工具的选择至关重要。SingleR和Seurat是目前最常用的两种工具,它们各有优势和局限性。
1、SingleR
原理:SingleR通过计算每个细胞与参考数据集的基因表达相似性,直接分配到最匹配的细胞类型标签。
步骤:1.计算每个细胞的基因表达谱的可变基因与参考细胞数据集表达谱之间的Spearman相关性;2.对每种细胞类型的得分进行计算并取固定分位数作为细胞类型得分;3.对所有的标签重复此操作,然后将得分最高的标签作为此细胞的注释;4.通过热图可视化注释结果。
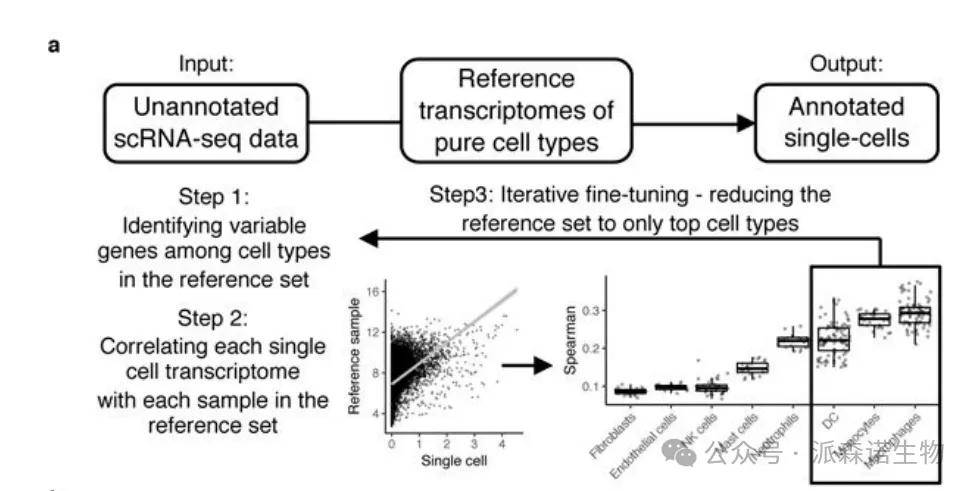 图一 SingleR自动化注释的流程与步骤
图一 SingleR自动化注释的流程与步骤
2、Seurat
原理:通过CCA或PCA对齐参考与查询数据,识别锚点细胞并转移标签,结合标记基因二次验证。
步骤:1.基于参考数据集的锚点转移:通过CCA(典型相关性分析)或PCA对齐查询数据与参考数据集,识别跨数据集的相似性细胞(称为锚点),利用这些锚点校正批次效应,并将参考数据的标签转移到查询数据中;2.基于标记基因的自动分类:结合已知标记基因数据库,通过差异基因分析筛选每个聚类的特征基因,自动匹配预定义的细胞类型标签。
在实际应用中,选择哪种工具取决于具体的研究需求和数据特点。如果参考数据与查询数据匹配度高,Seurat的锚点转移方法将是一个不错的选择,因为它能够提供更可靠和详细的注释结果。
二、人工注释:标记基因的黄金标准
尽管自动化注释工具在单细胞鉴定中发挥着重要作用,但人工注释仍然是确保鉴定结果准确性的关键环节。人工注释主要基于标记基因的表达模式,通过差异基因分析进一步确定细胞类型。在实际操作中,研究人员可以利用Doheatplot、Dotplot和Featureplot等可视化工具,直观地展示基因表达模式和细胞聚类结果,从而更精准的注释出细胞类型。
与自动化注释相比,人工注释虽然较为费时,但准确性相对较高,尤其在鉴定罕见细胞类型方面具有独特的优势。通过结合自动化注释和人工注释,研究人员可以充分发挥两者的优势,提高单细胞鉴定的整体效率和准确性。
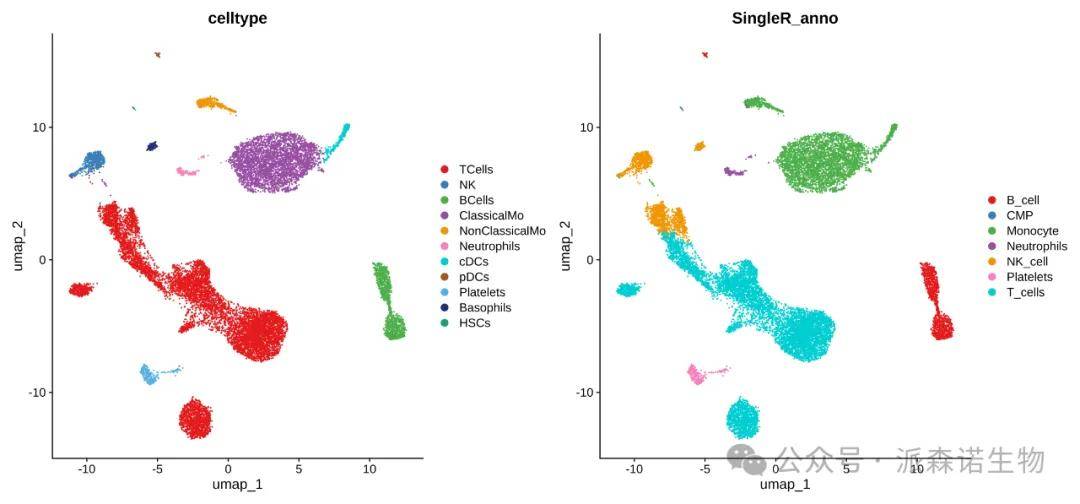 图二 自动化注释和人工注释的比对(左图为人工注释的结果,右图为自动化注释的结果)
图二 自动化注释和人工注释的比对(左图为人工注释的结果,右图为自动化注释的结果)
三、富集分析:解锁细胞新的功能
除了传统的基因表达分析和标记基因注释外,基于功能富集的注释为单细胞鉴定提供了更深入的视角。功能富集分析主要包括GO(Gene Ontology)富集分析和KEGG(Kyoto Encyclopedia of Genes and Genomes)通路富集分析。
GO富集分析通过分析确定基因列表在GO分类的生物过程(BP)、分子功能和细胞组分中的富集情况,帮助我们理解这些基因的功能和在细胞内的定位。
KEGG通路富集分析则侧重于确定基因列表在KEGG通路中的富集情况,揭示基因在代谢和信号传导等生物学过程中的作用。
通过基于功能富集的注释,研究人员可以从基因集层面深入解析细胞的功能状态,为单细胞鉴定提供更全面和深入的解读。例如,在一项对9例卵巢样本的研究中,研究人员通过降维聚类分群和经典标记基因注释得到了8种细胞类型,随后通过每种细胞类型的差异基因GO富集分析结果,从侧面验证了单细胞注释的准确性。
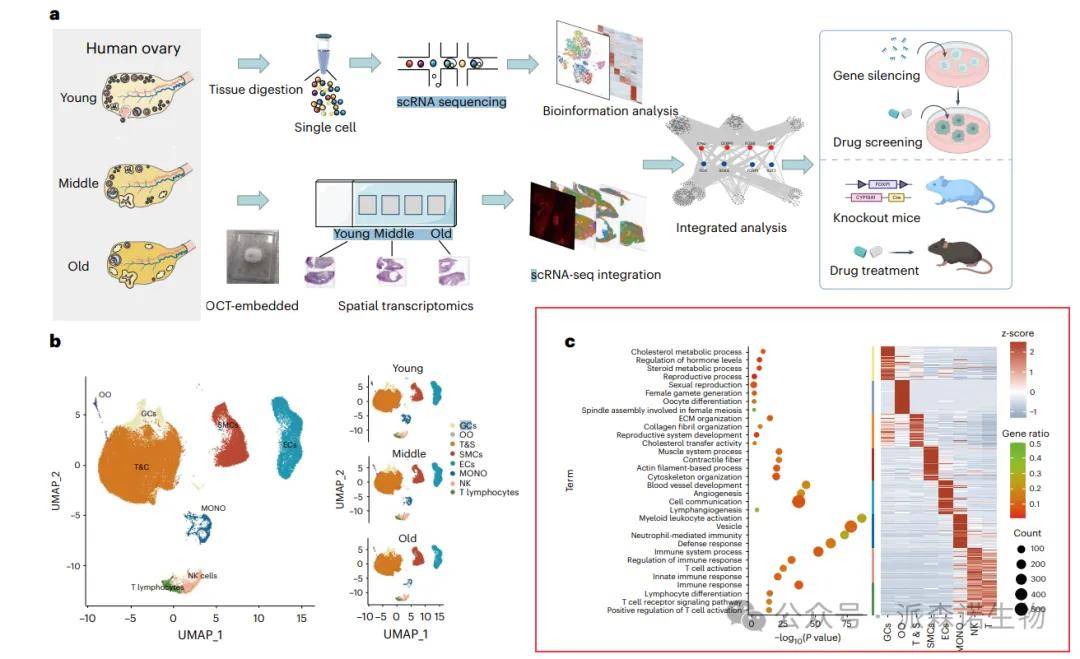 图三 GO富集结果验证注释的准确性
图三 GO富集结果验证注释的准确性
在单细胞测序分析中,功能富集工具如GSEA(Gene Set Enrichment Analysis)和GSVA(Gene Set Variation Analysis)也被广泛应用于辅助细胞类型注释和功能状态解析。GSEA通过评估预定义基因集在差异基因列表中的富集程度,帮助研究人员识别显著富集的基因集。而GSVA则是一种非参数的无监督分析方法,可以直接从基因表达矩阵中计算每个样本中特定基因集的变异分数。
这两种方法各有特点,GSEA需要预先进行样本间差异基因分析,而GSVA则不需要,这使得它们在不同的研究场景中具有各自的优势。
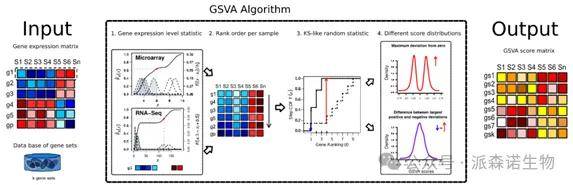 图四 GSVA的流程
图四 GSVA的流程
四、辅助判断细胞的其他手段:基因集打分与CNV分析
除了上述的注释方法外,基因集打分和CNV(Copy Number Variation)分析也为单细胞鉴定提供了有力的辅助手段。
1.基因集打分工具如AUCell、AddModuleScore和UCell。
AUCell:基于基因排序的累积富集面积(AUC值),适合单细胞功能活性评估;
UCell:快速秩次标准化,支持大规模数据(如10万细胞)的干性评分;
AddModuleScore:Seurat内置工具,适合初步筛选高活性细胞亚群。
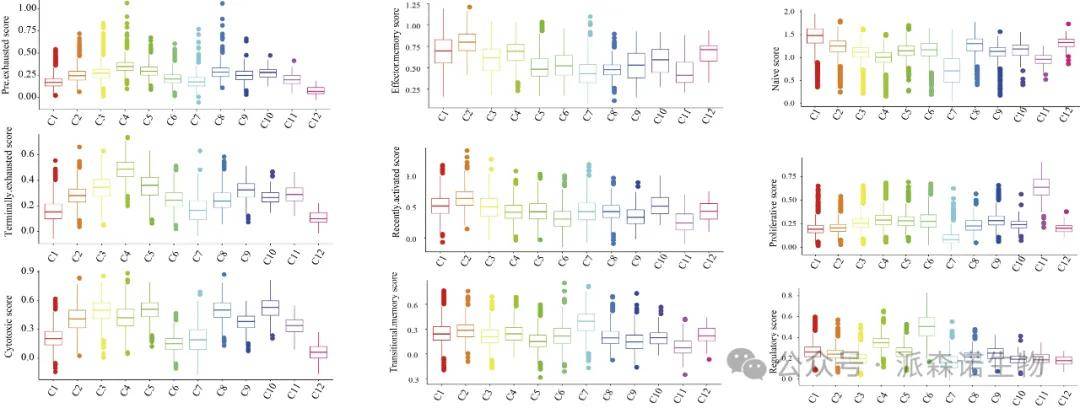 图五 基因集分析评估细胞的状态
图五 基因集分析评估细胞的状态
分享一篇来自2022年6月Front. Immunol的宫颈癌(CC)单细胞文章,主要研究正常宫颈、上皮内瘤变、原发性肿瘤和转移性淋巴结组织在单细胞水平,在不同阶段免疫细胞和间充质细胞的组成、谱系和功能状态。文章在对T细胞进行分析时,为了评估每个细胞的潜在功能并确认CC进展中每个阶段的免疫状态,使用了AddModuleScore计算每个细胞簇的功能模块得分。值得注释的是,cluster4表现出较高的耗竭评分,而clsuter3,5,8,10的细胞毒性的评分较高,该打分的结果在一定程度上能够帮助了解细胞的功能和状态,完善细胞注释的结果。
2.CNV分析则是鉴定肿瘤细胞的重要手段。常用的工具包括inferCNV和copyKAT。 这两种方法各有优势,inferCNV依赖正常细胞参考,结果更可靠,但计算耗时较长;copyKAT无需正常细胞参考,计算速度较快,适合大规模数据。
inferCNV通过比较肿瘤细胞基因表达与参考细胞基因表达,以热图的形式展示染色体上的基因相对表达量,从而确定体细胞大规模染色体拷贝数改变的证据。
 图六 inferCNV的数据结果展示
图六 inferCNV的数据结果展示
而copyKAT则结合贝叶斯方法与层次聚类,计算单个细胞的基因组拷贝数分布,并定义出亚克隆结构。
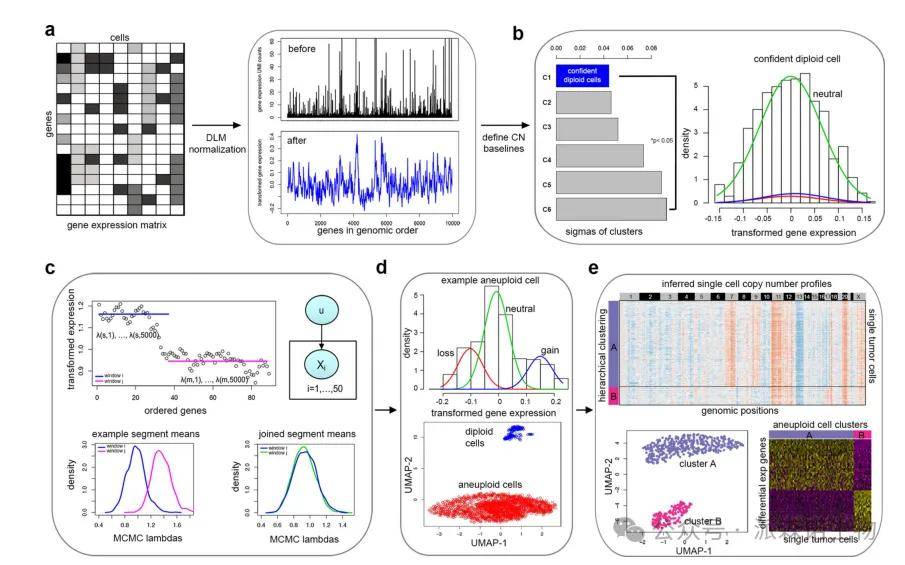 图七 copyKAT的流程
图七 copyKAT的流程
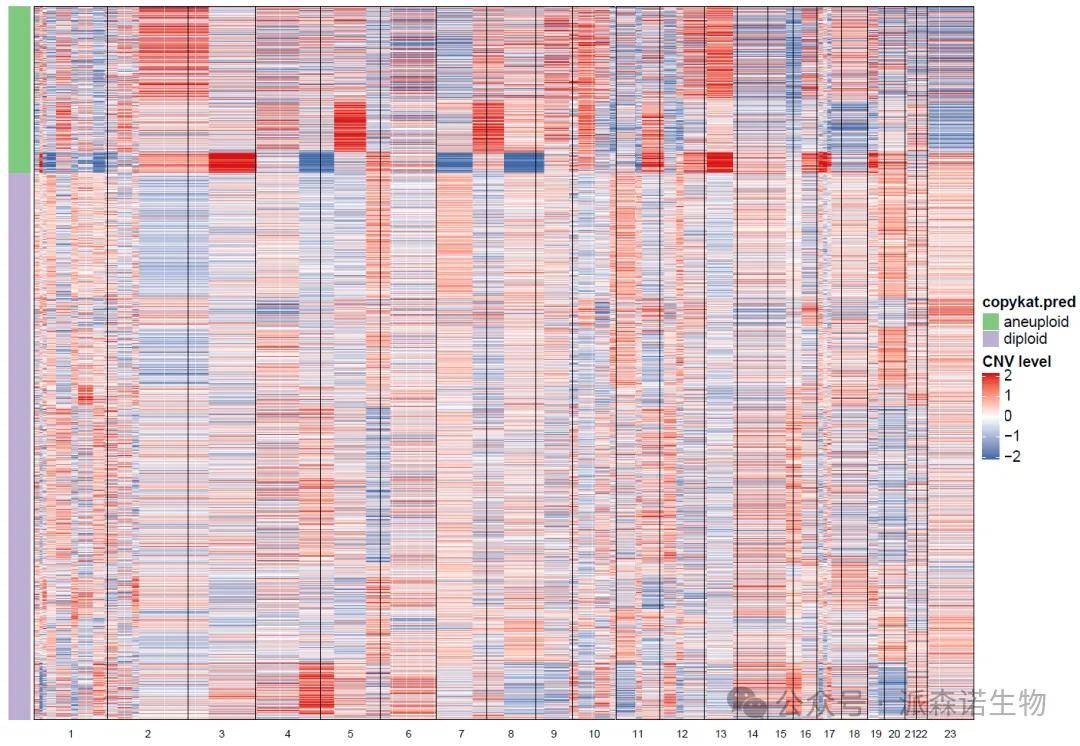
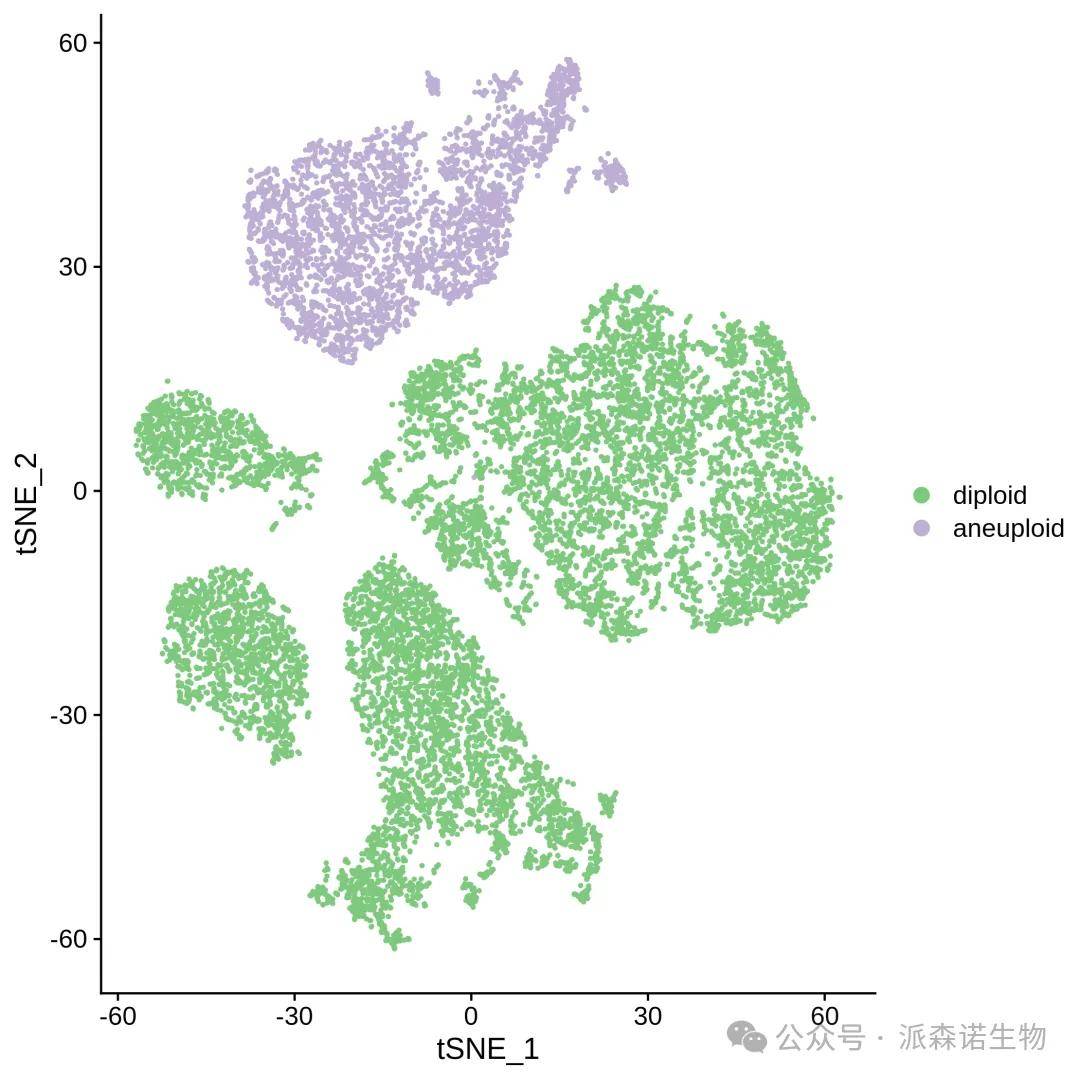 图八 copyKAT的结果展示
图八 copyKAT的结果展示
在实际应用中,研究人员可以根据研究需求选择合适的CNV分析工具。例如,在一项对鼻咽癌数据的研究中,研究人员使用inferCNV鉴定Epithelial cells中的恶性细胞,通过下载公开的正常上皮细胞的单细胞数据作为参考,成功鉴定出了2787个恶性细胞。

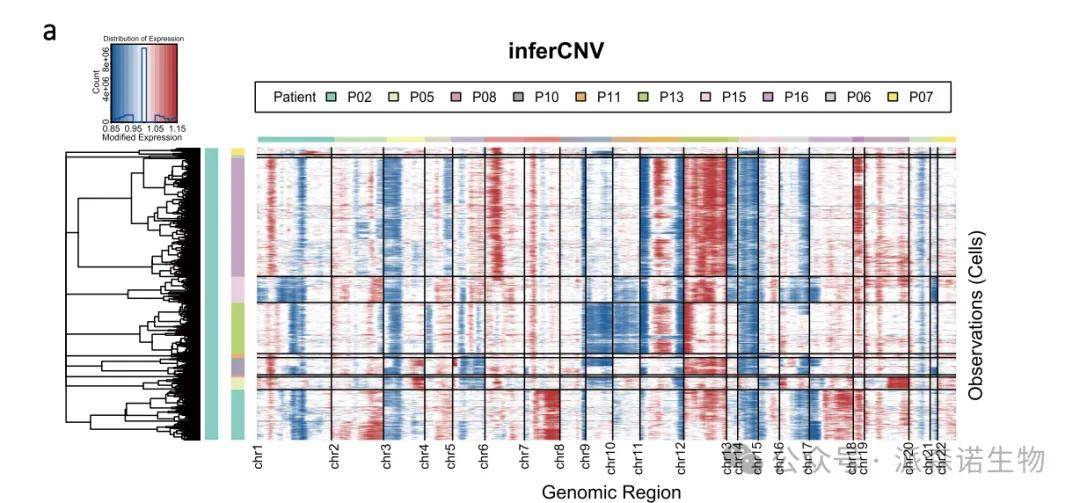 图九 inferCNV鉴定恶性细胞
图九 inferCNV鉴定恶性细胞
五、总结:巧用工具推动注释完成
细胞注释的本质,是将基因表达矩阵转化成一个个有生命、有意义的生物学故事。无论是利用何种工具进行注释,最终的目的都是为了揭开细胞异质性,为生物学发育、疾病的机制提供新的见解。