2025-05-08
Highlights
4种拟时序分析的差异:
monocle2:识别细胞分化路径及关键基因模块,适合简单线性或分支轨迹,计算效率高。
monocle3:基于UMAP降维的分支轨迹分析,支持非线性轨迹和大规模数据,分支分辨率更高。
Cytotrace:评估细胞分化水平,预测细胞分化的相对状态和方向,无需先验经验,适合探索性分化研究。
RNA velocity:结合剪接动力学预测细胞未来状态,揭示细胞命运的时间箭头。
拟时序分析可用于理解细胞在不同功能状态之间的转变,预测细胞的分化及发育轨迹,根据轨迹分布和特征基因的表达变化来确认分化起点和终点。
拟时序分析的各类软件之间有什么区别,适合什么场景呢?下面我们来详细解释。monocle2和monocle3是最常用的两种方法,因此详细介绍。
一、monocle2
原理:类似机器学习的方法,根据输入数据的特征基因构建轨迹树,细胞在树中的路径反映分化过程。之后优化细胞在低维空间中的位置,同时考虑细胞间的相似性,形成分化的轨迹,筛选随时间变化的基因模块,帮助识别驱动分化的基因。
输入文件:RDS
分析流程:
1. 用带有注释信息的RDS,构建CellDataSet(CDS)对象。
2. 提取用于分析的基因:
基于monocle的dispersionTable选择高可变基因:默认筛选条件为平均表达量>0.1。
另外还有基于differentialGeneTest的方法、基于seurat选择高变基因的方法以及基于seurat中FindAllMarkers方法,可以根据需求自行选择。
3.降维:
reduceDimension函数利用DDRTree降维算法将高维的单细胞基因表达数据映射到低维空间,便于推断细胞的发育轨迹或分化路径。
4.计算细胞拟时间
orderCells函数用于构建细胞轨迹的线性或分支结构、生成伪时间等,默认根是State 1(“State”是Monocle用来表示轨迹树分支的术语),可以使用root_state 指定根节点。
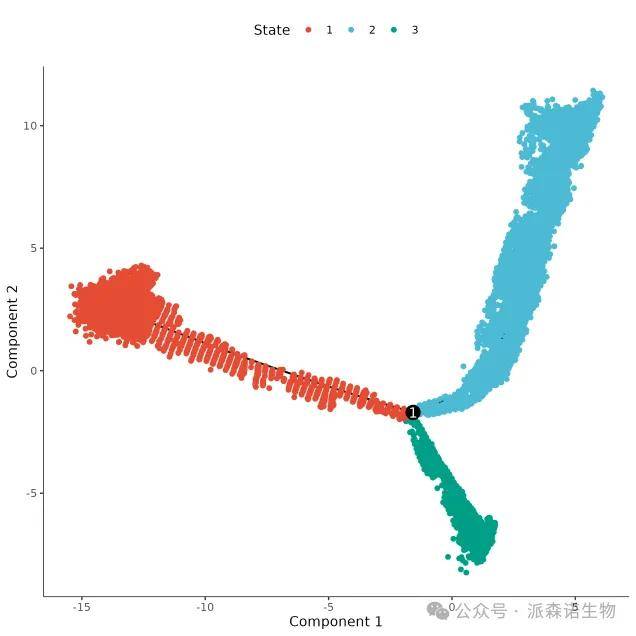
5.轨迹图展示
参数color_by 用于指定根据哪个变量(如细胞类型、伪时间、状态等)对细胞进行着色。
6.基因可视化
plot_genes_in_pseudotime函数用于分支分析中基因表达的可视化,展示基因在分支点的表达差异
输出结果:
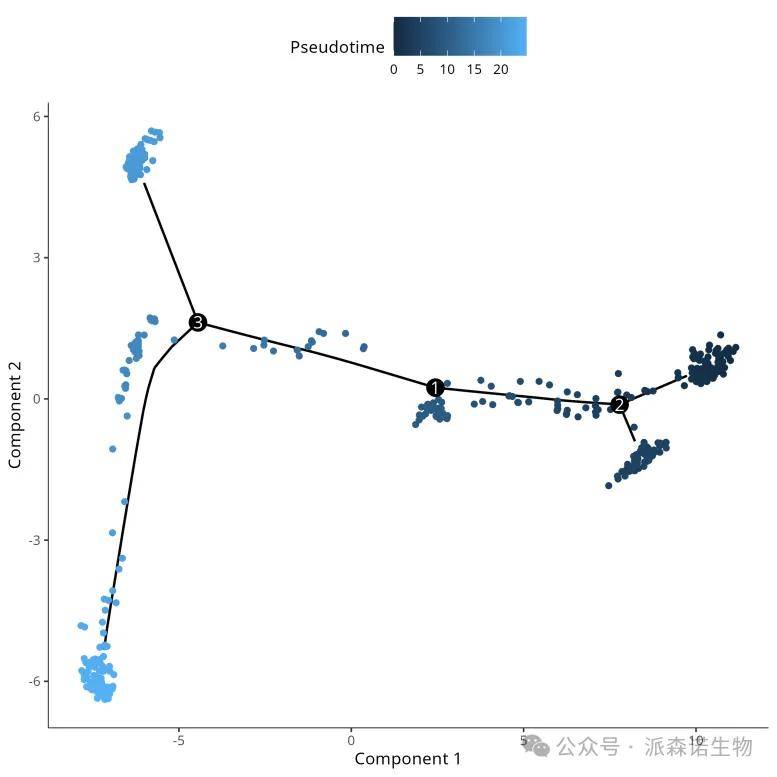
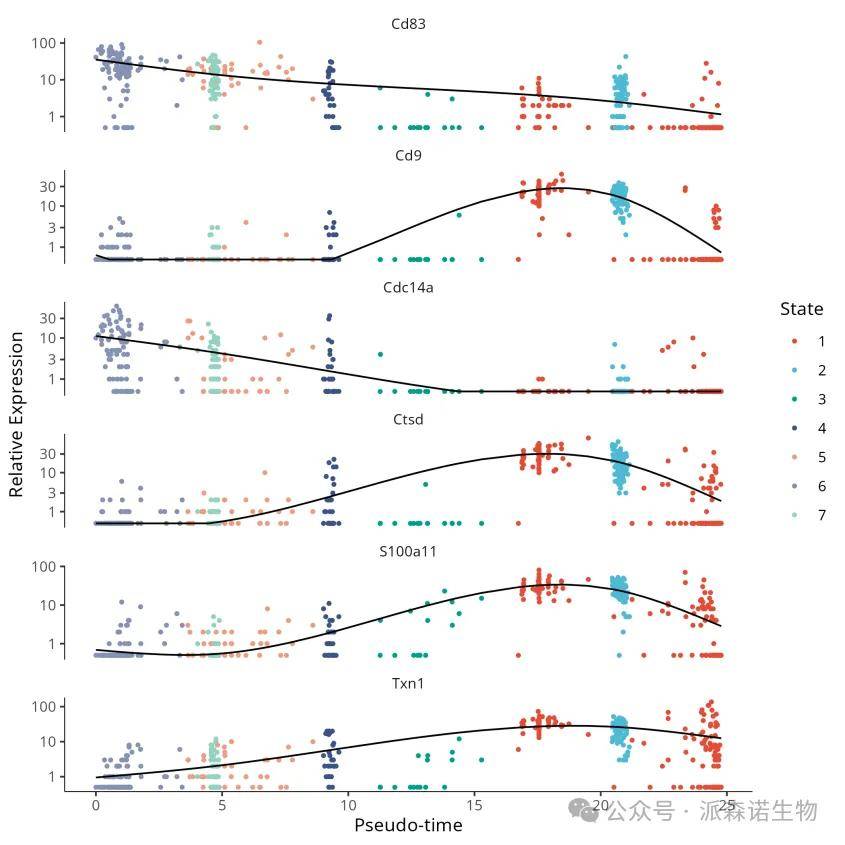
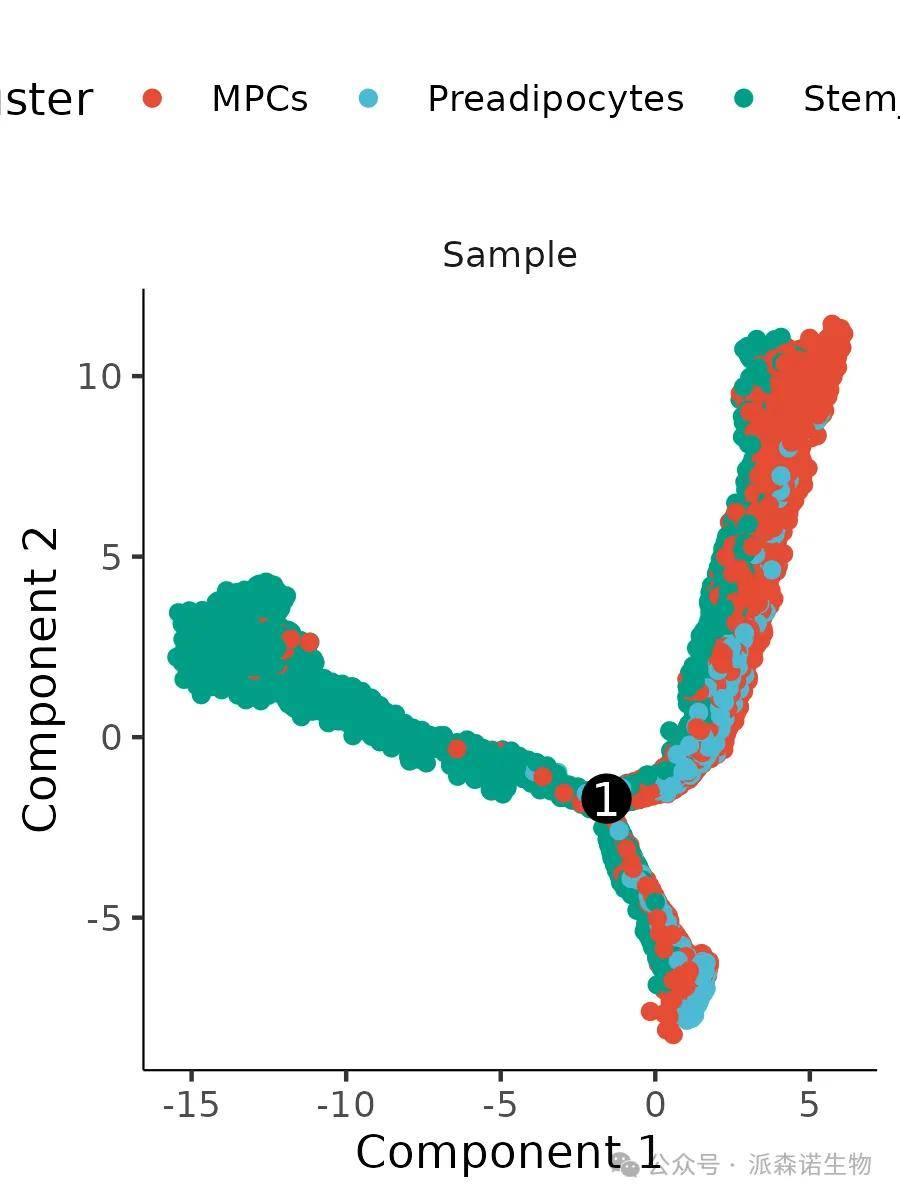
应用场景:
发育生物学:研究胚胎或器官发育中的细胞命运决定(如造血系统分化)
疾病机制:解析肿瘤演进轨迹,识别恶性转化中的关键基因
二、Monocle3
原理:用UMAP将细胞投影到低维空间,然后采用分区图抽象的方法,构建一个最近邻图,通过寻找图中的最长路径来识别分化轨迹。
输入文件:RDS
分析流程:
1. 用带有注释信息的RDS,构建CDS对象。
2. 降维,导入umap坐标,聚类分群
reduceDimension函数使用 PCA 对高维数据进行预处理,之后使用UMAP进行降维聚类分群。然后从RDS里导入umap坐标,使monocle3的结果与之前大类注释的结果一致。
3. 轨迹推断与细胞排序
learn_graph函数根据降维后的数据,学习细胞之间的邻接关系,并构建graph,反映细胞在发育或分化过程中的潜在路径。root_cells指定根节点
4. 拟时轨迹差异分析
(1)寻找拟时轨迹差异基因
graph_test函数用于识别沿细胞发育轨迹变化的基因,并通过计算莫兰指数来评估这些基因在细胞群体中的空间自相关性。
莫兰指数(morans_I),值在-1至1之间,0代表此基因没有空间共表达效应,1代表此基因在空间距离相近的细胞中表达值高度相似。
(2)挑选top基因画图展示
Trace_genes_sig函数筛选出空间自相关性(由莫兰指数衡量)最强的前10个基因,进行后续的分析或可视化。
(3)绘制基因表达趋势图
plot_genes_in_pseudotime函数用于分支分析中基因表达的可视化,展示基因在分支点的表达差异,筛选表达量大于0.5的基因。
(4)寻找共表达模块
find_gene_modules()、aggregate_gene_expression()等函数从基因表达数据中提取特定基因、进行模块检测、按细胞组聚合基因表达数据并最终生成热图以可视化基因表达模式
输出结果:
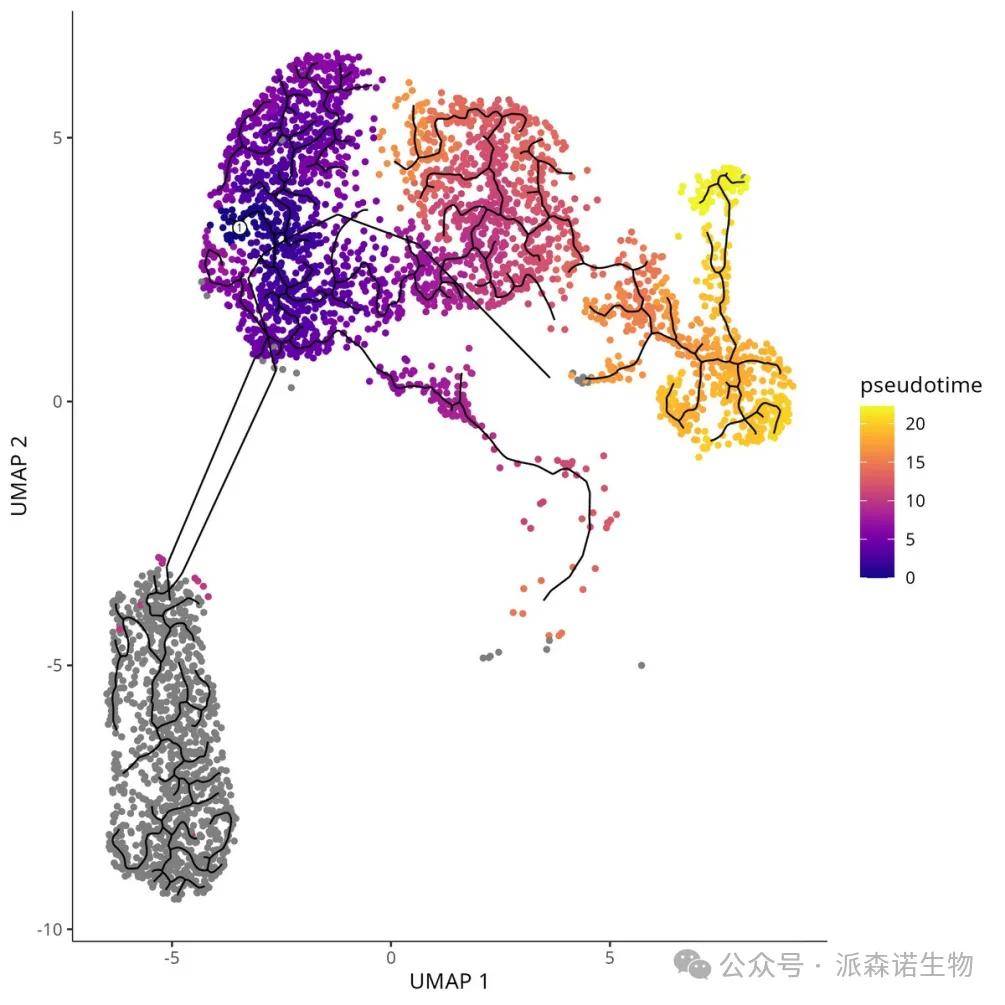
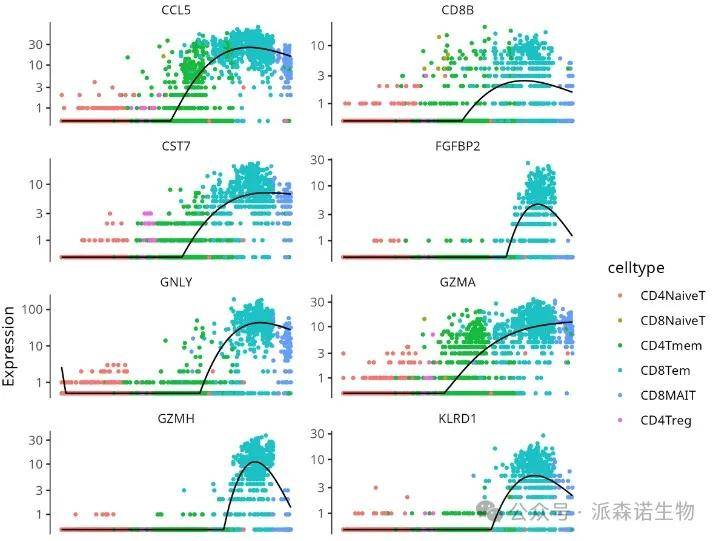
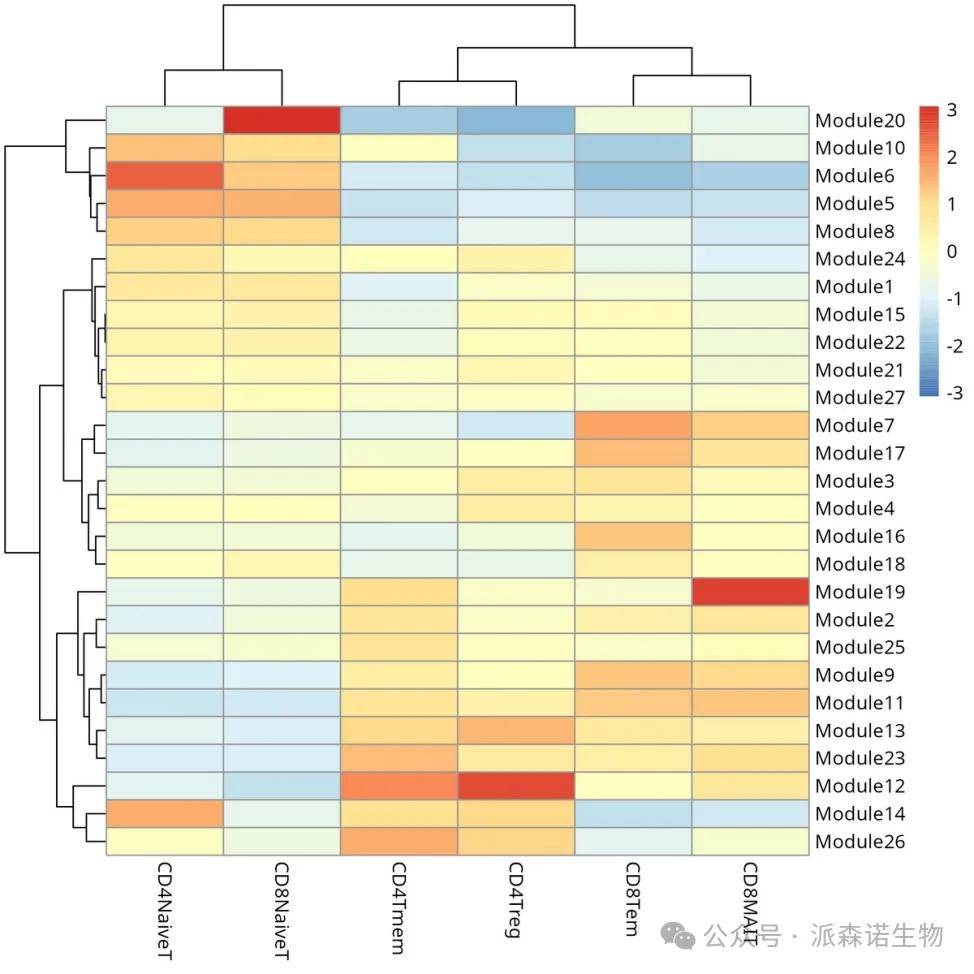
应用场景:
复杂系统分化:研究具有多分支路径的细胞命运选择
大规模数据:处理大样本量数据
与monocle2相比,monocle3的优势:
从UMAP图识别发育轨迹,可以继承Seurat的质控、批次校正和降维分析结果,实现“一张图”展现细胞的聚类、鉴定和轨迹分析结果。自动对UMAP图分区(partition),可以选择多个起点,轨迹分析算法的逻辑更符合生物学现实。
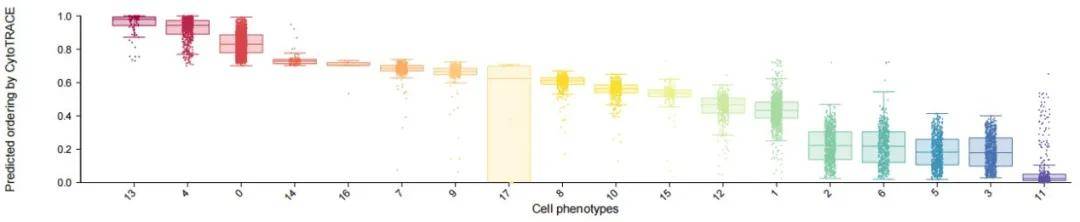
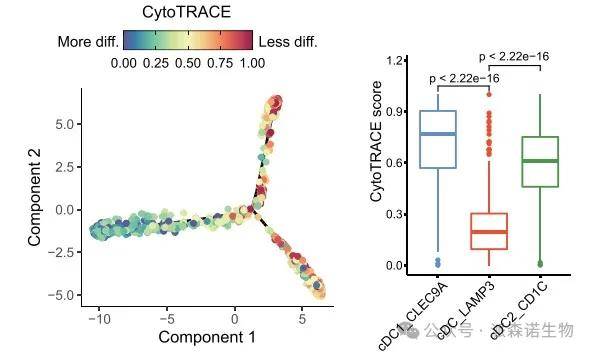
三、Cytotrace
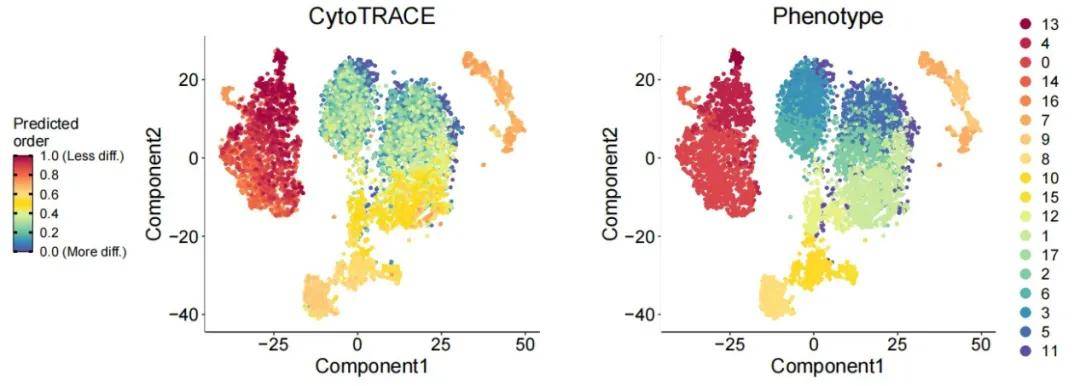
原理:
通过量化基因表达的随机波动来推断细胞的分化状态,其核心假设是:未分化的干细胞或祖细胞具有更高的表达噪声,以维持其多潜能性;而分化细胞因功能特化,表达模式更稳定。
输出结果:
四、RNA velocity
原理:
通过量化每个细胞中未剪切mrna和剪切mRNA的丰度,推测基因转录速率和斜率参数,因此Velocity可在不知发育过程的前提下,预测谱系的方向。
输出结果:
![]()
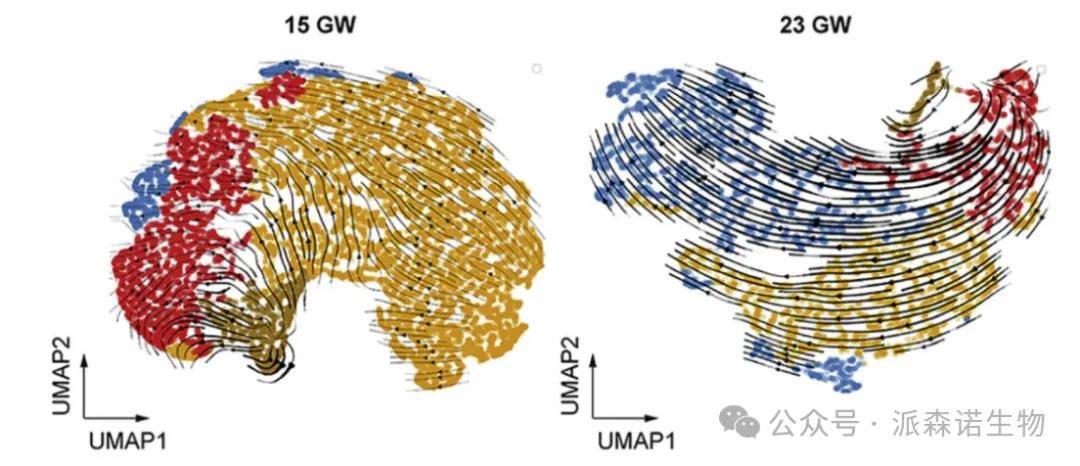
应用场景:
动态过程解析:研究组织中的细胞迁移方向
轨迹验证:为Monocle推断的轨迹提供方向性证据
瞬时状态捕捉:分析稳态组织中缓慢变化的细胞过程


