2025-05-21
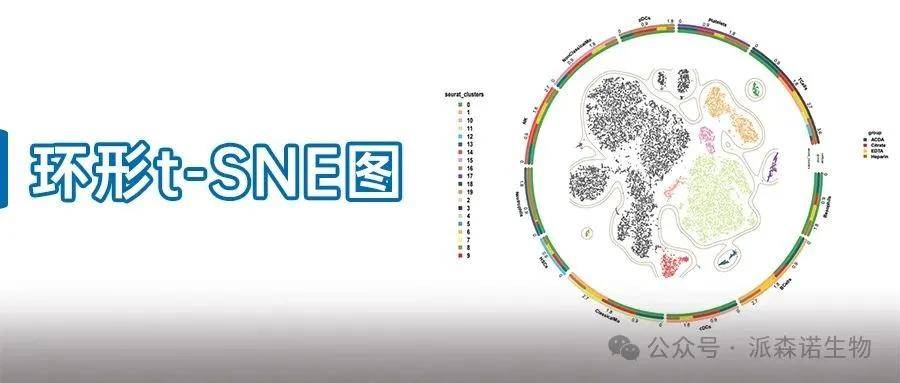
单细胞分析中的t-SNE(t-分布随机邻域嵌入)分析是一种强大的可视化方法,它通过将高维数据降维到二维或三维空间,把相似的细胞聚成不同的群。目前单细胞文章主图中基本都有此类分群图,但是只放一个t-SNE已经略显单调。为了增强主图的表现力,我们参考高分文章的plot1cell软件包,更新了环形t-SNE图,在降维图上更直观地展现了不同细胞群的分组/细胞类型间细胞数目变化。
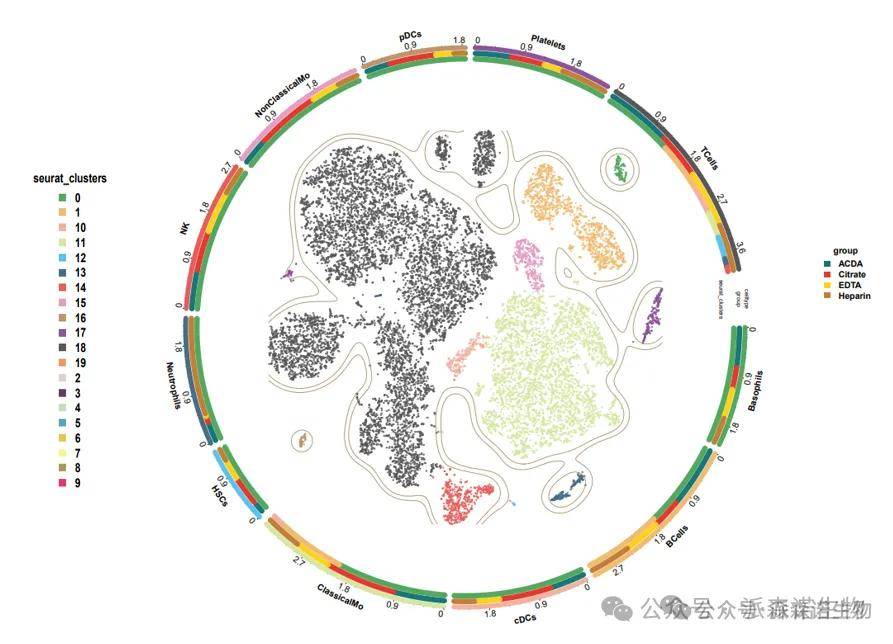
图1 环形t-SNE图
图1中展示出不同细胞类型的t-SNE分布图。外圈轨道三层分别为不同细胞类型中细胞类型/分组/clusters的占比,中心散点为不同细胞类型的t-SNE图,散点颜色对应细胞类型的轨道颜色。在绘图时,我们使用流程输出的RDS作为输入文件,绘制代码示例如下:
1、数据准备
从RDS中提取降维信息和meta信息,可以是t-SNE或者umap的维度;
seu_obj = readRDS(“rename_seurat.rds”)
metadata<-seu_obj@meta.data
metadata$Cluster<-seu_obj@active.ident
metadata$dim1<-as.numeric(seu_obj[[‘tsne’]]@cell.embeddings[,1])
metadata$dim2<-as.numeric(seu_obj[[‘tsne’]]@cell.embeddings[,2])
metadata$x<-transform_coordinates(metadata$dim1, zoom = 0.7)
metadata$y<-transform_coordinates(metadata$dim2, zoom = 0.7)
metadata$cells<-rownames(metadata)
rownames(metadata)<-metadata$cells
data_plot <- cell_order(metadata)
此时我们会得到一个类似于meta.data的矩阵,包含如下核心信息:
表1 绘图准备数据
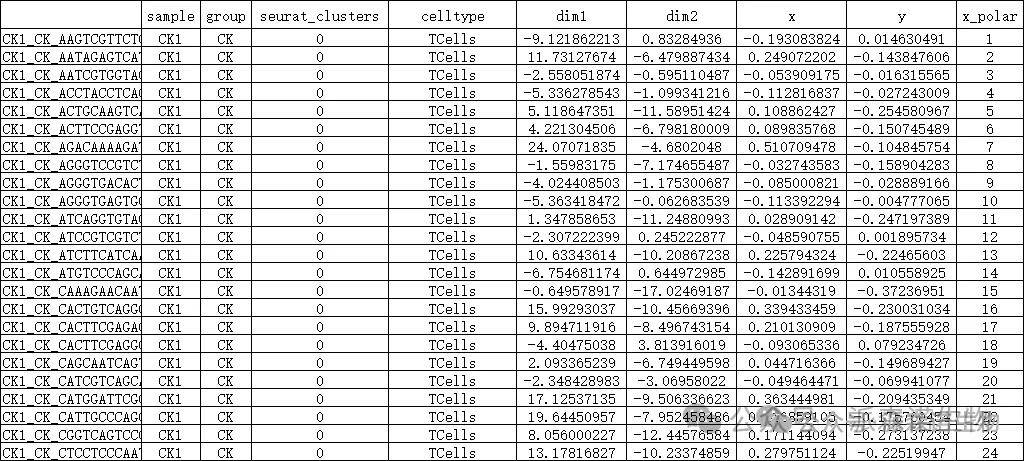
可以看出,相比meta.data信息,绘图数据新增了dim1/dim2维度信息,缩放后的x/y维度,以及细胞的排序。
2、中心降维图
基于1整理的绘图数据,我们首先绘制t-SNE 分群图,以及最外层的细胞类型的占比环形轨道。可以看出,轨道的不同类型细胞也是按照细胞群对应的颜色标注。点的色板和大小均可自定义调整。
plot_circlize2(circ_data,do.label = F, pt.size = 0.3, kde2d.n = 1000, repel = T,
label.cex = 1.4,col.use = my36colors[1:length(unique(seurat_ob$celltype))],
bg.color = background)
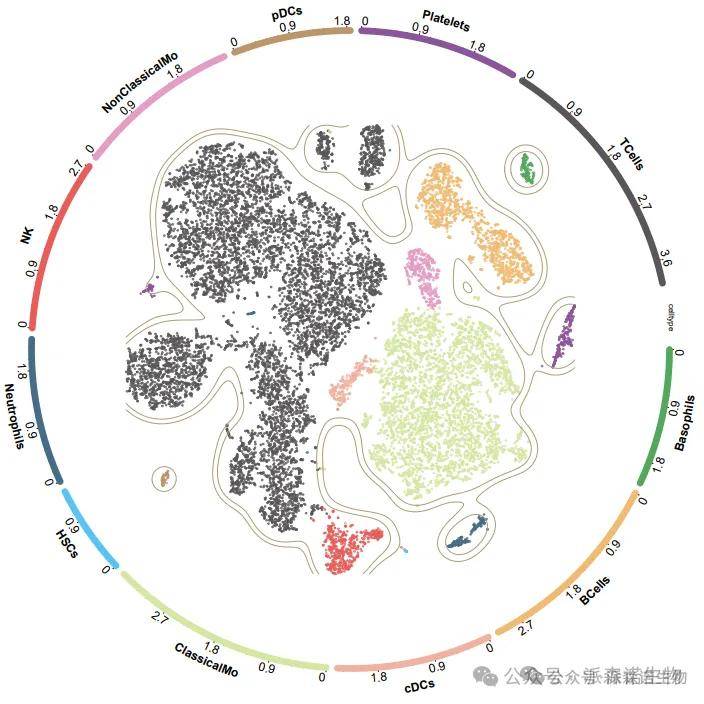
图2 中心降维图
3、添加自定义轨道
然后我们就把group/clusters的轨道添加到图上,也可以根据自定义的分类增加轨道。这里建议3个轨道左右,可视化效果相对会更美观一些。当然,其中的颜色,字体大小和图例位置都可以根据需求调整代码参数。
circ_data$group = factor(circ_data$group,levels= sort(unique(as.vector(circ_data$group))))
group_colors <- Retro_color[1:length(names(table(seurat_ob$group)))]
legend(x=1.25,y=0.3,fill= group_colors ,border=color,box.col=color,title='group',
legend = sort(unique(as.vector(circ_data$group))),text.font = myfont)
add_track2(circ_data, group = "group",colors = group_colors, track_num = 2, track_lwd = 10)
circ_data$seurat_clusters = factor(circ_data$seurat_clusters,levels = sort(unique(as.vector(circ_data$seurat_clusters))))
sample_colors <- Retro_color[1:length(names(table(seurat_ob$seurat_clusters)))]
legend(x=-1.4,y=0.3,fill= sample_colors ,border=color,box.col=color, title='seurat_clusters',legend= sort(unique(as.vector(circ_data$seurat_clusters))) , text.font = myfont)
add_track2(circ_data, group = "seurat_clusters",colors = sample_colors, track_num = 3, track_lwd = 10)

图3 增加自定义轨道
环形t-SNE不仅可以绘制单细胞的降维聚类可视化图,还能够通过在同一图片中整合多种元数据轨道(如细胞类型注释、样本来源信息等),精细化呈现不同样本,不同细胞群体的分布特征,为科研人员对复杂细胞数量变化特征及样本间细胞的组成差异研究提供直观的可视化依据。
绘制图片或者复现代码过程中,如果老师遇到疑惑,欢迎拨打我们的热线电话或者联系我们的驻地销售。
参考文献:
1.Mapping the single-cell transcriptomic response of murine diabetic kidney disease to therapies. Cell Metab. doi: 10.1016/j.cmet.2022.05.010


