
2025-05-29

文章信息
文章题目:Nine high-quality Anas genomes provide insights into Anas evolution and domestication
中文标题:九个高质量的鸭属基因组为鸭属的进化和驯化提供了见解
发表期刊:Cell Reports
影响因子:7.5
发表时间:2025年4月
涉及组学:基因组组装,转录组
摘 要
目前,鸭子的物种多样性以及驯化情况还不太清楚。为此,研究团队构建了 8 个染色体水平的 Anas(鸭属)基因组,加上北京鸭的基因组,共同来研究鸭属的进化和驯化过程。结果发现,相比较常染色体,Z染色体受渗入影响小些,且其局部系统发育比较稳定。从 Z 染色体角度来看,鸭属中的绿头鸭(Anas platyrhynchos)和灰鸭(Anas zonorhyncha)物种形成时伴随着持续的雌性偏向基因流动,鸭子的驯化历史也发生了改变。基于此,通过构建鸭属的泛基因组,确定了家鸭和野鸭之间好几种差异化的结构变异(SVs),这些 SVs 可能会调节它相邻的基因,比如 GHR 基因和 FER 基因,这两类基因是有潜力成为 “驯化基因” 的。还发现长末端重复(LTR)反转录转座子爆发活动加速了鸭子的驯化,特别是促使 MITF 基因和 IGF2BP1 基因的功能发生了显著转变。这些发现生动形象地为理解动物进化过程提供了参考案例。
前 言
研究自然和家庭环境中生物体的进化过程意义重大,驯化生物不仅为人类提供必要资源,还在社会发展中发挥关键作用,但密集的选择性育种导致其遗传多样性受限,阻碍性状改进并降低适应性和抵抗力。尽管最近研究尝试引入野生物种的遗传物质来增加驯化生物的遗传多样性,但在动物中面临诸多挑战,如跨物种杂交困难、野生亲缘物种的有限多样性以及功能变异的未充分探索。
鸭子是 Anas 和其他 Anatidae 物种的统称,广泛分布且表现出多种表型,大多数物种具有迁徙性和性二态性。家鸭品种繁多,且野鸭具有广泛的跨物种杂交能力,这为跨物种育种提供了广阔潜力。鸭子是研究动物系统发育、进化和驯化的理想模型,但其进化历史和驯化仍不清楚,尤其是只有绿头鸭被驯化的原因。本研究组装了1只家鸭和7种野鸭的高质量基因组,并结合北京鸭基因组进行全面进化分析,构建基于SV的泛基因组,探索鸭子间差异SVs对功能基因的影响,并研究转座子元件在鸭驯化中的作用。
研究思路
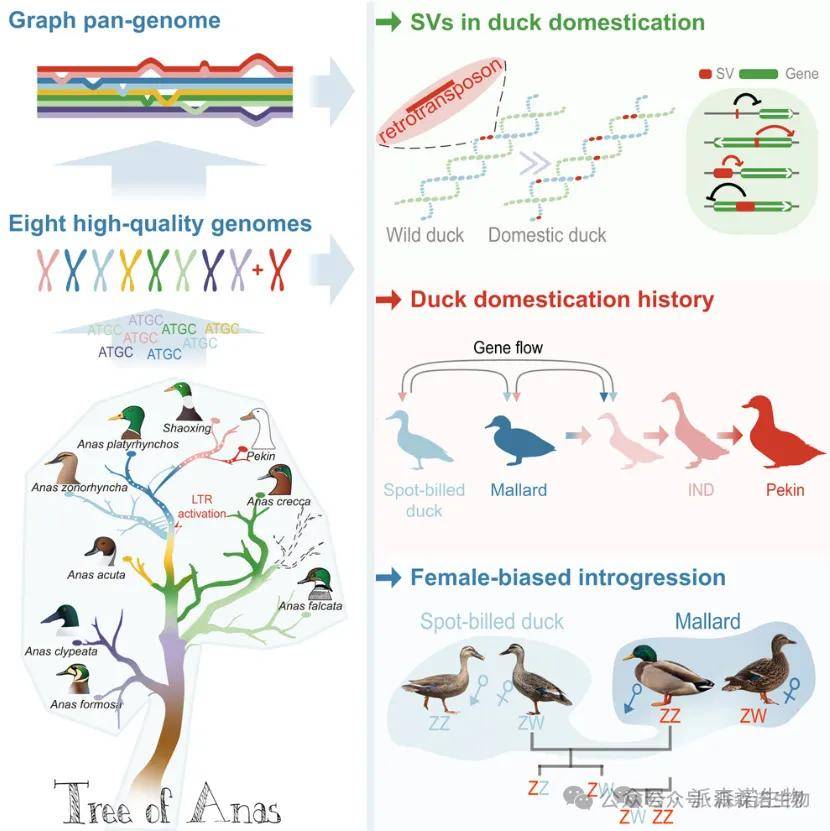
研究内容
1、高质量的基因组组装和注释
对 Anas 属的8种鸭进行了基因组测序,包括1只家鸭(绍兴鸭)和7种野鸭(绿头鸭、中国斑嘴鸭、针尾、欧亚绿翅蓝鸭、镐状蓝绿色鸭、北方铲子鸭和贝加尔湖蓝绿色鸭)。测序采用正常 Nanopore 或 PacBio HiFi 读数、超长 Nanopore 读数、NGS 读数以及 BioNano 和 Hi-C 数据组合,成功组装出8个基因组,分别锚定在40-41条染色体上,其重叠群 N50 和支架 N50 分别在11.9-32.8 Mb和67.2-77.6 Mb之间。进一步分析发现,8个鸭基因组的重复元件含量在13.61%-20.90%之间。其中,短散布核元件占比约0.05%且相对稳定,而其他四种转座因子(TE)的含量在不同基因组中变化较大,范围在11.36%-18.40%之间。值得关注的是,微型反向重复 TE 在8个基因组中表现出显著差异,从0.09%(北方铲子鸭)到6.30%(中国斑嘴鸭),导致约81 Mb的序列变异。
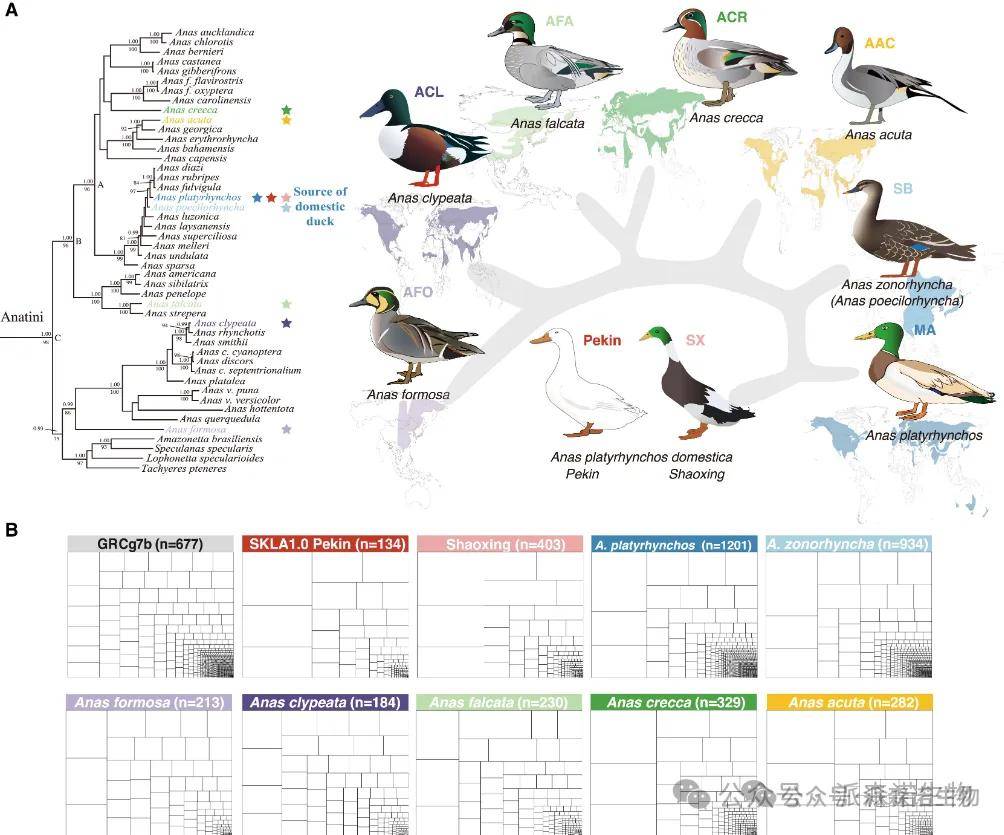
图1 物种信息和基因组组装树状图
2、Anas基因组的保守和分化
全基因组比对显示 Anas 基因组高度同步,染色体重排数量有限,染色体长度变化微小。研究鉴定了不同类型的保守元件和加速进化元件,发现家鸭和 Anas 特有的高度保守元件分别在基因侧翼区域富集,并与特定的生物过程相关。通过比对鸭子的 NGS 数据,鉴定了大量的 SNP 和插入缺失等变异,这些变异在基因侧翼区域表现出富集,且许多具有功能后果。Fst分析揭示了 Anas 的两种不同的分歧模式,表明基因调控区域可能在鸭子物种形成和驯化中起关键作用。
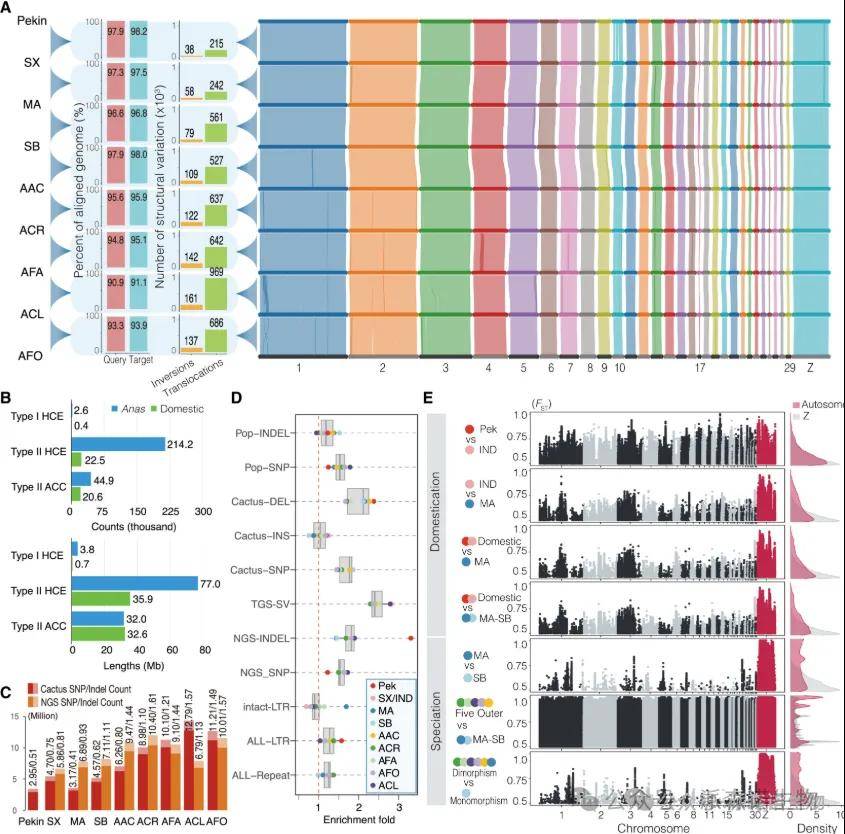
图2 Anas 基因组比较分析
3、Anas 基因组中广泛的系统发育不一致
为了解 Anatidae的系统发育,研究人员利用9个Anas物种和16个非Anas Anatidae物种的4倍简并位点构建系统发育树,并用MCMTREE估计发散时间。结果显示,Anas物种约在1800万年前与Anserinae分化,约386万年前进一步分化,ACL和AFO约331万年前分化,与PSMC分析结果一致。Anas物种经历了两次种群下降,与冰期相吻合。进一步用Z染色体的4d位点构建系统发育树,发现与常染色体树存在不一致。为评估ILS影响,研究人员生成局部系统发育树,发现常染色体和 Z染色体4d位点树的支持率随窗口大小减小而降低,且局部系统发育的不稳定性主要在 “野鸭复合体” 中观察到。聚合树与4d位点树保持一致,进一步加强了常染色体和 Z 染色体系统发育之间的不一致。研究人员还对154个组合进行 D 统计量分析,检测到显著的基因流信号,表明杂交和 ILS 导致 Anas 中存在广泛的系统发育不一致,使其成为系统发育研究的宝贵模型。
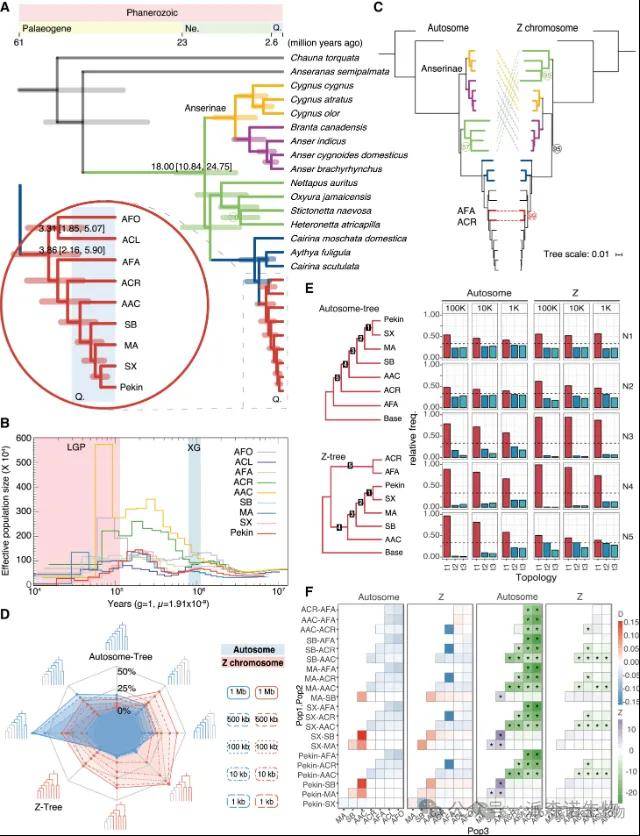
图3 Anas系统发育和广泛的系统发育不一致
4、MA 和 SB 的物种形成和鸭驯化历史的推断
Z染色体表现出较低的基因流或不完全谱系分选(ILS)影响,其系统发育树显示绿头鸭(MA)与其他物种的分化更为明确,而常染色体树中部分MA与其他MA分化并与斑嘴鸭(SB)聚集。在ADMIX分析中,Z染色体分析显示MA与SB明显区分,而常染色体分析中部分MA与SB无法区分,表明从SB到MA存在不对称基因渗入。通过拓扑加权分析,发现Z染色体上存在抗渗入区域,包括关键的DMRT位点和SLC24A2基因。这些分析结果表明,MA与SB之间的分化和驯化过程涉及复杂的基因流动和选择事件。基于这些分析,构建了一个鸭子驯化历史模型,包括4d位点树、从SB到MA的连续不对称渗入以及初始驯化阶段的基因流动,估计SB与MA分离约142千年前,家鸭祖先从MA分化约19千年前,北京鸭从IND分离约887年前。
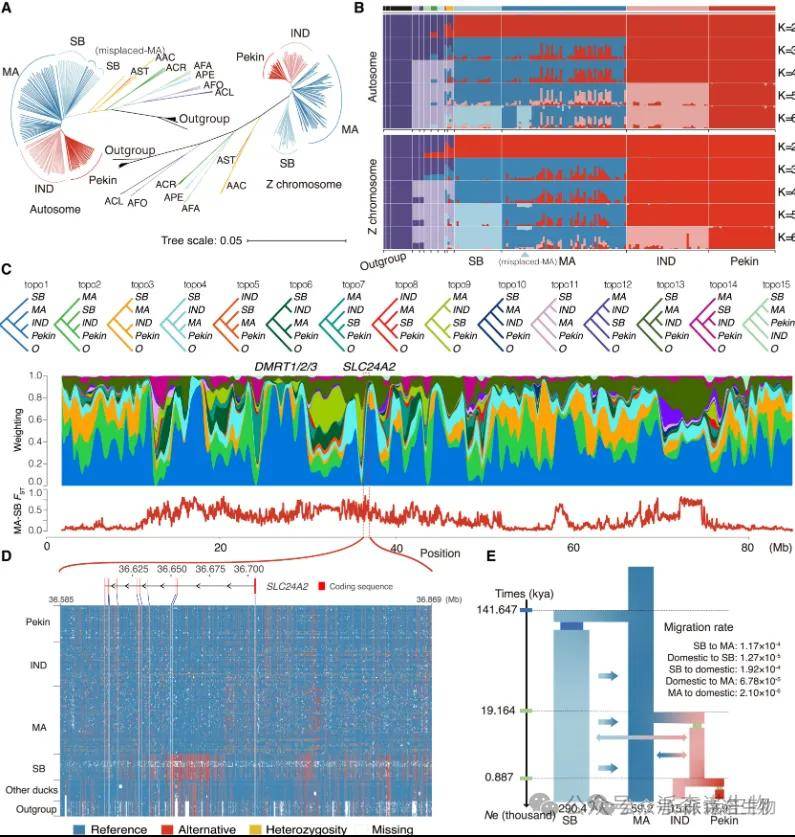
图4 常染色体Z染色体比较和驯化模型
5、Anas泛基因组的构建和差异 SVs 的鉴定
为了构建参考基因组并研究结构变异(SVs)在鸭子驯化和物种形成中的作用,研究人员利用北京鸭基因组(SKLA1.0)和新组装的8个Anas基因组生成了Anas泛基因组。进一步对22个鸭种/品种的200个个体的NGS读数进行比对,鉴定出234,931个SVs,其中22,072个SVs被所有Anas物种共享,而一些SVs在特定物种或家鸭中特异存在。通过比较家鸭与野鸭之间的不同SVs,发现与生长激素受体(GHR)、视网膜发育相关基因(FER)和羽毛颜色相关基因(如MITF和ADAMST12)的表达有显著差异,这些SVs可能通过影响基因表达促进了鸭子的驯化。
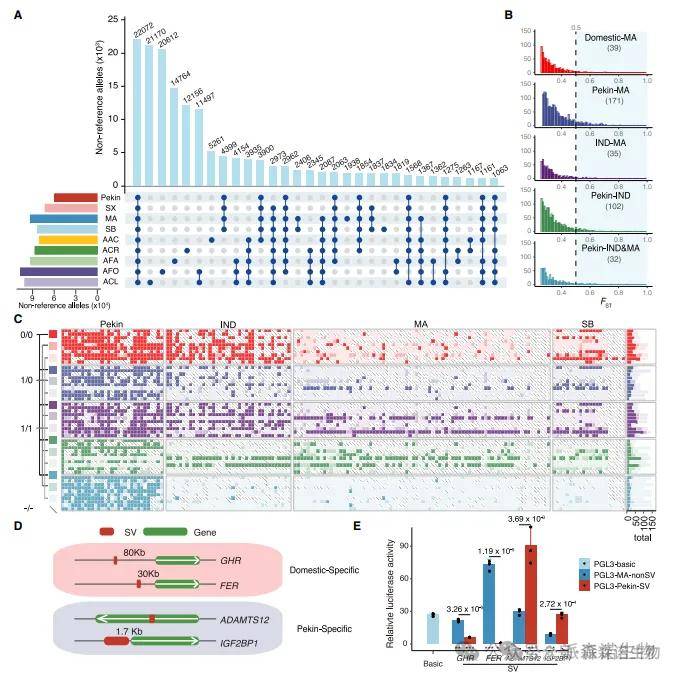
图5 鸭子SV分布及其对基因表达的影响
6、Anas泛基因组的构建和差异 SVs 的鉴定
在Anas属的进化过程中,LTR反转录转座子(RTs)是结构变异(SVs)的重要来源,对基因表达产生影响。研究发现,Anas属经历了两次LTR RT突发:一次近期爆发约122千年前,涉及家鸭及其近缘物种(如北京鸭、绍兴鸭、绿头鸭和斑嘴鸭);另一次古代爆发约924千年前,涉及其他五个远亲物种。每个Anas基因组包含独特的完整LTR RTs(完整LTR),数量从159到665不等,其中北京鸭与绍兴鸭共享51个完整的LTR,表明近期的LTR RT爆发在鸭驯化过程中持续发生。完整LTR的分歧时间较短,表明其最近产生。LTR序列的系统发育树显示出六个分支包含大量扩增的LTR RTs,内分支长度较短,与LTR RT最近出现的突发峰值一致。LTR爆发对基因功能的影响显著,超过53.1%的完整LTR位于基因或其侧翼区域,影响了153个基因,其中67.4%的基因在家鸭和野鸭之间表现出差异表达。功能分析显示,这些LTR RTs通过产生SVs和调节基因表达,在鸭子的进化轨迹和驯化中发挥重要作用,增强了鸭子对家庭环境的适应能力。
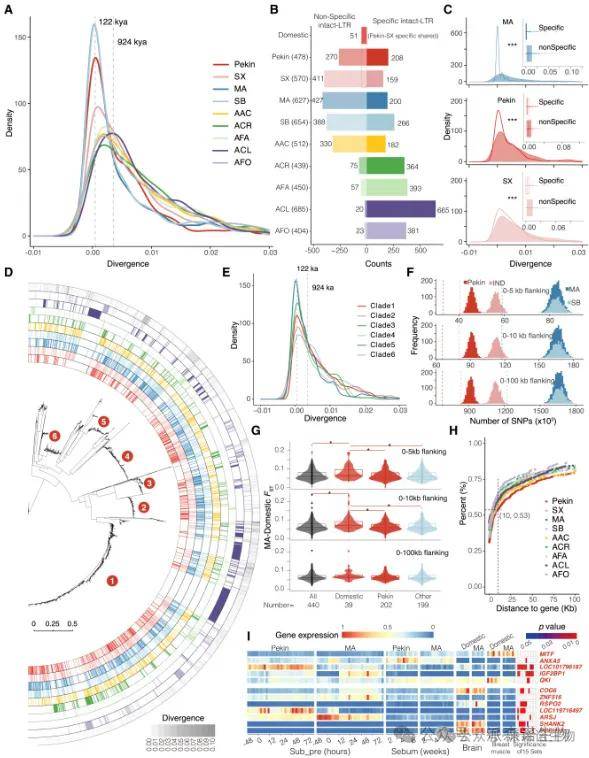
图6 常染色体Z染色体比较和驯化模型
总 结
研究团队通过分析鸭属基因组,发现Z染色体相对稳定,绿头鸭和灰鸭存在雌性偏向基因流动,驯化历史发生改变。
泛基因组揭示了家鸭和野鸭间的差异化结构变异,这些变异可能影响驯化相关基因(如GHR、FER、MITF和IGF2BP1),为动物进化研究提供了新见解。