2025-06-03
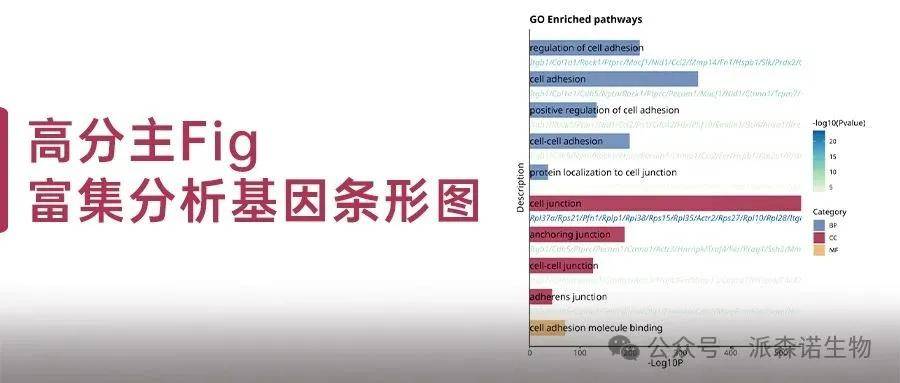
在单细胞转录组测序基因功能研究的进程中,GO\KEGG富集分析以其系统性和深度解析能力,成为科研人员挖掘基因功能奥秘的核心工具。为了更直观地呈现基因在不同通路的富集特性,我们利用如下的 GO 富集通路图为例,精妙呈现基因在各类生物通路中的分布与富集态势,为探索基因功能网络架起桥梁。
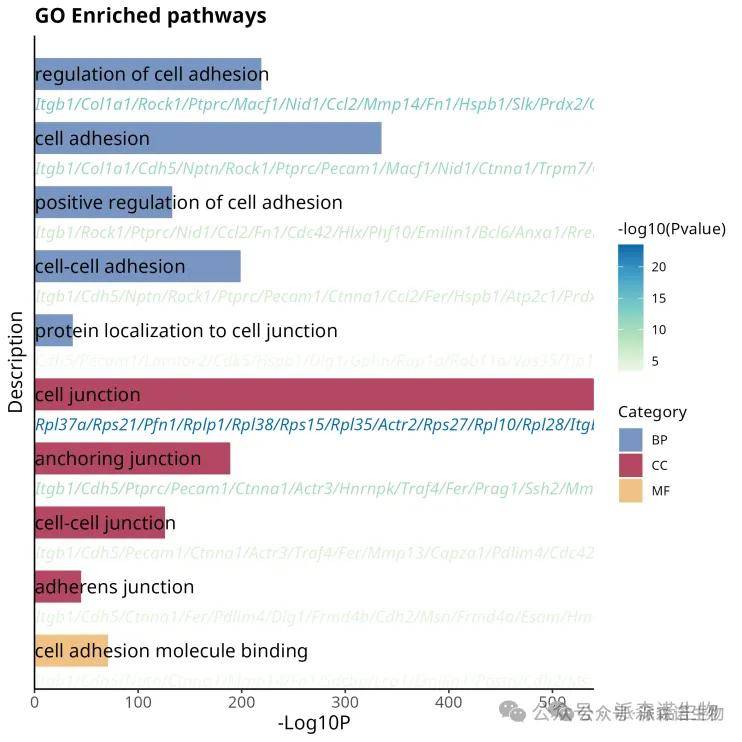
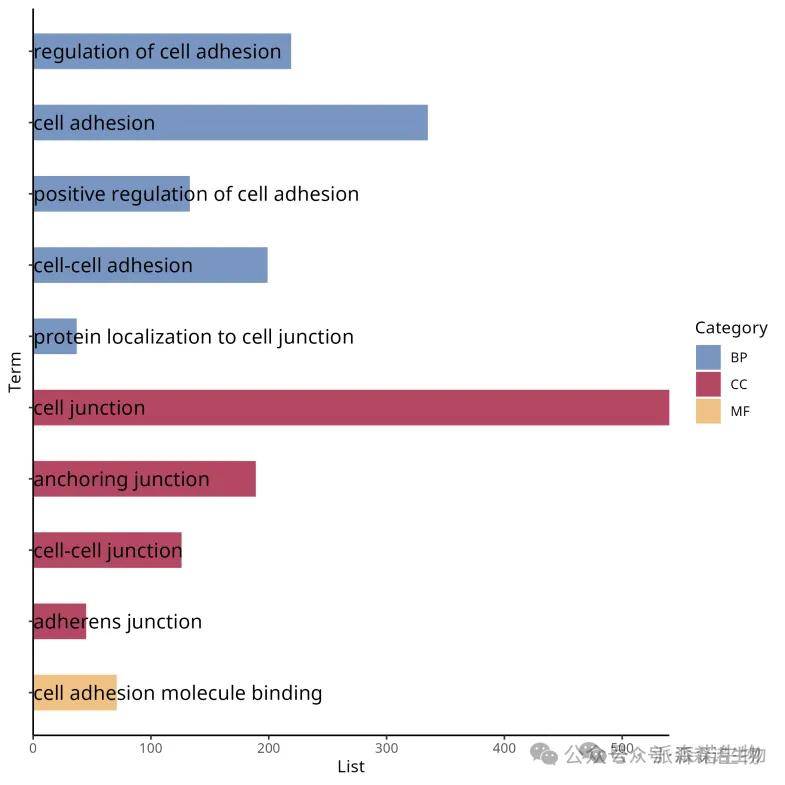
这张 “GO Enriched pathways” 图,纵轴为富集的通路名称,如 “regulation of cell adhesion”(细胞粘附调节)等;横轴 “-log10 (P)” 体现富集显著性,数值越大(条带越长),富集越显著。颜色区分 GO 三大类别:蓝色(BP,生物过程)、深红色(CC,细胞组分)、黄色(MF,分子功能)。各通路下列出相关基因,直观呈现其基因组成,助力研究者快速掌握基因在生物过程、细胞组分及分子功能层面的集中趋势,为深入探究基因功能机制提供清晰导向。下面我们就看看图形是如何一步步绘制出来的吧!
第一步骤:整理数据格式
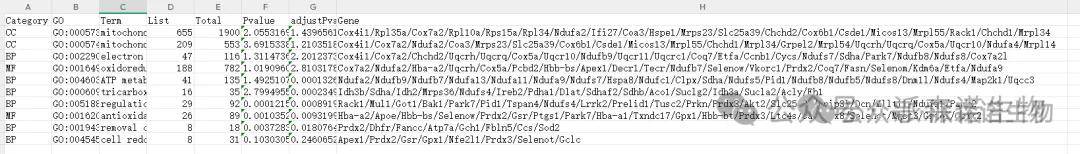
我们需要将得到的富集结果进行筛选,留下所需要的通路,同时对Category和Pvalue排序
df = data[data$GO %in% c(“GO:0051881”,”GO:0019430”,”GO:0045454”,”GO:0006099”,”GO:0022900”,”GO:0046034”,”GO:0005743”,”GO:0005739”,”GO:0016209”,” GO:0016491”
df <- df %>%
arrange(Category, desc(-log10(Pvalue)))
得到如图所示数据
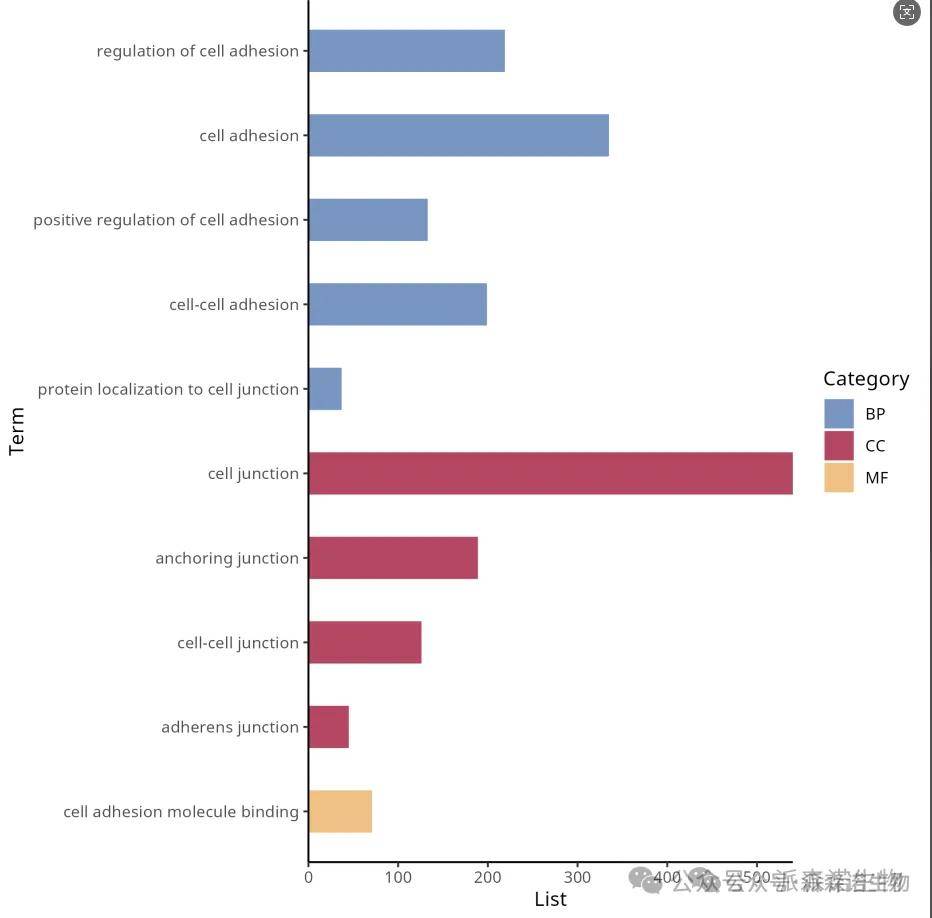
第二步骤:绘制基础条形图
p <- ggplot(df) +
geom_bar(aes(x = List, y = Term, fill = Category),
width = 0.5, stat = 'identity') +
theme_classic() +
scale_x_continuous(expand = c(0, 0))+
scale_fill_manual(values = c("#7895C1", "#B54764", "#F0C284"))
得到下图:
第三步:去掉Y轴标签,添加Term文本
p1 <- p +
geom_text(aes(x = 0.1, y = Term, label = Term),
size = 4.5, hjust = 0)
第四步骤:添加Gene标签,并用 -log10(Pvalue) 着色
p2 <- p1 +
geom_text(aes(x = 0.3,
y = Term,
label = Gene,
color = -log10(Pvalue)),
size = 4, fontface = 'italic') +
scale_color_gradientn(colors = c("#D1E9D6", "#008837", "#022C6B"))
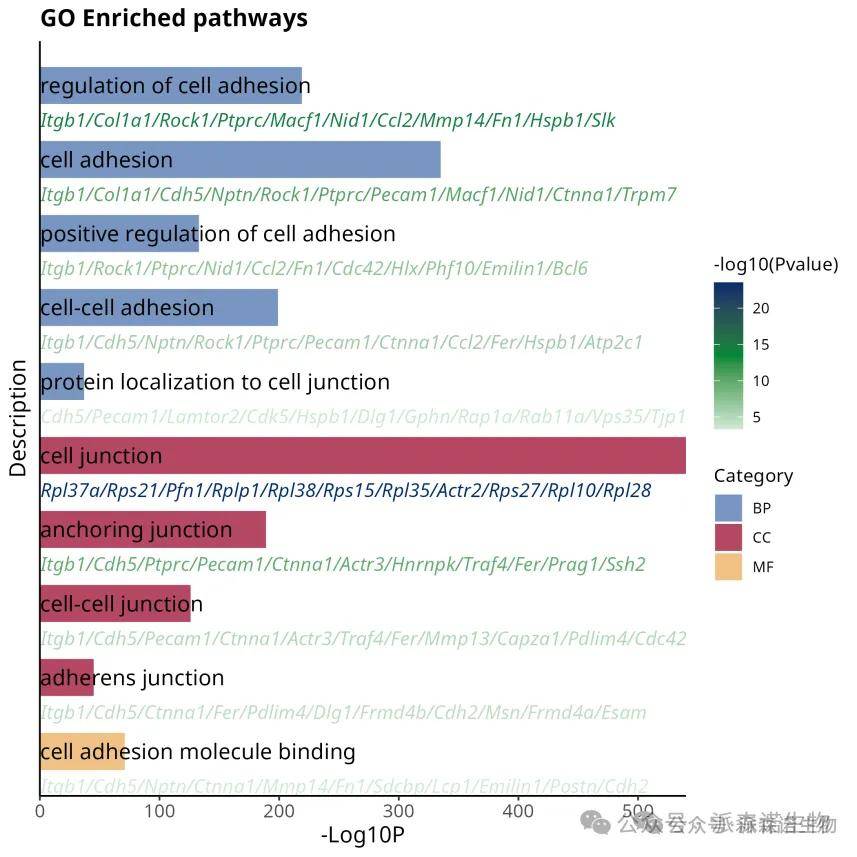
好看且内容丰富的富集图就完成啦!!
绘制图片或者复现代码过程中,如果老师遇到疑惑,欢迎拨打我们的热线电话或者联系我们的驻地销售。


