2025-06-04

SCENIC(Single-Cell Regulatory Network Inference and Clustering)是一种基于单细胞转录组数据的生物信息学分析流程,通过整合共表达网络、转录因子motif分析和调控活性评分,系统性地推断基因调控网络并鉴定驱动不同细胞状态的转录因子。
在SCENIC分析中,Connection Specificity Index (CSI) 是一种用于评估Regulon之间调控关联性的关键指标。其计算分为两个阶段:首先通过Pearson相关系数(PCC)衡量所有Regulon两两之间的表达相关性;随后针对每一对Regulon组合,统计其他相关组合中PCC值低于该组合的比例,得到0-1范围的CSI值,该值越高表明两个Regulon的关联越特异。基于CSI矩阵进行层次聚类可划分功能模块,每个模块的活性通过成员Regulon的平均活性得分表征,从而揭示不同细胞状态下协同作用的转录调控网络。高CSI值的Regulon组合往往共同调控特定生物学功能,为解析细胞状态转换的分子机制提供重要线索。
绘制CSI热图需要我们导入scenic生成的loom文件,绘制代码如下:
lf = lp.connect("pyscenic_output.loom", mode='r', validate=False )
auc_mtx = pd.DataFrame( lf.ca.RegulonsAUC, index=lf.ca.CellID)
pear_corr_reg = pd.DataFrame(np.corrcoef(auc_mtx.T))
读取后,计算CSI分数,并且导出结果表格
corr_mat= pear_corr_reg
csi_mat = np.zeros(corr_mat.shape)
for i in range(corr_mat.shape[0]):
a = corr_mat.index[i]
for j in range(corr_mat.shape[1]):
b = corr_mat.columns[j]
c = corr_mat.iloc[i][j] - 0.05
conn_pais_a = set(corr_mat.index[corr_mat.loc[i , :] >= c])
conn_pais_b = set(corr_mat.index[corr_mat.loc[: , j] >= c])
conn_pais_ab = len(conn_pais_a.union(conn_pais_b))
n = corr_mat.shape[0]
csi = 1 - (conn_pais_ab / n)
csi_mat[i,j] = csi
csi_mat_df = pd.DataFrame(data=csi_mat, index=auc_mtx.columns.values, columns= auc_mtx.columns.values)
导出的转录因子之间的CSI 表格
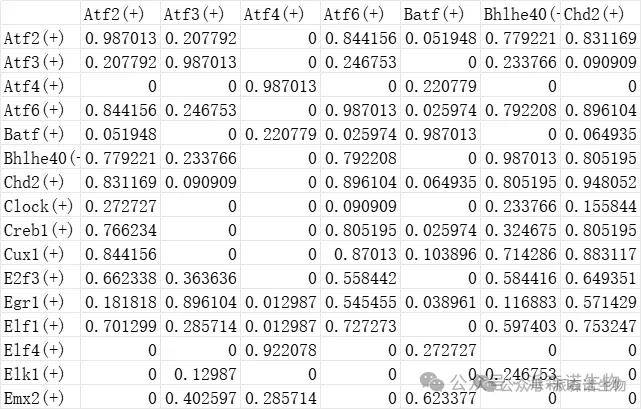 表1 CSI结果表格
表1 CSI结果表格
得到CSI数据后,就可以绘制热图了,代码如下:
g= sns.clustermap(csi_mat_df,
row_colors=row_colors.values,
figsize=(20,20),
xticklabels = [],
yticklabels = [],
cmap ="viridis",
annot_kws={"size": 3},
rasterized = True
)
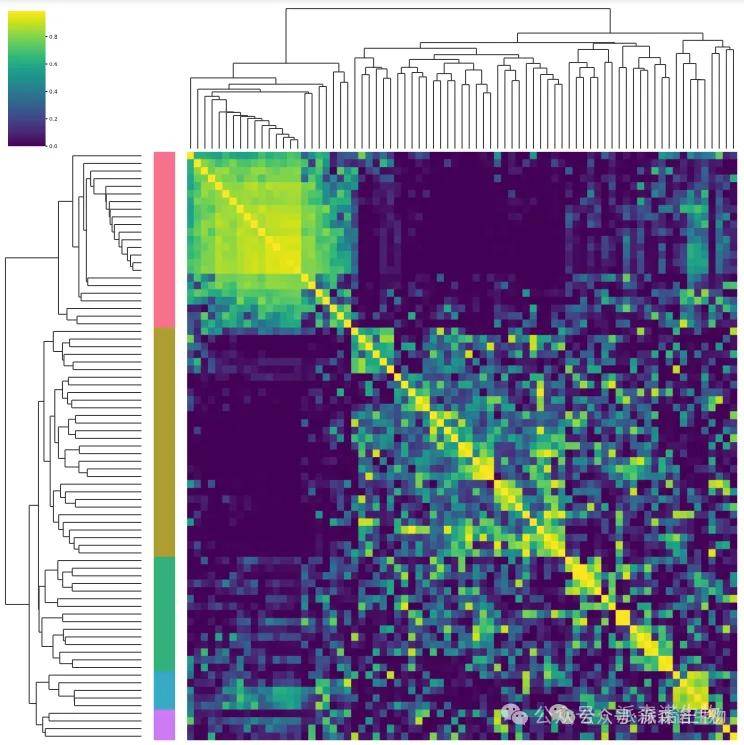 图1 CSI热图
图1 CSI热图
上图Regulon模块的CSI关联性聚类热图,行列均表示regulon,颜色由蓝变黄表示CSI关联性值由低到高。CSI值都较高的regulon可能具有相似的细胞功能,共同调控下游基因。


