
2025-06-05

文章信息
英文题目:A pangenome reference of wild and cultivated rice
中文题目:野生与栽培水稻的泛基因组参考
发表期刊:Nature
影响因子:50.5
发表时间:2025年4月
涉及组学:基因组De novo、全基因组重测序、转录组
摘要
亚洲栽培稻(Oryza sativa)的野生祖先——普通野生稻(Oryza rufipogon)是水稻育种的重要遗传资源。本研究基于145个染色体水平的基因组组装(包含129份普通野生稻材料和16个栽培稻品种),构建了一个野生-栽培稻泛基因组。该泛基因组包含3.87 Gb未出现在粳稻参考基因组(日本晴品种)中的序列,并通过捕获初级组装中缺失的杂合信息(替代组装),共鉴定出69,531个泛基因,其中28,907个为核心基因,13,728个为野生稻特有基因。研究发现,野生稻中抗病基因类似物(RGAs)的丰度和多样性均显著高于栽培稻。群体分析表明,两个栽培稻亚群(intro-indica 和basmati)的形成源于南亚栽培稻品种间的基因交流。同时,本研究为"亚洲栽培稻单次驯化"理论提供了强有力证据。此外,研究系统解析了籼-粳亚种间855,122个分化SNP和13,853个存在/缺失变异(PAVs),揭示这些变异可追溯至祖先群体的分化事件,且粳稻经历了更严重的遗传瓶颈效应。本研究成果不仅为水稻育种提供了精准的基因组参考资源,更深化了人类对水稻起源与驯化历程的认知。
前言
水稻(Oryza sativa)作为全球最重要的粮食作物之一,其野生祖先——普通野生稻(Oryza rufipogon)蕴含丰富的遗传多样性,是改良栽培稻抗逆性和产量的关键资源。尽管已有多个栽培稻参考基因组(如粳稻日本晴和籼稻93-11),但单一线性参考基因组难以全面反映物种的遗传变异,尤其无法涵盖野生稻中大量未被利用的优异等位基因。近年来,泛基因组研究成为解析物种遗传多样性的重要手段。然而,现有水稻泛基因组研究主要集中于栽培稻,对野生稻资源的系统性分析仍显不足。此外,关于亚洲栽培稻的起源(单次驯化vs.多次独立驯化)及其亚种的形成机制仍存在争议。
研究内容
1、149份水稻基因组的从头组装
研究人员对149份水稻基因组(129份野生稻和16份栽培稻)进行了高质量的从头组装,通过整合PacBio HiFi、Nanopore和Hi-C多平台测序数据,获得了染色体水平的参考基因组。评估显示这些基因组具有极高的完整度(BUSCO 98.55%)和准确性(HiFi QV 57.92),其中与T2T-NIP参考基因组相比仅存在32.7 kb错配。研究发现野生稻基因组平均大小(395 Mb)显著大于栽培稻(385 Mb),主要差异源于转座元件(TE)含量的增加(野生稻TE占比53.23% vs 栽培稻52.32%)。、基因注释显示每个基因组平均含有40,273个基因,野生稻基因密度(98.5 genes/Mb)略低于栽培稻(100.2 genes/Mb)。端粒(CCCTAAA重复)和着丝粒(CentO卫星序列)的精确定位进一步验证了组装的可靠性。
2、基因注释及抗病基因类似物(RGAs)的特征解析
研究人员通过整合同源预测、转录组数据和从头预测方法,对149个水稻基因组进行了系统注释,平均每个基因组鉴定出40,273个基因(平均长度2,566 bp),其中92%的基因含有功能域,72%至少在一种组织中表达。重点对抗病基因类似物(RGAs)进行了系统分析,基于保守结构域(如NBS、RLK、TM-CC和RLP)共预测出野生稻平均1,710个RGAs,显著高于栽培稻(平均1,652个)。研究发现野生稻RGAs不仅数量更多(P<0.01),其多样性也更为丰富,其中,
结构变异:野生稻中27.55%(638个)RGA位点为特有,且多形成串联重复簇;功能分化:关键抗病基因在野生稻中保留更多功能等位基因,其抗稻瘟病单倍型(如Y476)在野生群体中高频存在;进化动态:RGAs在野生稻中呈现更年轻的扩张特征,与近期环境适应相关。
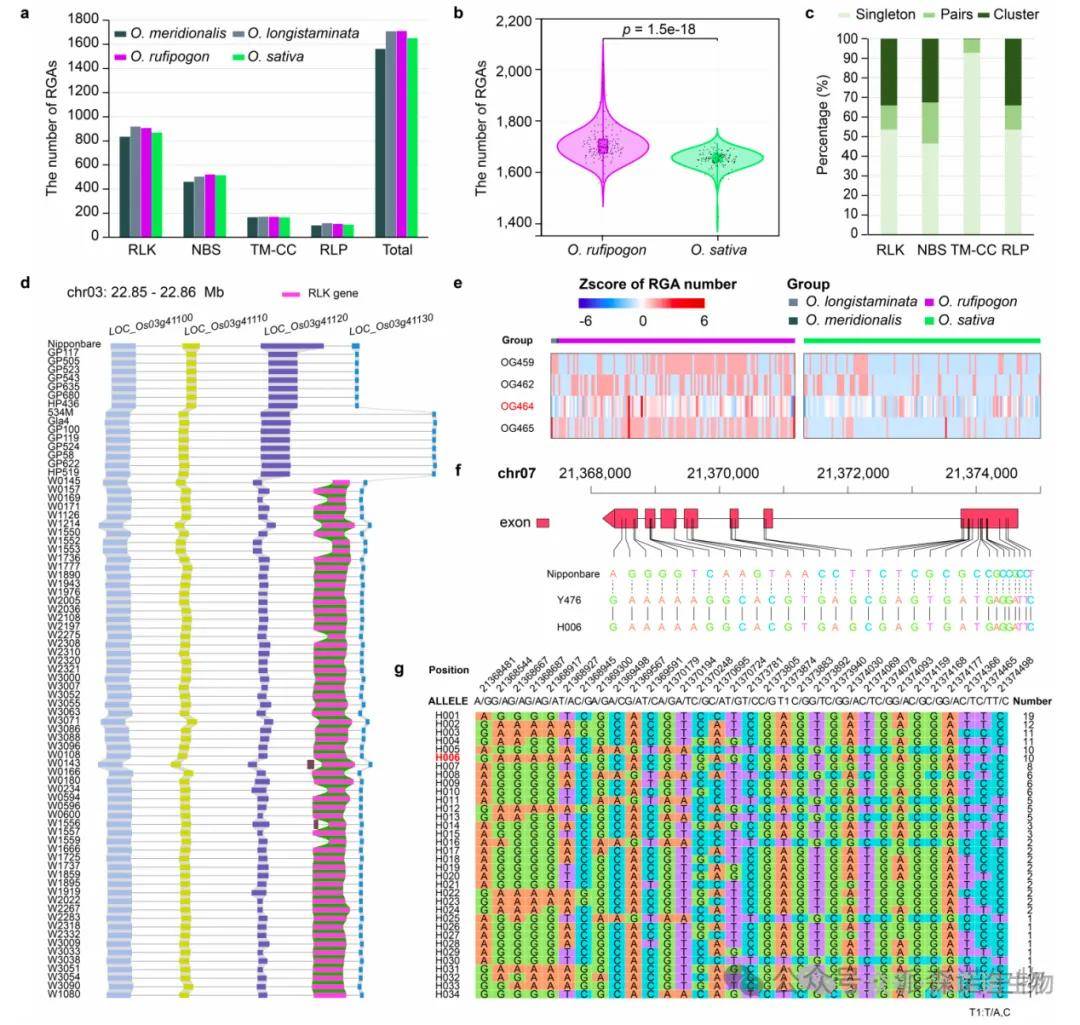 图1 野生和栽培水稻的RGAs特征分析
图1 野生和栽培水稻的RGAs特征分析
3、基于基因和基于图的泛基因组构建
研究人员通过整合145个水稻基因组(129野生稻+16栽培稻)构建了双轨泛基因组:基于基因的泛基因组鉴定出69,531个泛基因(含28,907个核心基因和13,728个野生稻特有基因),揭示野生稻特有基因显著富集于抗病等适应性功能;基于图结构的泛基因组包含3.87Gb非参考序列,解析了855,122个籼-粳稻分化SNP和13,853个PAVs,其变异检测灵敏度较线性参考基因组提升23%。
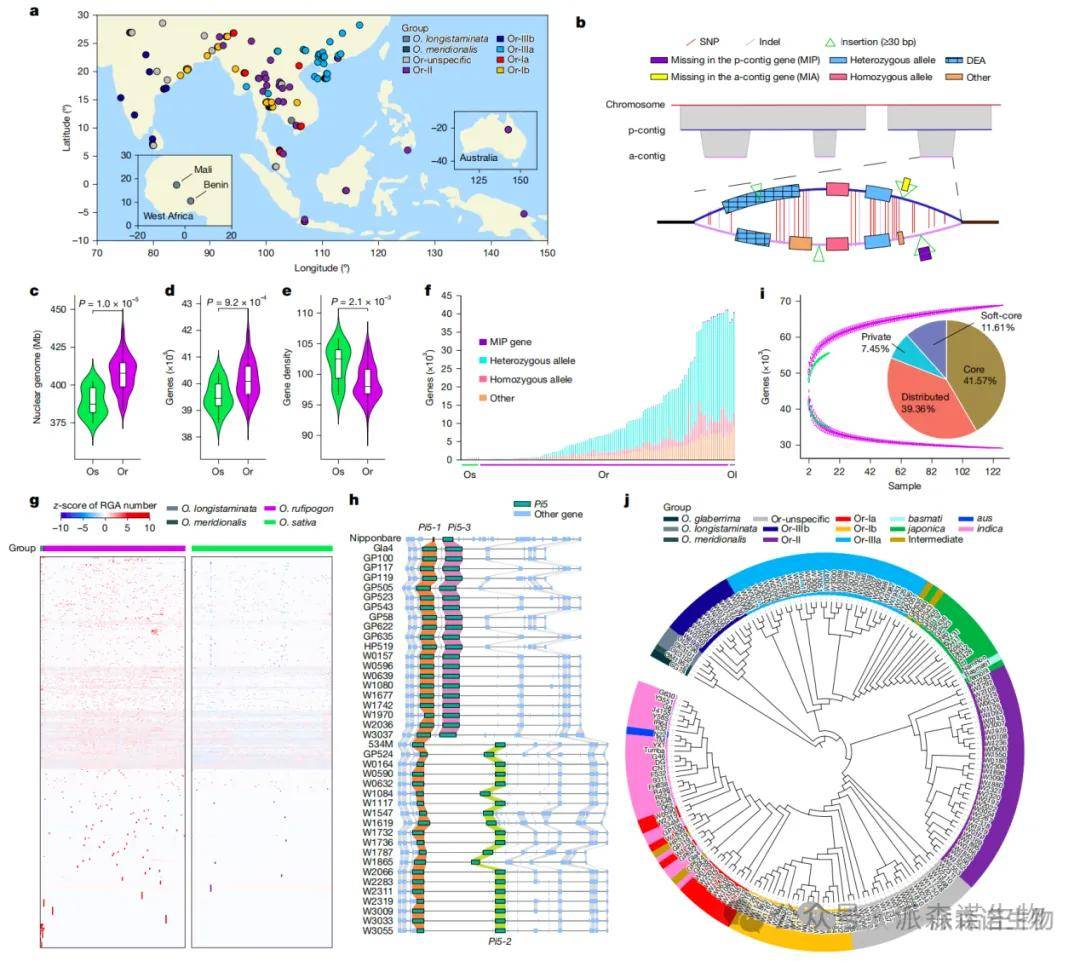
图2 野生和栽培水稻泛基因组的组装和分析
4、全基因组变异和转座元件分析
研究人员进行了全面的变异和转座元件(TE)分析,共鉴定出11.08百万SNP、10.97百万小片段插入缺失(indel)以及362,194个缺失、531,356个插入、4,495个倒位和357,655个易位等结构变异(SV)。研究发现野生稻的转座元件(TE)含量(53.23%)显著高于栽培稻(52.32%),其中Gypsy类LTR反转录转座子在野生稻Or-IIIa群体中表现出明显的近期扩张(约2.5万年前),贡献了57.78%的基因组大小差异。通过比较17个关键Gypsy家族,发现野生稻中9 Mb的差异源于新近扩增,而8 Mb差异由非法重组导致的TE丢失造成。这些变异在基因侧翼区域显著富集于碳水化合物代谢(P=3.2e-5)和细胞表面信号通路(P=1.8e-4),揭示了TE驱动的水稻环境适应性演化机制。
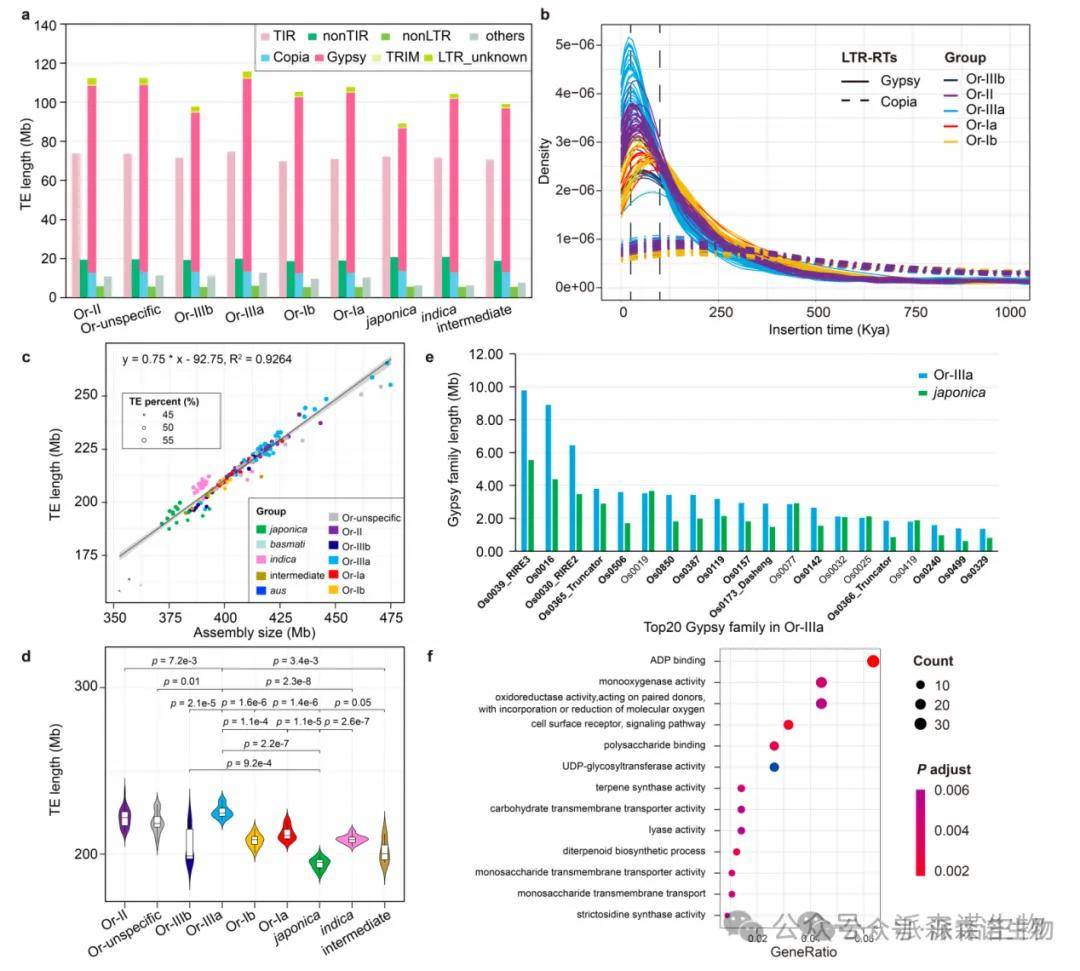
图3 野生和栽培水稻的TE分析
5、亚洲栽培水稻的进化分析
研究人员通过整合280份水稻种质(包括野生稻和栽培稻)的全基因组数据,系统解析了亚洲栽培稻的演化历程。研究证实所有亚洲栽培稻均单次起源于中国南部的野生稻Or-IIIa群体(粳稻祖先),而籼稻则是由早期粳稻向东南亚扩散后与当地野生稻Or-I群体杂交形成。群体遗传分析发现,南亚地区的栽培稻(如Basmati和Aus)存在复杂的基因交流:Basmati由粳稻与渗入籼稻(intro-indica)杂交形成,而渗入籼稻本身是籼稻与Aus稻的杂交后代。选择性清除分析显示,栽培稻中55.3%的驯化区域遗传组分来自Or-IIIa,关键驯化基因(如sh4、PROG1等)的等位变异模式进一步支持了"单次驯化-多次渐渗"的演化模型。
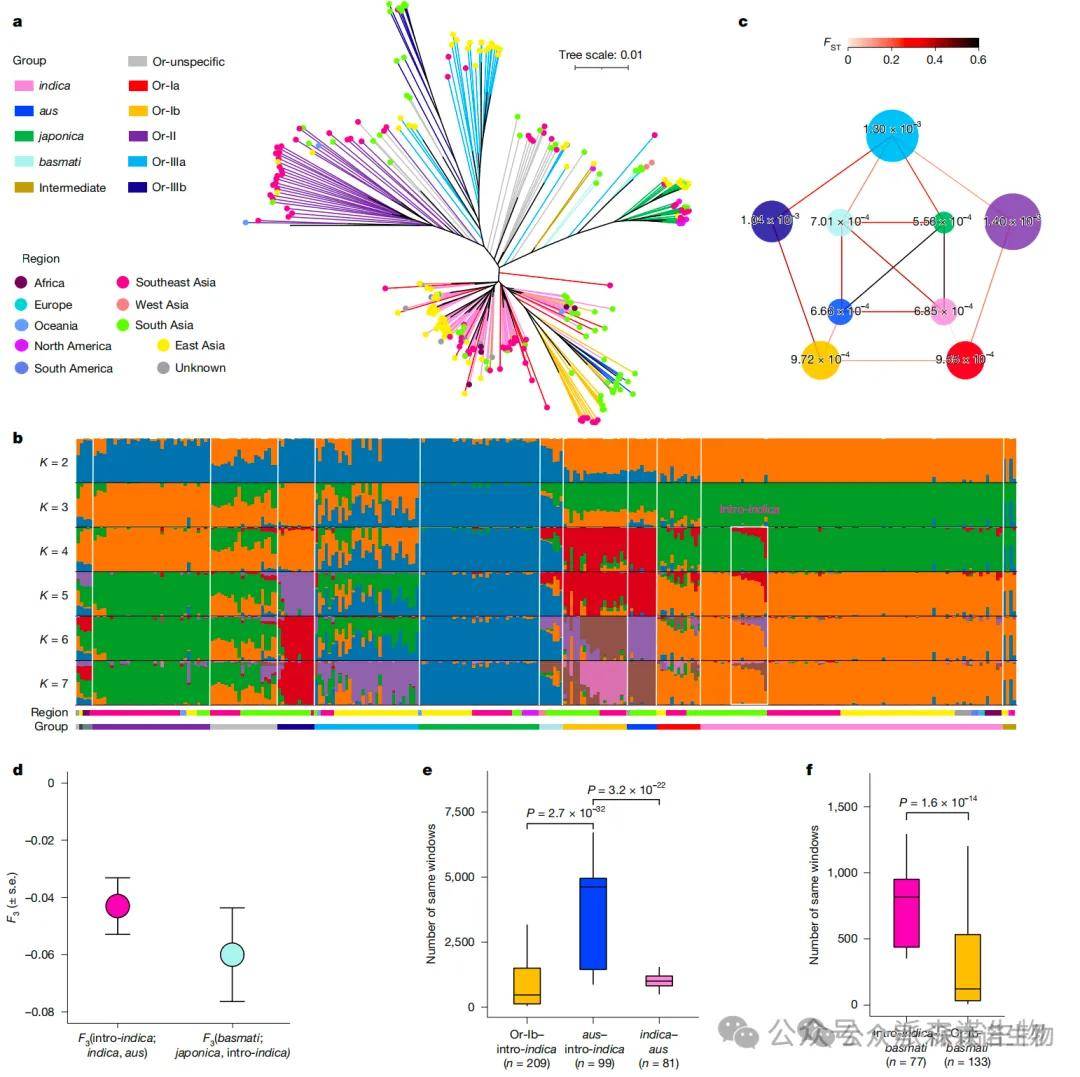
图4 野生和栽培水稻的群体进化分析
6、亚洲栽培水稻的驯化分析
研究人员通过全基因组分析揭示了亚洲栽培稻的驯化历程,证实所有栽培稻均起源于中国南部的野生稻Or-IIIa群体(粳稻祖先),支持"单次驯化"理论。研究发现籼稻的形成经历了"粳稻南迁-与当地野生稻Or-I群体杂交"的二次驯化过程,而南亚特有的Basmati和Aus稻则是栽培稻间多次基因交流的产物。选择性清除分析鉴定出50个强选择信号区域(共12.35Mb),其中55.3%的驯化区域遗传组分来自Or-IIIa群体。11个关键驯化基因(如控制落粒性的sh4、调控株型的PROG1等)的等位基因溯源显示,栽培稻中这些基因均与Or-IIIa的单倍型直接相关,而籼稻和Aus稻中部分基因通过后续基因流获得。
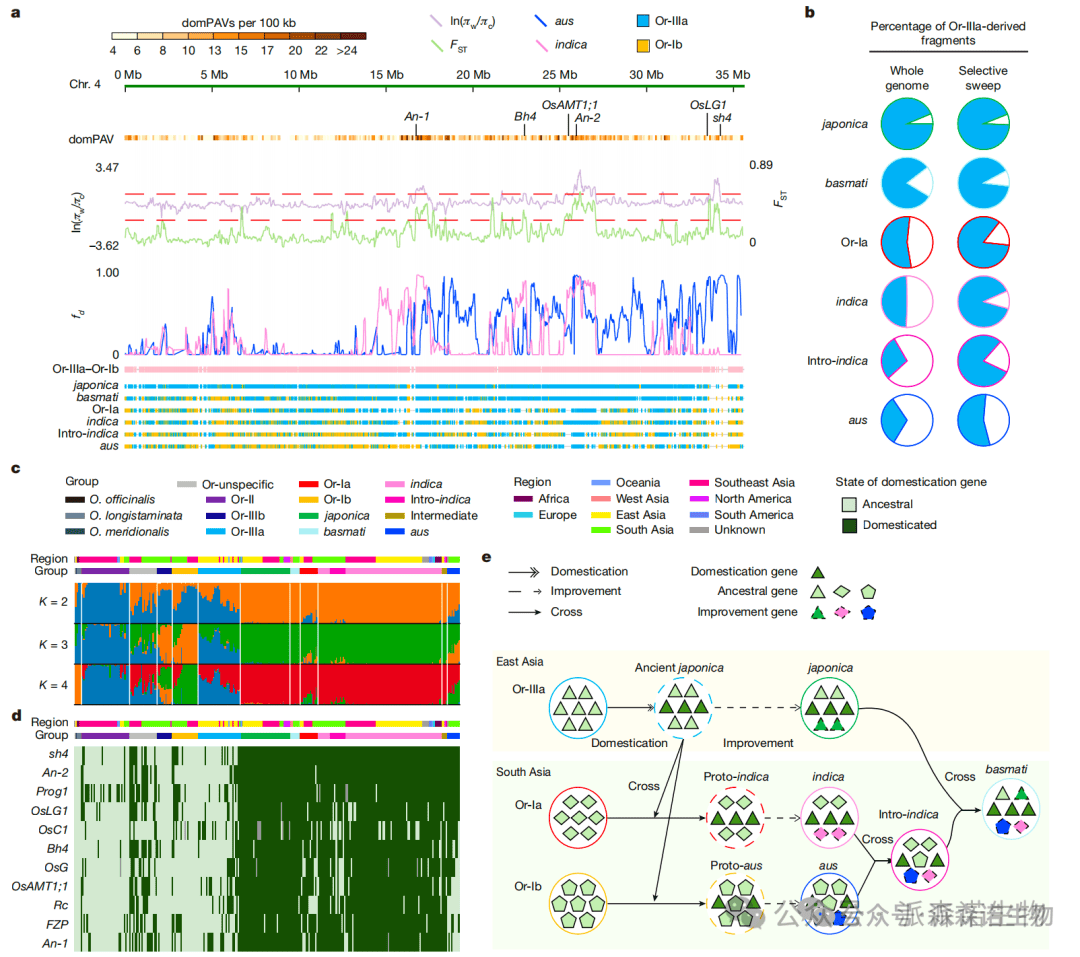
图5 野生和栽培水稻的选择性清除分析
总结
1.整合145个高质量水稻基因组,构建了首个野生-栽培稻泛基因组图谱,鉴定出69,531个泛基因,包括28,907个核心基因和13,728个野生稻特有基因。
2. 证实亚洲栽培稻为单次驯化起源,所有栽培稻均来自中国南部的野生稻Or-IIIa群体(粳稻祖先)。籼稻由早期粳稻南迁后与当地野生稻Or-I群体杂交形成,而Basmati和Aus稻则是栽培稻间基因交流的产物。
3. 野生稻基因组大小(平均395 Mb)大于栽培稻(385 Mb),主要差异由转座元件(TEs)驱动,尤其是Gypsy类元件的近期扩张。