2025-06-09

目录
一、云平台重要文件下载
二、云平台结果调整
三、筛选基因分析绘图
四、常见问题解答
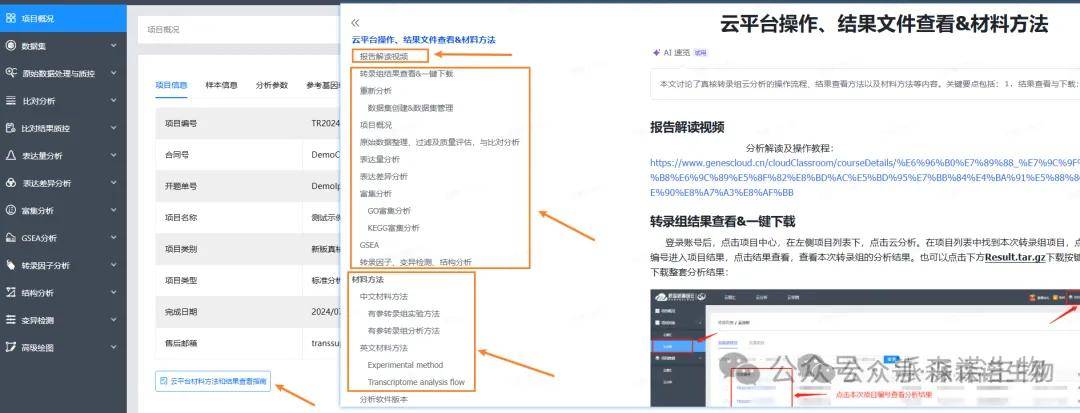
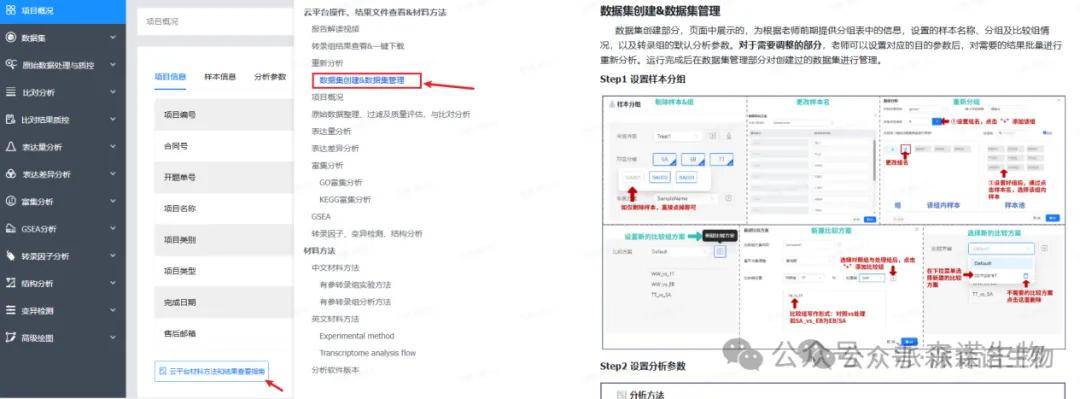
老师们做完转录组(真核有参、原核)后,对分析结果不清楚的话,可以直接在项目概况里点击云平台材料方法和结果查看指南,里面有报告解读视频、结果查看介绍、材料方法,方便老师们对分析结果的理解掌握。
一、云平台重要文件下载
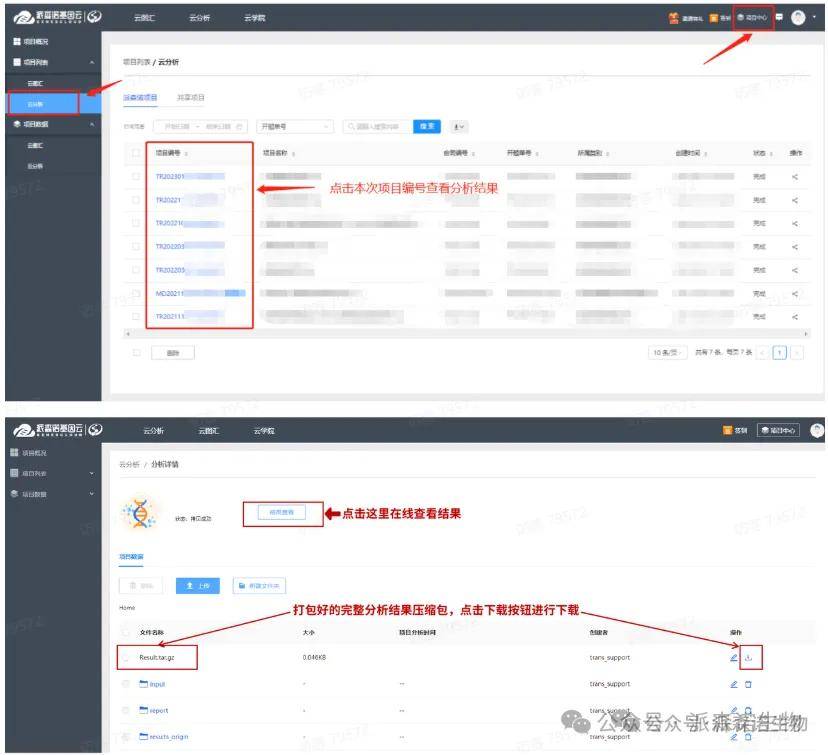
1、所有标准分析结果的下载:登录账号后,点击项目中心,在左侧项目列表下,点击云分析;在项目列表中找到本次转录组项目,点击项目编号进入项目结果,点击下方Result.tar.gz下载按键,打包下载整套分析结果。
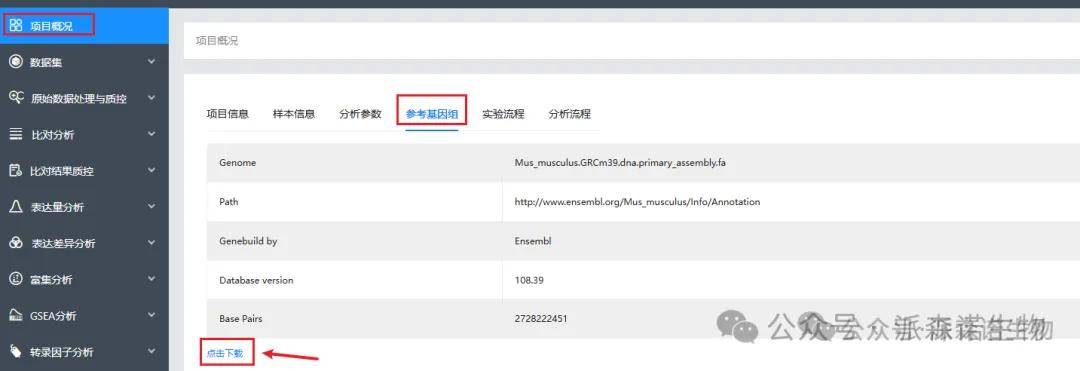
2、基因注释表下载(Annotation.xls,包括基因ID、染色体、起始终止位置、正负链、外显子长度、基因name、各个数据库的注释):项目概况→参考基因组→点击下载。
3、表达量结果下载(Expression_with_annotation.xls,包括表达量和注释):表达量分析→表达量分析→表→下载表格文件。
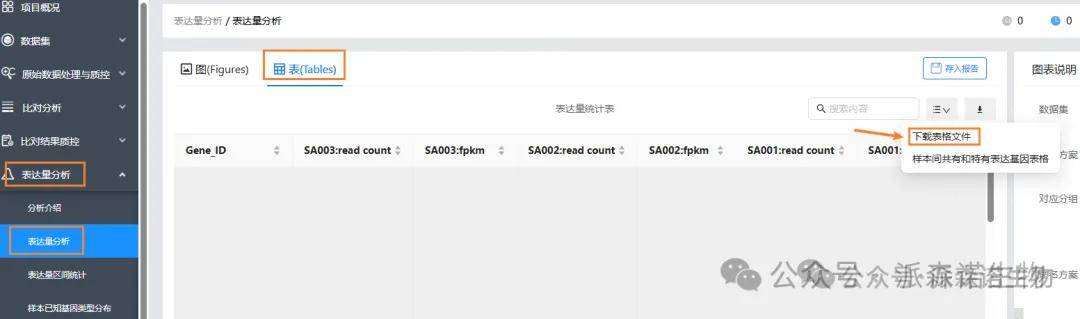
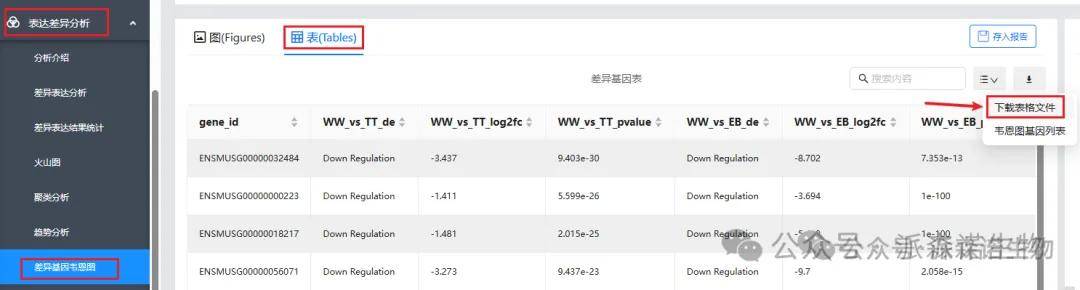
4、差异分析结果下载(上下调基因详细信息,XX_vs_XX.DEseq2.xls,包括差异和注释):表达差异分析→差异表达结果统计→表→DEG_results.tar.gz。
下载的文件为gz格式压缩包,里面有各个比较组的差异总表。如果解压软件打开不了gz压缩包的话,可以下载安装bandizip(https://www.bandisoft.com/bandizip/)解压软件。
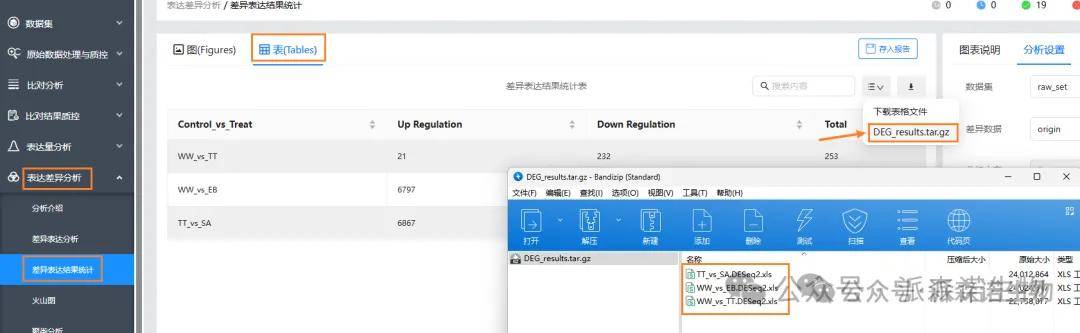
5、差异基因在各组中的情况(diff_gene_of_all_groups.xls,包括基因ID和不同比较组的up/down、log2FC、p值;需要加上注释信息可以用excel的vlookup函数在Annotation.xls里查找添加):表达差异分析→差异基因韦恩图→表→下载表格文件。
Vlookup 函数应用方法:干货 | EXCEL小技巧助力转录组数据挖掘
6、GO富集总表(**_vs_**_GO_download_enrichment.xls):富集分析→GO富集分析→表→下载表格文件。
下载的文件为Download_table.tar.gz压缩包,里面是各个比较组的富集总表。
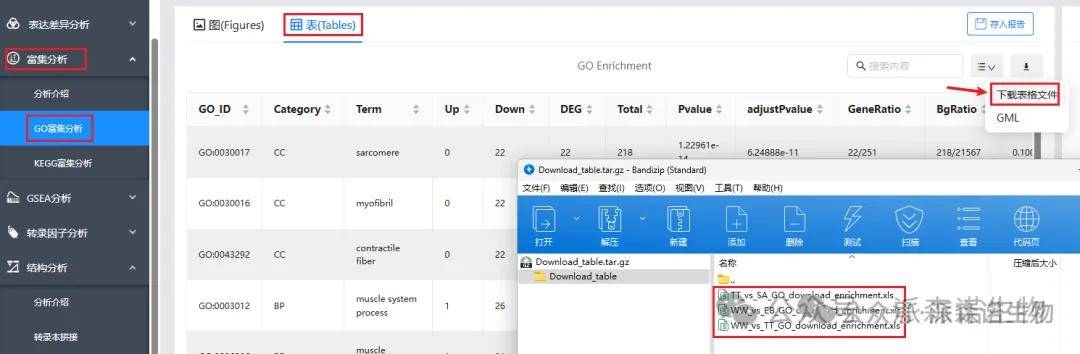
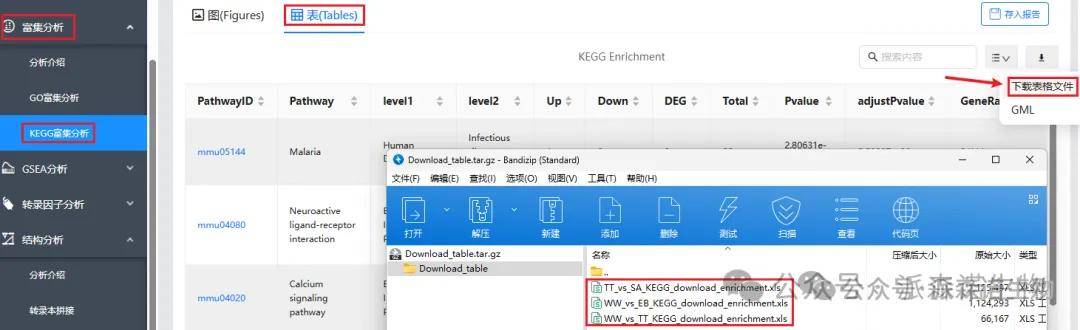
7、KEGG富集总表(**_vs_**_KEGG_download_enrichment.xls):富集分析→KEGG富集分析→表→下载表格文件。
下载的文件为Download_table.tar.gz压缩包,里面是各个比较组的富集总表。
二、云平台结果调整
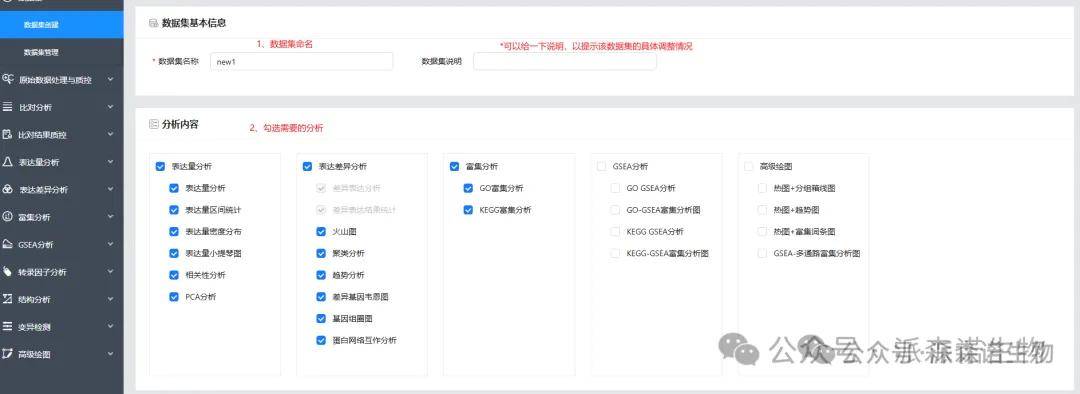
需要对分析结果进行调整的话,老师可以数据集里批量设置。数据集的使用也可以直接在项目概况里点击云平台材料方法和结果查看指南,查看里面的数据集创建&数据集管理部分。
1、剔除离群样本:
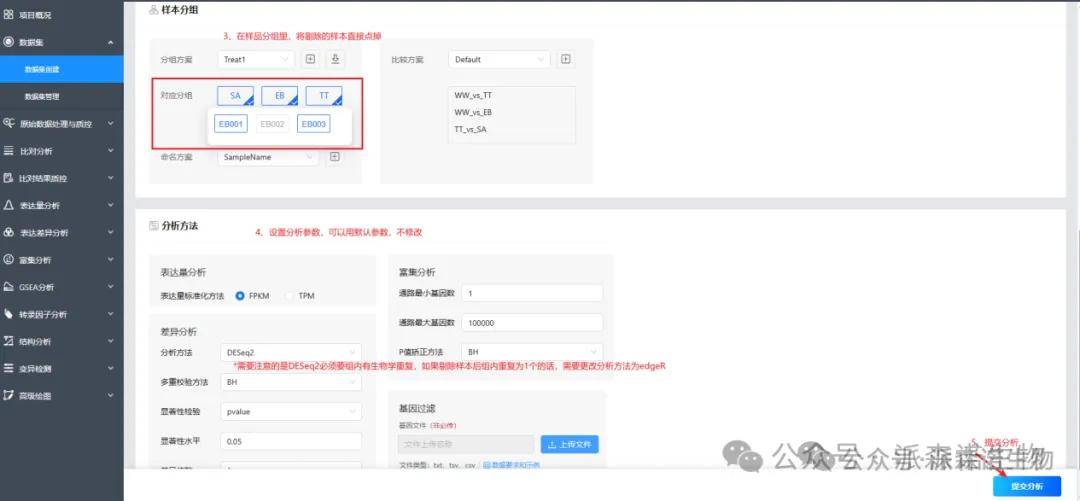
2、调整差异筛选标准:

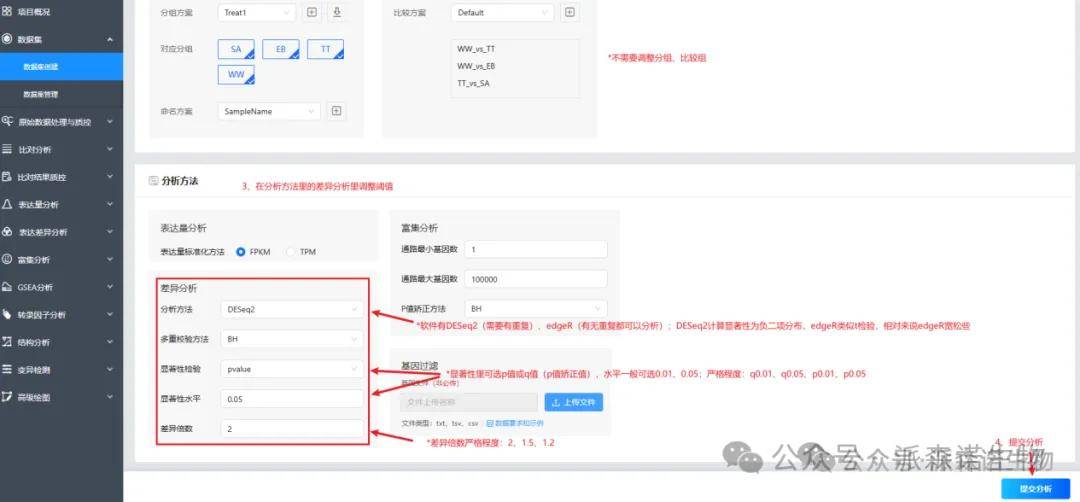
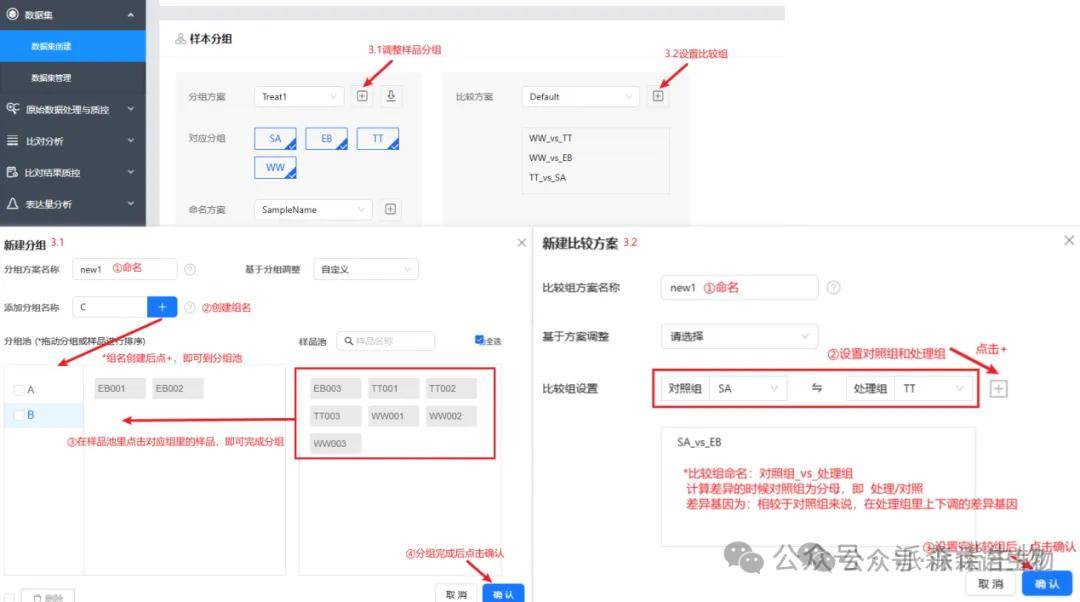
3、调整分组、比较组:

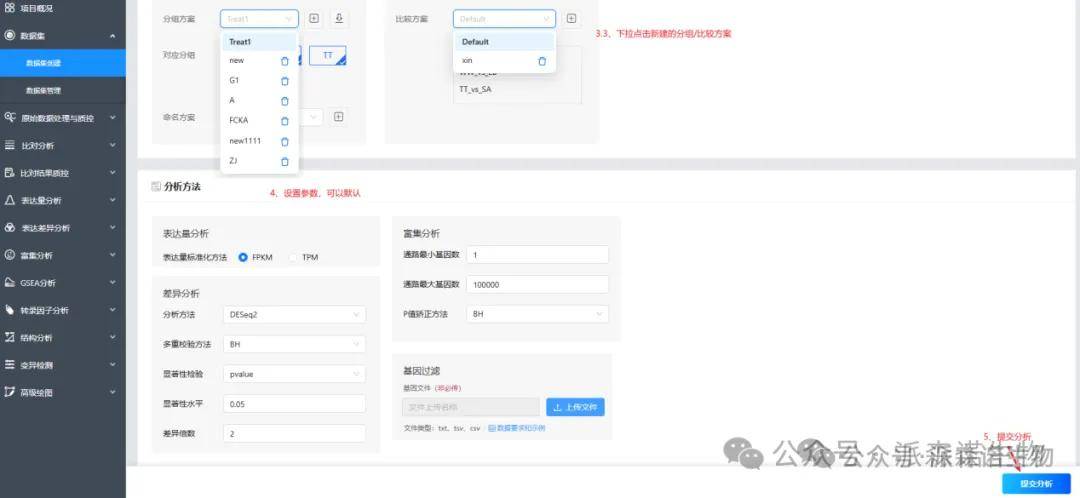
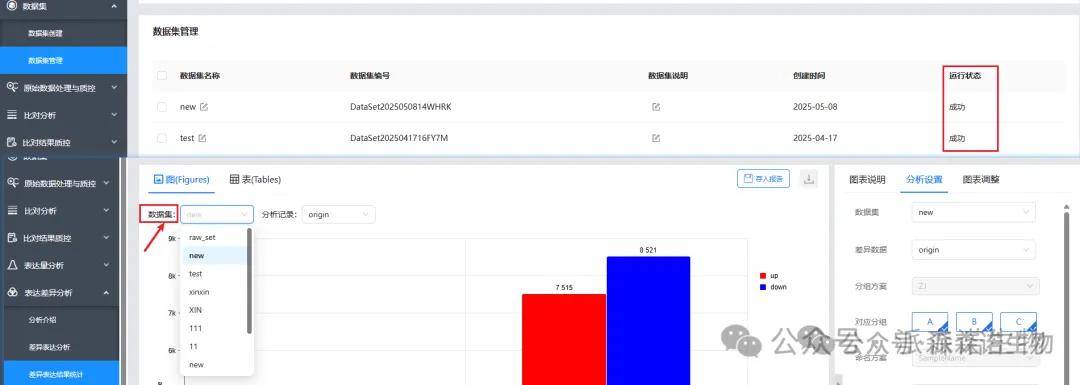
分析完成后(数据集管理里可以看是否运行完成),在各部分(2里勾选的分析)的 数据集 里选择新命名的数据集就可以展示新的分析结果了。
三、筛选基因分析绘图
针对部分筛选出来的目标基因(可以通过venn图筛选多个比较组共有、特有差异基因;可以根据富集结果筛选关注通路上的差异基因等),老师可以通过数据集里上传基因列表的方式去分析。
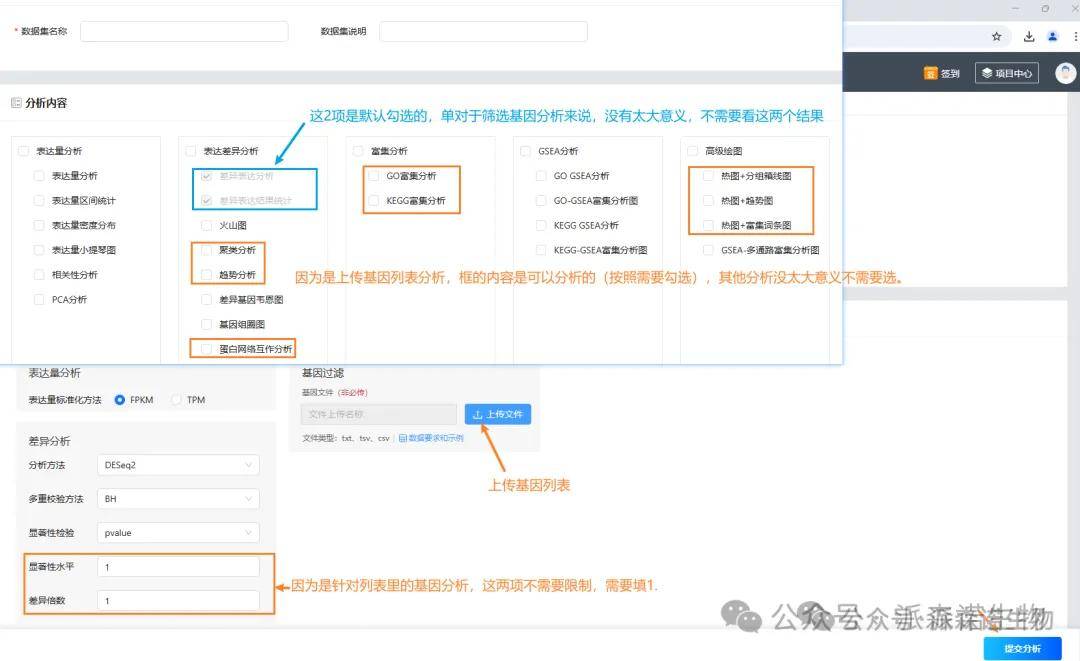
四、常见问题解答
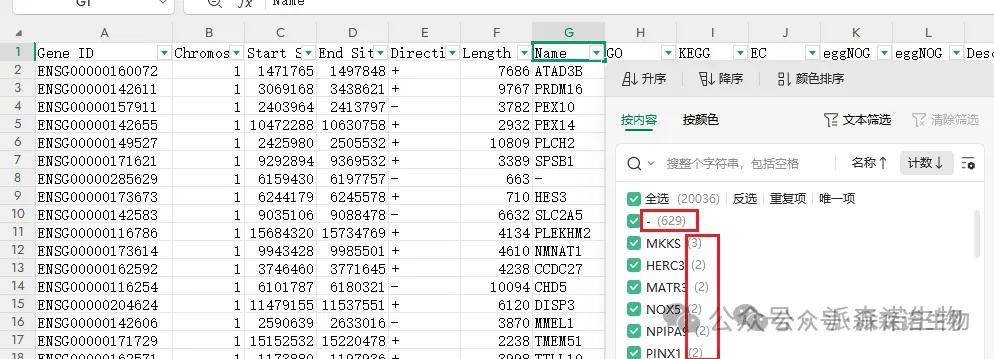
1、为什么分析不能直接用基因name去分析呢?
答:因为基因name并不是所有基因都有的,有些基因是没有name注释的,有些基因的name有重名的情况(以下图ensembl数据库里人的基因组注释为参考),因此分析都是用唯一且都有的基因ID进行的。我们在分析结果的注释、表达、差异表中都加上了基因ID和基因组里注释的Name的对应关系,可以直接在这3个表里查看。
2、表达量表格中的read count和FPKM有什么区别,需要看哪个呢?
答:read count是比对到基因上Reads的数量,作为基因的原始表达量;FPKM是基于测序深度和基因长度对read count标准化后的表达量结果。一般描述基因表达量(如绘制热图)用FPKM;read count主要是用于差异分析(因为差异软件自己也会进行标准化,所以不能用FPKM做差异分析)。
3、组内重复性从哪里来看呢?
答:在我们分析中PCA、相关性分析、聚类热图这三者都是一定程度反映样品重复性。其中PCA是通过线性变换,将高维数据降低至二维或三维,同时保持各方差贡献最大的特征(即降低数据复杂度),进而进行的主成分分析;相关性分析是基于样品里所有基因的表达量去计算样品之间的皮尔逊相关系数;聚类热图是基于所有比较组差异基因并集在样品里的表达量去绘制的双向聚类热图。三者针对的数据源不一样,一般情况下可以选择其中一者在文章中展示。
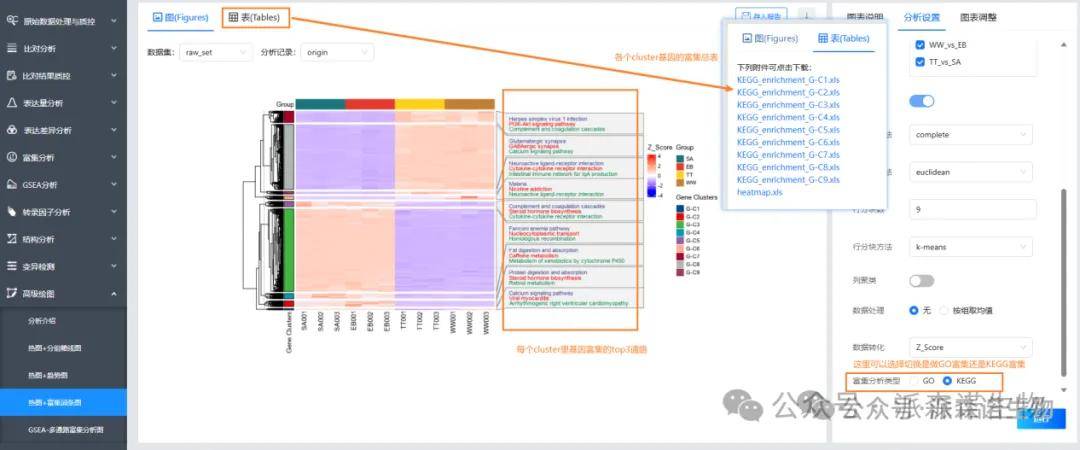
4、聚类分析里分的9类是什么意思,这9类有什么功能呢?
答:是基于差异基因并集在样品里的表达量情况,划分为9个cluster,每个cluster里的基因表达模式相近。也可以在分析设置里自定义需要划分的cluster数量,去重新运行。需要对cluster里的基因做功能分析的话,可以到高级分析的热图+富集词条图里去查看。
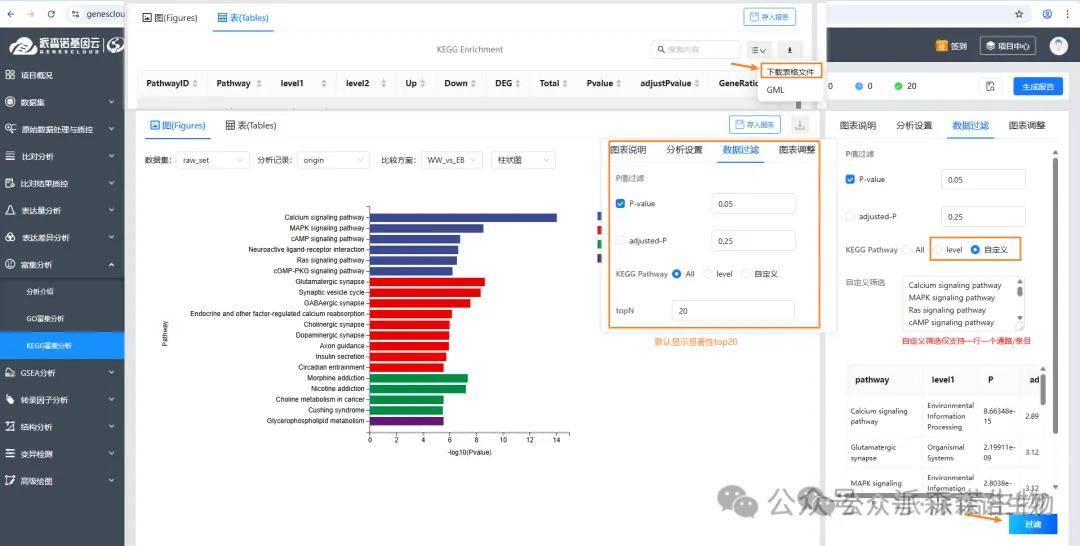
5、为啥富集图里的功能和我关注的不一样?
答:富集分析默认是展示显著性top20 GO term或KEGG通路的,但是本身来说可能富集到的通路会更多的,老师可以下载富集表格,在表格里筛选下关注的通路,看对应的富集情况。然后可以在数据过滤里针对关注的通路,去自定义绘图。
6、富集分析和GSEA分析有什么区别呢?
答:富集分析是针对比较组里的差异基因,去分析这些差异基因在GO term或KEGG pathway里的富集显著性;GSEA(基因集富集)分析是一种计算方法,不需要指定明确的差异基因阈值,把所有基因按照在两组样本中的差异表达程度进行排序,然后采用统计学方法检验预先设定的基因集合是否在排序表的顶端或低段富集。
富集分析侧重的是差异基因在通路的富集显著性;GSEA侧重这条通路整体是被激活还是抑制的。两者可以结合着使用,也可以只展示其中一者结果。
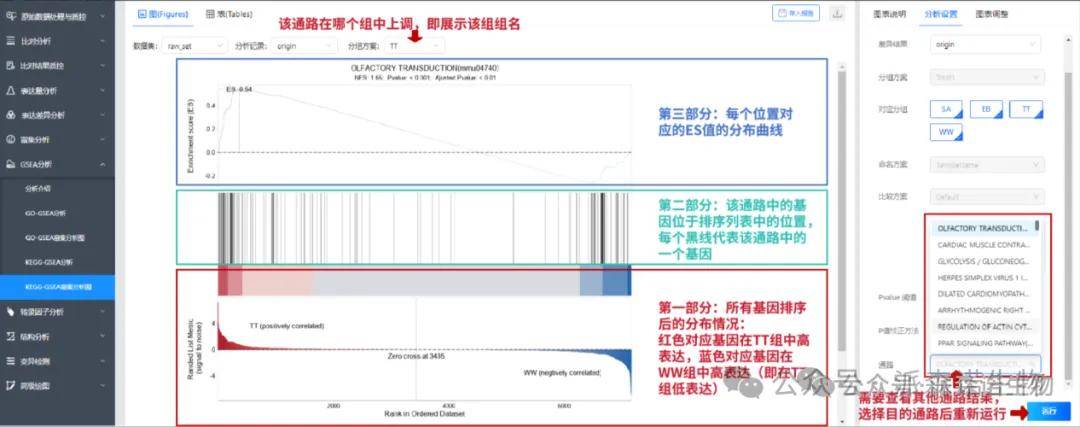
7、GSEA结果怎么看呢?
答:GSEA主要看富集图:第一部分是通路里所有基因对应的ES值的分布曲线,最高峰处的得分(垂直距离0.0最远)便是基因集的ES值;第二部分是用线条标记了对应通路中基因出现在排序列表中的位置,每条竖线代表该通路的一个基因;第三部分是所有基因排序后分布情况,其中红色部分对应的基因在处理组中高表达,蓝色部分对应的基因在对照组中高表达。一般认为|NES|>1,NOM p-val<0.05,FDR q-val<0.25的通路是显著富集的。其中NES正值代表这个通路在这个比较组中是激活的,NES负值代表这个通路在比较组中是被抑制的。