2025-06-11
在生命科学微观研究中,单细胞转录组测序(scRNA - seq)作为解析细胞功能与异质性的核心技术,其高维数据的分析常常借助降维可视化方法来帮助理解和解释数据。UMAP 算法因其在保留数据全局结构和局部结构方面的良好性能,成为单细胞图谱研究的常用方法。然而,传统二维 UMAP 图在展现复杂细胞谱系关系(如肿瘤微环境中)时存在一定局限,由于二维平面无法完全呈现高维空间中细胞的复杂关系,易因邻近亚群的重叠而掩盖细胞间潜在关联,影响对细胞异质性的解析。而三维动态 UMAP 图在一定程度上解决了这一问题,它通过引入第三维度更好地呈现细胞亚群的分布关系,并且借助计算机软件或工具的动态交互功能,如实时旋转、缩放与切面观察等,使研究者能够从多个角度捕捉二维视角下易被忽略的差异,为单细胞数据研究提供了全新的立体洞察视角。
从代码层面,我们需要从3维角度,对整个细胞进行umap降维聚类。不用担心很复杂,RunUMAP有个关键的参数n.components可以设置。代码如下:
data.combined <- RunUMAP(sc, reduction = "pca", dims = 1:20,n.components = 3L)
Embeed = data.combined@reductions$umap@cell.embeddings
data.combined = AddMetaData(Embeed ,data.combined)
运行完可以通过导出三维坐标进行绘图,绘图的表格有四列,其中前三列为细胞的三维坐标,第四列为细胞的标签,可以为细胞类型 、样本、组别和clusters等任一细胞标签,绘图代码如下:
plot.umap <- FetchData(object = data.combined, vars = c("UMAP_1", "UMAP_2", "UMAP_3",”celltype”))
plot.data <- as.data.frame(plot.umap)
head(plot.data)
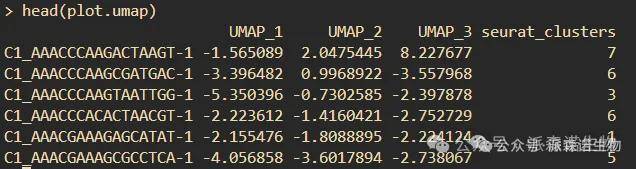
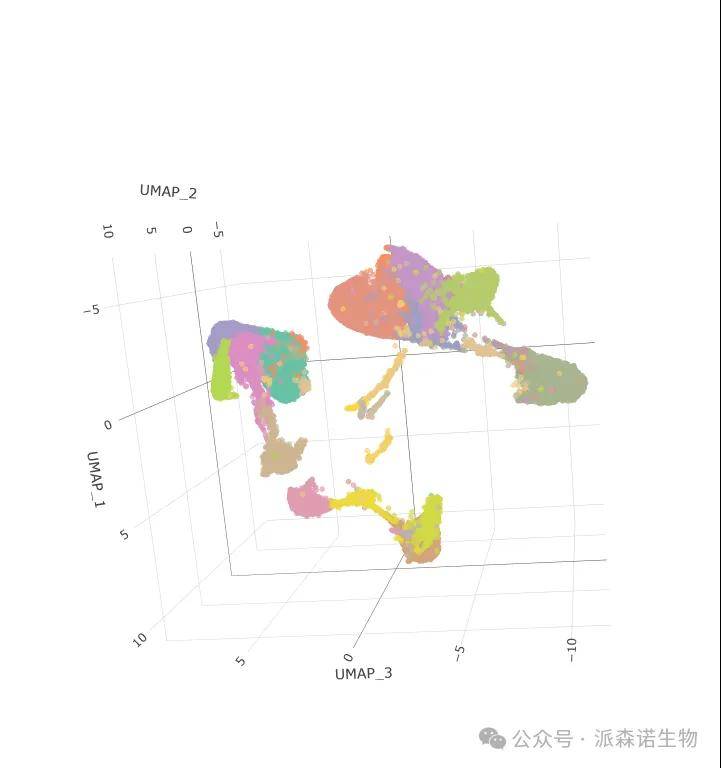
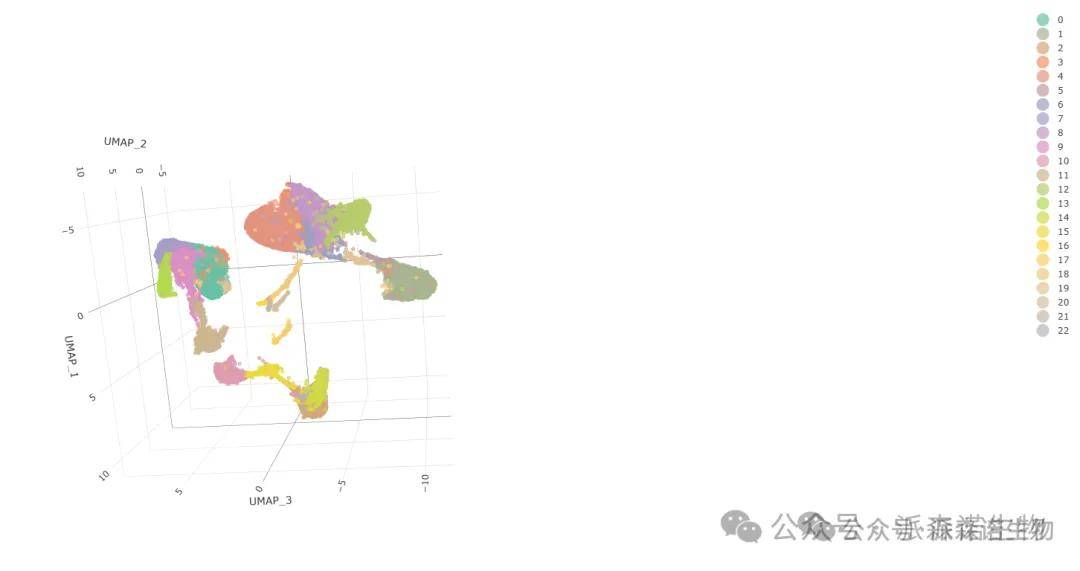
fig <- plot_ly(plot.data, x = ~UMAP_1, y = ~UMAP_2, z = ~UMAP_3, color =~seurat_clusters,size=2,colors= cb_palette.use)
saveWidget(as_widget(fig), paste0("3Dumap.html"))
总 结
本文介绍单细胞转录组数据分析中三维动态 UMAP 图复现方法。其通过第三维度和动态交互功能,解决二维 UMAP 图亚群重叠问题。代码上用 RunUMAP 设 n.components=3L 降维,借 plotly 生成交互式图,为单细胞数据图谱解析提供多维视角。绘制图片或者复现代码过程中,如果老师遇到疑惑,欢迎拨打我们的热线电话或者联系我们的驻地销售。


