2025-06-16
Highlights
1.群体遗传结构与系统进化分析为GWAS提供基础框架,通过主成分分析(PCA)、系统进化树和群体遗传结构解析样本间的亲缘关系与群体分层。
2.基于基因组关系矩阵(GRM)校正个体间亲缘关系,结合连锁不平衡衰减(LD decay)和LD Block联合分析,精准定位变异并区分信号,从而提升关联结果的生物学可解释性。
3.派森诺提供全基因组关联分析(GWAS)一站式解决方案:从样本检测到基因定位,专业解析复杂性状遗传机制,助力精准育种与医学研究突破。
全基因组关联分析(GWAS)是一种通过高通量测序或芯片技术检测动植物群体中的遗传变异(如SNP),并结合表型数据,挖掘与重要农艺性状或适应性特征显著关联的基因位点的方法。在动植物研究中,GWAS通常利用自然群体或人工选育群体,分析基因型与表型(如产量、抗病性、生长速度等)的统计学关联,从而定位关键候选基因或调控区域。
常见GWAS关联分析结果图详解
一、系统进化树
系统进化树(phylogenetic tree):用来表示群体内物种间亲缘关系远近的树状结构图。
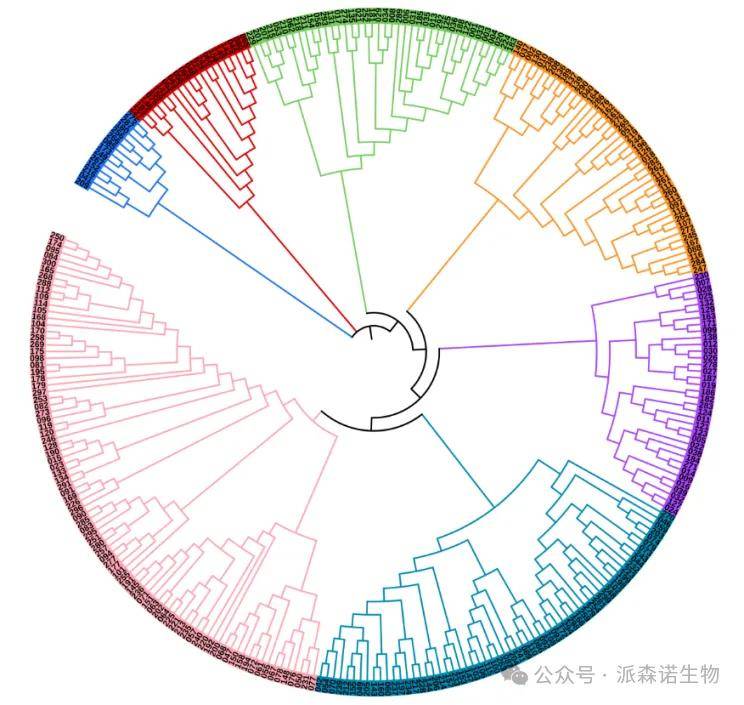
在树中,每个节点代表其各分支的最近共同祖先,而节点间的线段长度对应演化距离(如估计的演化时间)。根据不同物种间的亲缘关系远近,将各个物种分别放置在有分枝的树状图上。进化树上每个叶子结点代表其中一个物种,两个叶子结点之间的距离长短表示相应的两个物种之间的分化程度。
二、主成分分析
主成分分析(Principal Component Analysis,PCA):一种分析、简化数据集的统计方法。PCA 分析经常用于减少数据集的维数,同时保持数据集中的对方差贡献最大的特征。在群体研究中,通常利用个体基因组 SNP 的差异,将不同个体按照主成分聚类成不同的亚群。
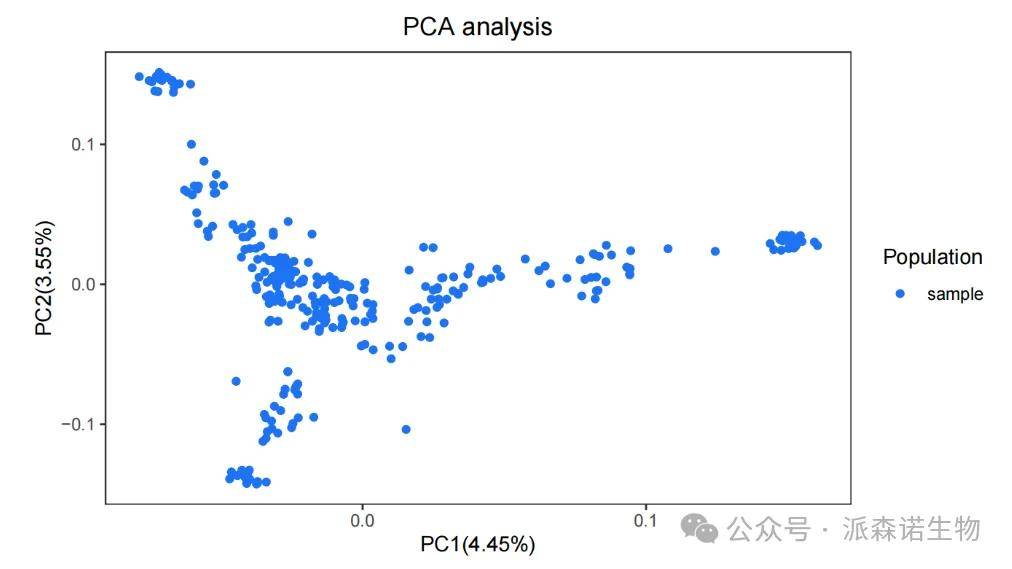
将待分群的多个个体的 SNP 数据组成矩阵,提取该矩阵向量的特征向量(主成分,通常为 3 个),并用其中两个特征向量绘制散点图。根据散点图的分布情况,推知待分群个体的亚群划分。该方法通常和群体遗传结构分析方法、基于 SNP 的系统进化分析等方法相互印证。图中不同的点代表不同的个体。
三、群体遗传结构
群体遗传结构:指遗传变异在物种或群体中的分布。群体的遗传结构受到多个因素的影响,包括突变、选择、迁移、群体大小、环境条件等等。
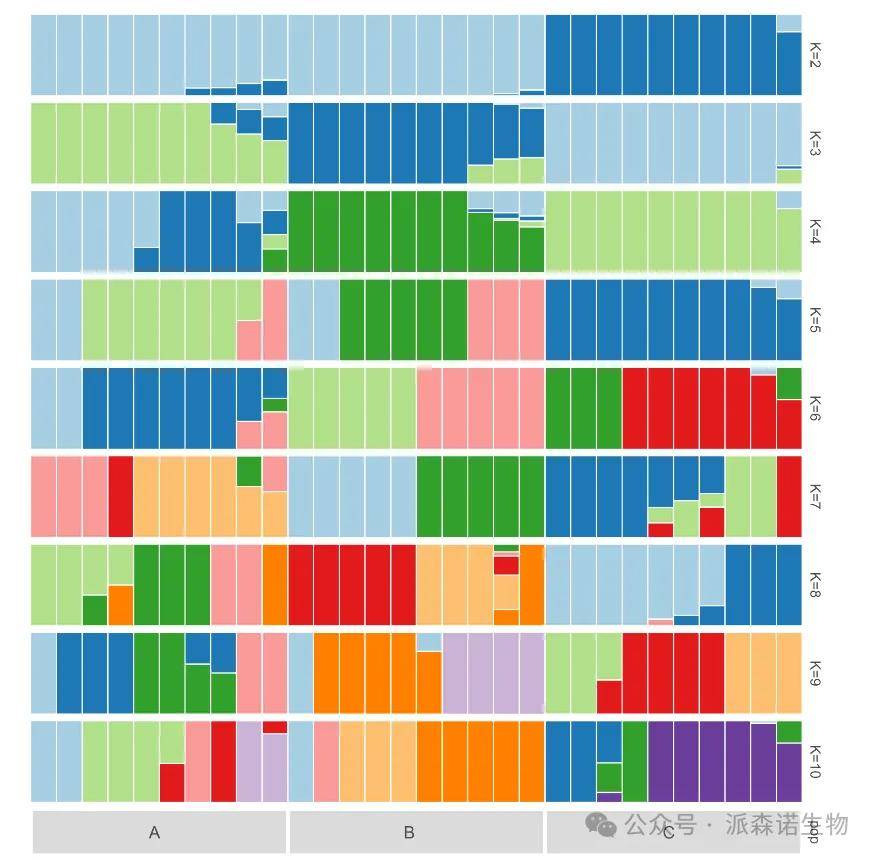
利用 SNP 信息分析群体的遗传结构,设置不同K值(即假设存在多个祖先群体)模型选择为混合模型,图片中每一列代表一个个体,不同颜色片段的长度表示该个体基因组中某个祖先群体片段所占的比例。
四、全基因组关系矩阵(Genomic relationship matrix)
全基因组关系矩阵(GRM)是用SNP标记去估计个体间亲缘关系的一种计算值。
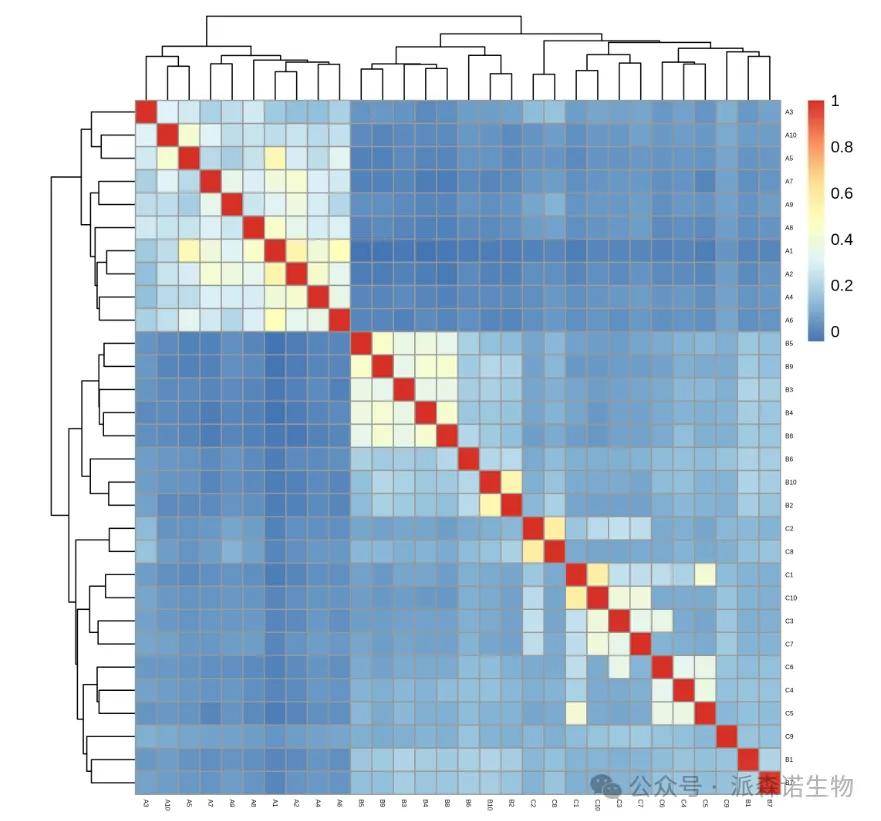
G值是GRM中的元素,表示两个个体间标准化的基因组相似性。横纵坐标为样本ID,即每一个方块表示对应横纵坐标的两个样本间的G值大小。方块的颜色越接近红色,表示G值越大,关系越近;颜色越蓝,表示G值越小,关系越远。其中,对角线元素表示个体自身的基因组相似性;非对角线元素表示个体间的亲缘关系相似性。
五、连锁不平衡衰减(LD decay)分析
连锁不平衡衰减(LD Decay)分析是研究基因组中连锁不平衡(Linkage Disequilibrium, LD)随物理距离增加而减弱趋势的分析方法,其核心目的是评估SNP标记之间的关联程度如何随距离增大而下降。
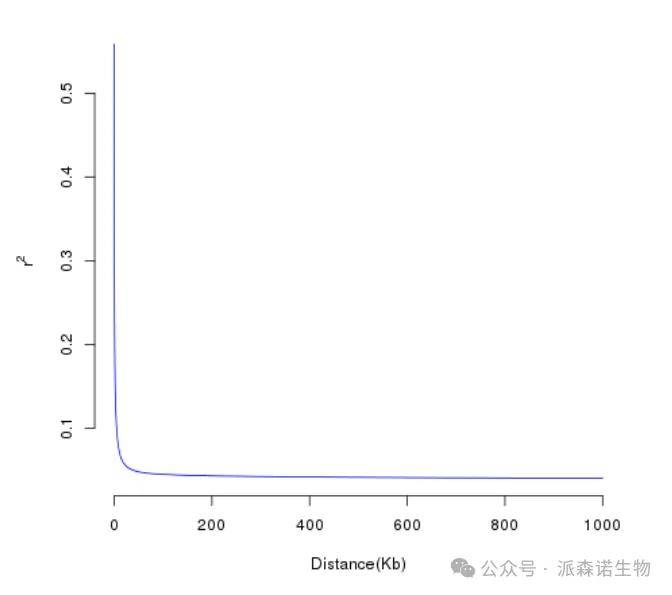
连锁不平衡是指不同位点上的等位基因非随机组合的现象。LD衰减是指随着SNP间物理距离(如碱基对,bp)的增加,LD强度逐渐降低的现象。衰减速率受群体历史(如瓶颈效应、选择)、重组率、突变率等因素影响。常用 r 2(相关系数平方)衡量两SNP的关联强度。例如:r 2=1 表示完全连锁不平衡,r 2=0 表示完全独立。图中横坐标为SNP间的物理距离,纵坐标为平均LD值( r 2表示)。
六、全基因组关联分析
全基因组关联分析(Genome-Wide Association Study, GWAS)是一种利用高通量基因分型技术(如测序),在全基因组范围内扫描与目标性状(如农艺性状、疾病等)显著关联的遗传变异(如SNP)的分析方法。常用曼哈顿图和QQplot图来展示分析结果。
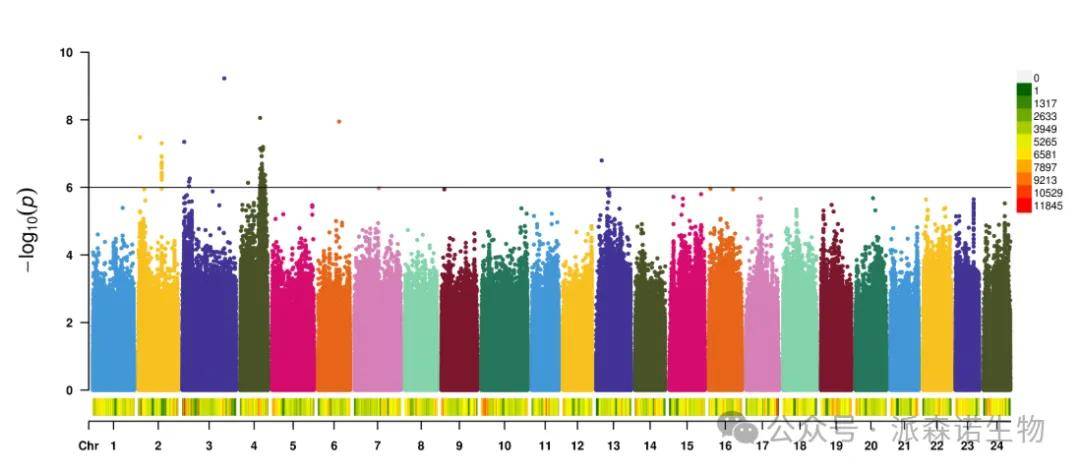
曼哈顿图,横坐标为染色体物理位置,纵坐标为P值取-log10。一个点代表一个SNP。P值越小,即-log10(P value)越大,该位点与性状的相关性越强。虚线表示-log10(P)的阈值,超过阈值的是和性状关联的候选位点。
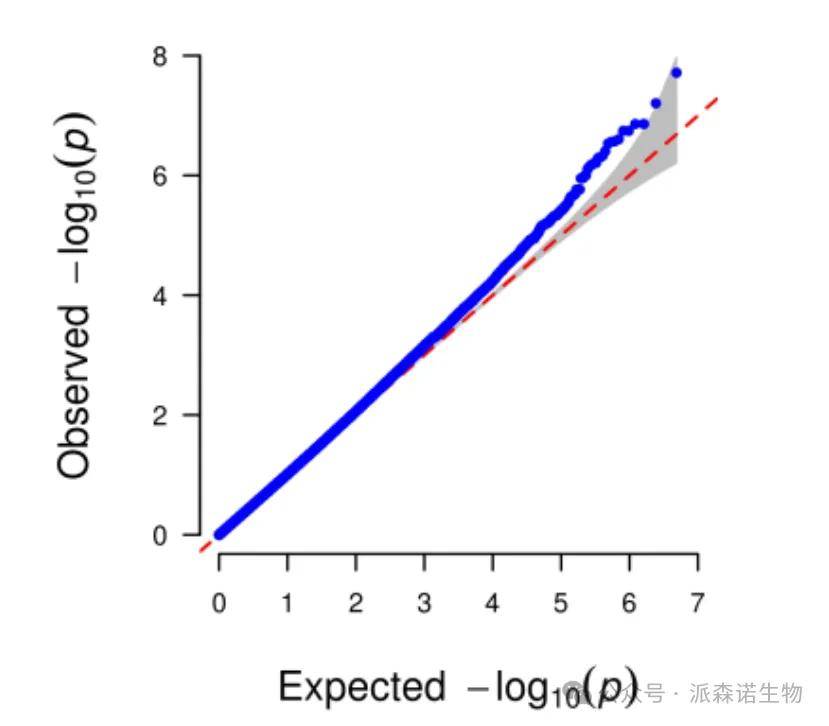
QQ图,横坐标为-log10(P value)的期望值,纵坐标为其观测值。QQ图为评价GWAS结果的可靠性,用于检验关联分析得到的p值分布是否符合预期。通常在越靠近横坐标0的位置,期望值和观测值应该比较接近,横坐标值越大,观测值应该高于期望值。换句话说散点紧贴对角线分布,仅在尾部(高 -log₁₀(p) 区域)略微上翘为正常情况。若整体偏离对角线提升分析可能存在误差问题。
七、LDBlock联合分析
连锁不平衡区块(LD Block)联合分析是GWAS研究中用于精细定位和解释遗传信号的重要方法,其核心目的是利用SNP之间的连锁不平衡(LD)结构,将关联信号划分为具有生物学意义的连续区块,从而更准确地定位潜在因果变异或功能基因。
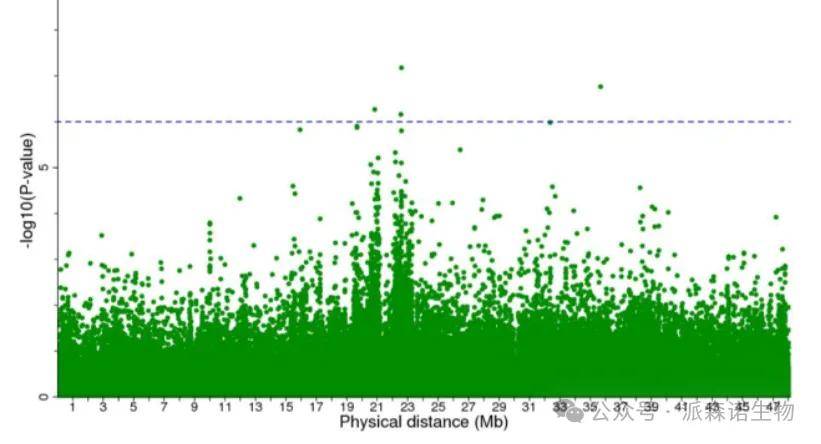
一般情况下,全基因组关联分析的曼哈顿图常常与染色体位置、LD-Block图进行联合分析,根据显著关联的SNP位点确定注释的染色体区域,同时在该区域进行LD-Block分析,找到与该位点连锁较强的其他SNP位点,并对其所在的基因进行功能注释。
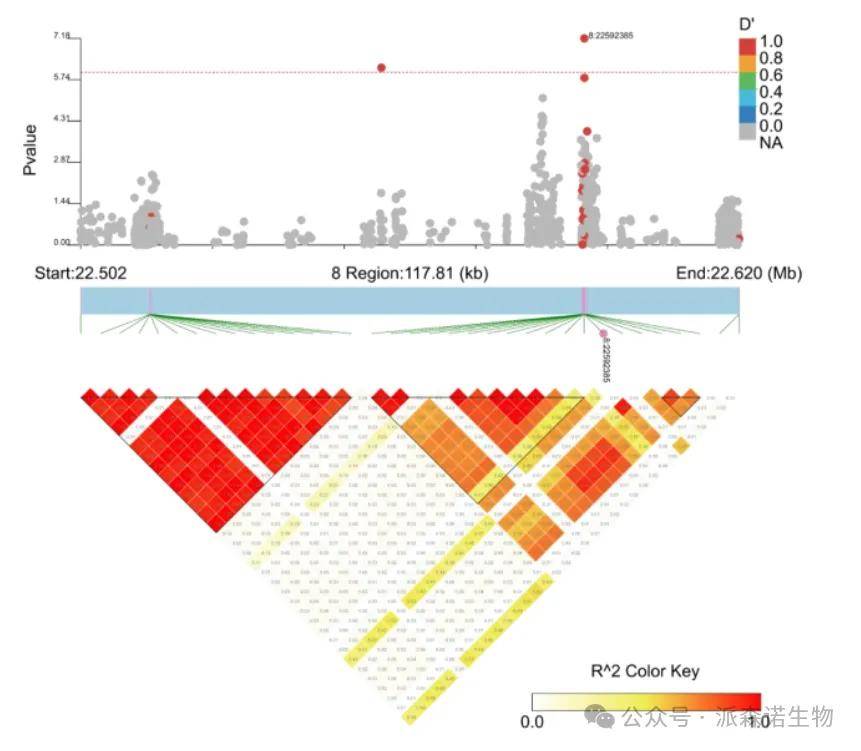
上图曼哈顿图表示关联位点,横坐标表示分布于每条染色体上的SNP位点,纵坐标表示每个位点的-log10(P)值;下图表示关联区域内的BLOCK分析,图上部为标记在染色体上的位置,下部的倒三角图表示每个SNP位点和其它位点之间的r2值,颜色越深表示连锁关系越强。


