2025-06-23
星云图是单细胞转录组研究中一种独特的可视化方法,它通过将不同细胞类型的空间分布与基因表达模式相结合,形成多样 “星云”,展现基因表达异质性、聚类状态及细胞类型间差异关联。相较常规单细胞测序细胞类型可视化,它能更突出细胞类型间基因表达整体差异,依基因表达谱和聚类信息绘制,借 “星云” 特征观察聚类、识别模式,助力挖掘分子特征与细胞分化关联 。
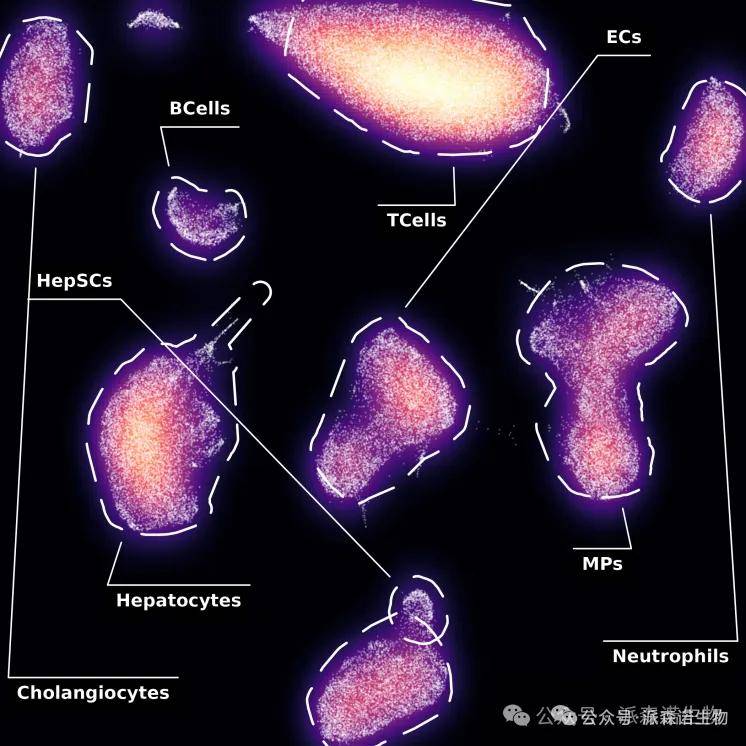
这是单细胞转录组测序数据分析里的单细胞转录测序星云图,把不同细胞类型(像图里的 B 细胞、T 细胞 、肝细胞等 )的基因表达信息,以类似 “星云” 的可视化形态呈现。不同色彩、形态的 “星云” 区域,对应特定细胞类型(如标注的各类细胞),能直观展现细胞群体在基因表达层面的异质性,想自己动手绘制?安排!
1、数据准备
library(Seurat)
library(ggplot2)
Obj <- readRDS(‘你的SeuratObject.rds’)
group.by <- ‘celltype’ #细胞按什么分类
Reduction <- ‘umap’#细胞用什么降维方法展示
df <- data.frame(dim1=Obj[[reduction]]@cell.embeddings[,1],
dim2=Obj[[reduction]]@cell.embeddings[,2],
cluster=Obj@meta.data[[group.by]])
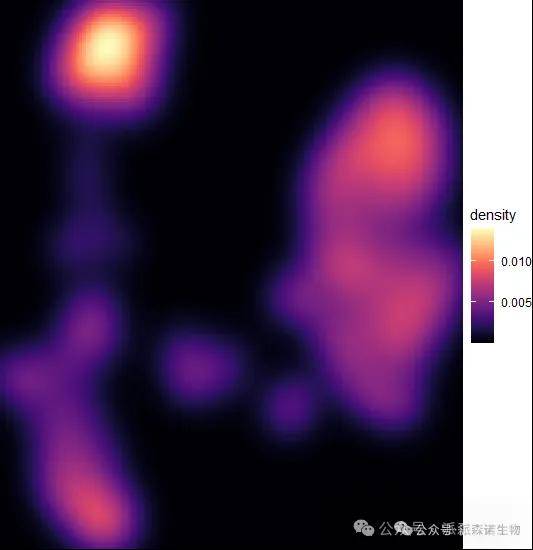
2、绘制”云图”
p <- ggplot(df, aes(x = dim1, y = dim2)) +
stat_density_2d(
geom = "raster",
aes(fill = after_stat(density)),
contour = FALSE, n = 200
) +
viridis::scale_fill_viridis(option = "magma", direction = 1)+
coord_cartesian(expand = F) +
theme_void()
p
p
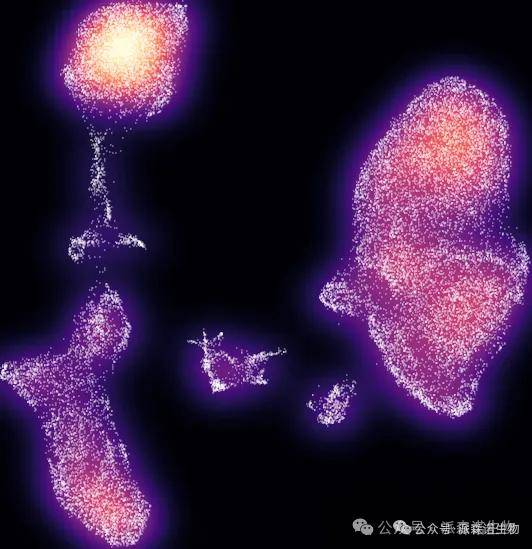
3、绘制”星图”
p <- p+ geom_point(color = "#FFFFFFAA", size = 0.005, alpha = 0.2)+
theme(legend.position = "none")
p
4、添加细胞分类框
remove_outlier <- function(x,y,proportion=0.9){
stopifnot(length(x)==length(y))
distance_to_centroid = (x-mean(x))^2 + (y-mean(y))^2
kept_site <- order(distance_to_centroid)[1:ceiling(length(x)*proportion)]
return(kept_site)
}
df2 <- lapply(unique(df$cluster),function(x){
new_df <- df[df$cluster==x,,drop=F]
return(new_df[remove_outlier(new_df$dim1,new_df$dim2),,drop=F])
}) |> dplyr::bind_rows()
p <- p + ggforce::geom_mark_hull(data=df2,
aes(x=dim1,y=dim2, group = cluster,label = cluster),
linewidth=0.8,
concavity = 5,
color='white',
con.colour='white',
con.type='straight',
label.fontsize=12,
label.colour='white',
label.fill='black',
expand=unit(3, "mm"),
linetype='f8')
p
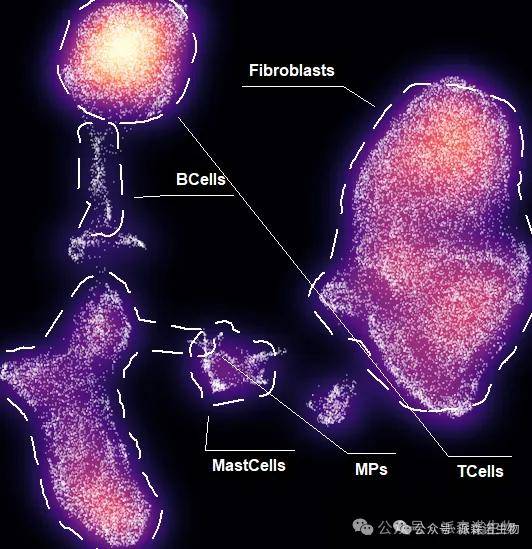
这样,细胞密度星云图就画完了,是不是比普通的Umap散点图漂亮呢?但是要注意只有细胞分类在降维图上的边界很清晰星云图才好看,不然的话可是会丑哭的呦~


