2025-07-22

Highlights
突破传统参考基因组的限制——T2T基因组精准定位表观修饰在传统"基因组暗物质"中的调控作用(如DNA甲基化、组蛋白修饰);
作物驯化与抗逆性表观机制——解析驯化基因(如玉米tb1)的表观调控,追溯人工选择对表观组的塑造;环境胁迫(干旱/病害)诱导的表观记忆,为分子设计育种提供新靶点。
高通量数据处理——派森诺擅长技术创新与多组学整合,通过长读长测序(PacBio HiFi/ONT) + 表观组学(WGBS/ATAC-seq/ChIP-seq)实现"序列-功能"双重解析。
1、简 介
T2T(端粒到端粒)基因组提供了完整的、无缺口的基因组序列,结合表观遗传学(DNA甲基化、组蛋白修饰、染色质可及性等),可以更全面地解析动植物基因组的调控机制、进化适应性和重要农艺性状的形成。
2、主要应用
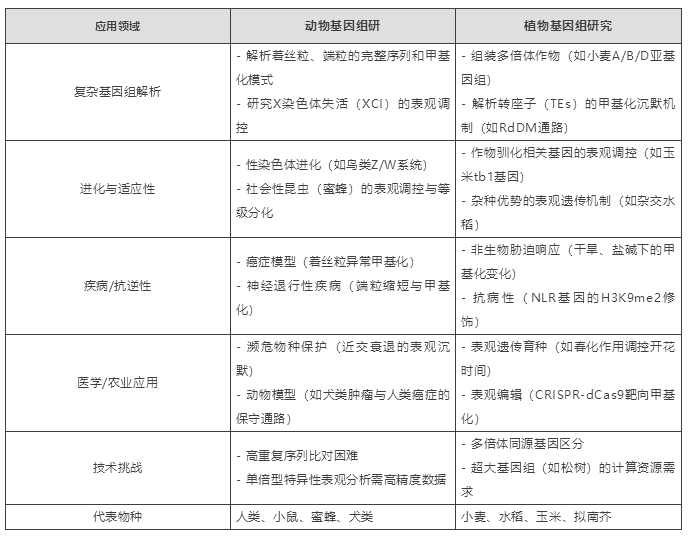
填补基因组空白:T2T解析着丝粒、端粒等重复区域,表观揭示其调控机制。
单倍型分辨率:区分父源/母源等位基因的表观差异(如印记基因、杂种优势)。
多组学整合:结合Hi-C、WGBS、ATAC-seq等数据构建全基因组调控网络。
经典案例

期刊:iMeta
影响因子:33.2/Q1
发表单位:中国农业科学院北京畜牧兽医研究所、重庆市畜牧科学院、黑龙江省农业科学院、岳麓山实验室、湖南农业大学、西北农林科技大学、扬州大学、墨西拿大学、四川农业大学
分析内容:T2T基因组、泛基因组、转录组、表观组(CUT&Tag)、SV&SNP-GWAS、GS。
方法:该研究使用了图谱全基因组方法来评估猪育种中结构变异(SV)的作用,并将其应用于六个屠宰特征的关联分析和基因组选择(GS)。研究人员收集了来自大型白猪×民猪F2群体的540个个体的重测序数据,并使用已建立的图谱全基因组对其进行基因分型。然后进行了基于SNP和SV的全基因组关联分析(GWAS),并利用GS对六个屠宰特征进行了预测。研究人员还比较了不同标记组合在不同屠宰特征上的准确率,并通过多重转录组分析探讨了温度适应与SV的关系。
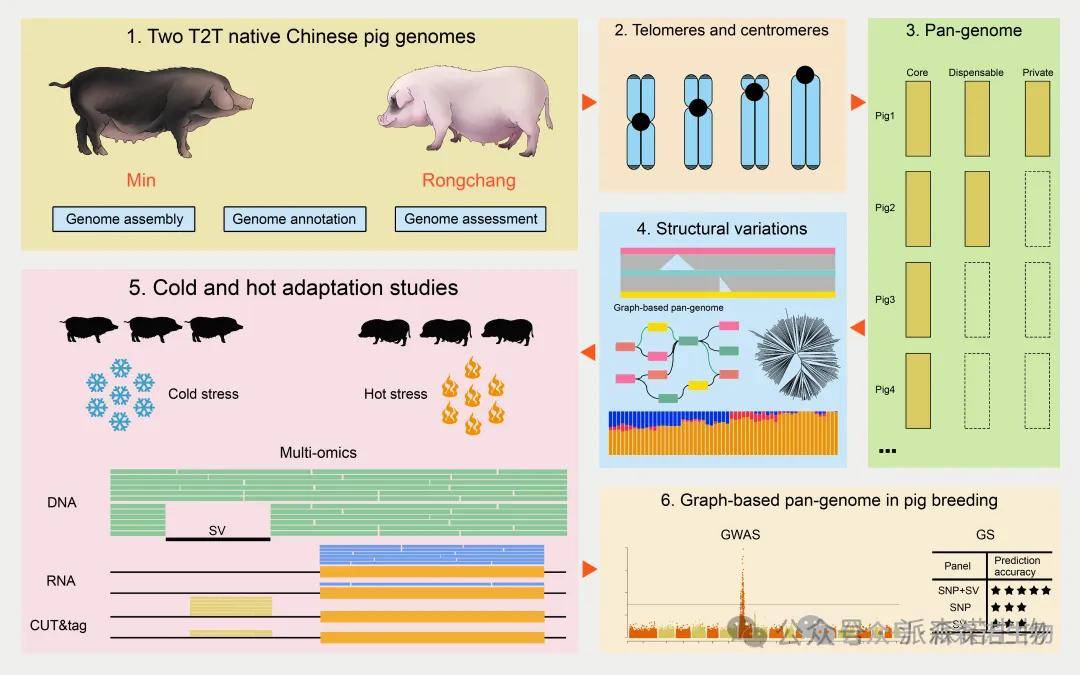
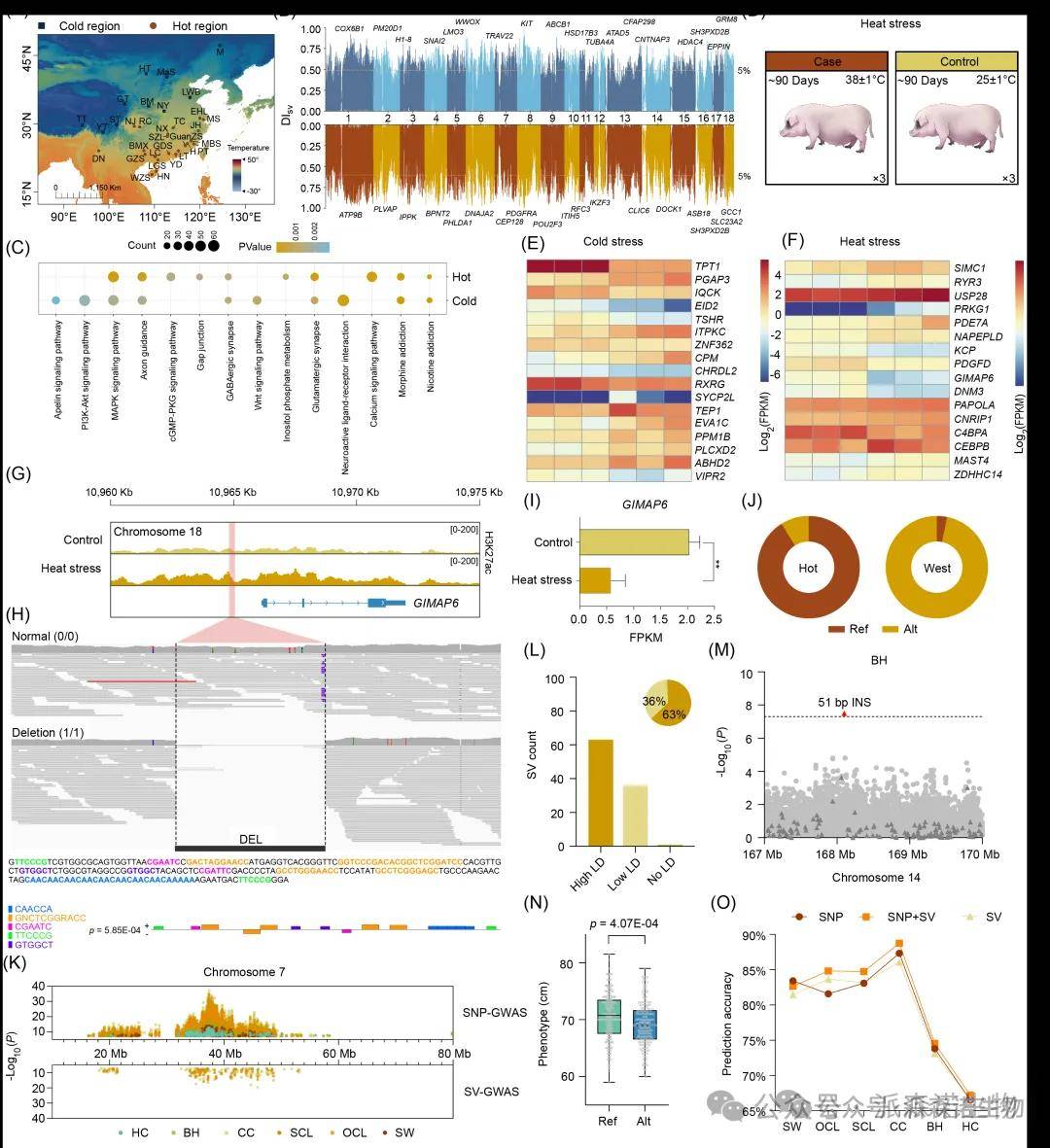
结果:在这项研究中,研究人员采用了多组学数据来探究猪的结构变异(SV)与其遗传适应性的关系。首先,他们对两个中国本土猪种——东北地区的“民猪”和西南地区的“荣昌猪”的全基因组进行了组装,并对其进行了注释。通过比较两个猪种的基因组,研究人员发现了一些新的基因,并预测了它们的功能。同时,他们还研究了SV在不同环境应激下对基因表达的影响。通过对SV区域的转录本测序和定量PCR验证,研究人员发现了一些与温度适应相关的基因,并确定了一些潜在的调控元件。最后,他们还使用了cut-and-tag技术来进一步研究SV与染色质互作的关系。这项研究表明,SV在猪的遗传适应性和表观遗传调节中起着重要作用,这为未来的猪遗传改良提供了重要的见解和资源。
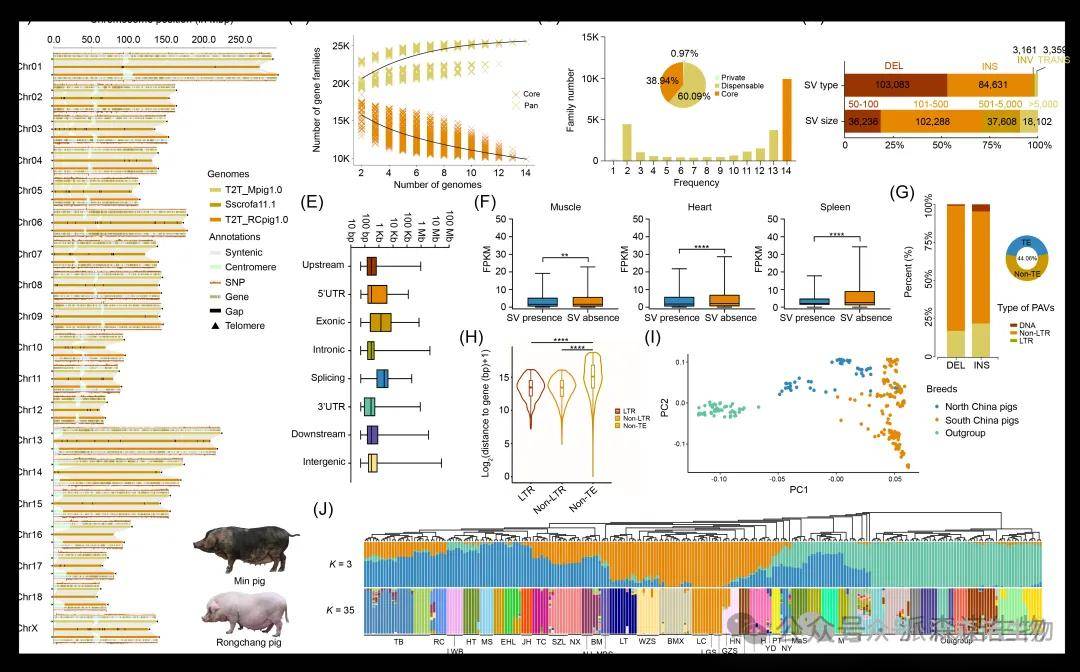
[图]两个 T2T 猪基因组组装和泛基因组研究
[图] 多组学分析猪的温度适应性与图形泛基因组在猪育种中的应用
结论:这项研究表明,结构变异(SV)在猪的遗传适应性和表观遗传调节中起着重要作用。通过比较两个中国本土猪种的基因组,研究人员发现了新的基因,并预测了它们的功能。此外,他们还研究了SV在不同环境应激下对基因表达的影响,并确定了一些潜在的调控元件。最后,他们还使用了cut-and-tag技术来进一步研究SV与染色质互作的关系。这些发现为未来的猪遗传改良提供了重要的见解和资源。
参考文献:
Wencheng Zong, Li Chen, Dongjie Zhang, et al. “Two telomere-to-telomere pig genome assemblies and pan-genome analyses provide insights into genomic structural landscape and genetic adaptations”. iMeta 4: e70013. https://doi.org/10.1002/imt2.70013.


