
派森诺基因云全新推出定量蛋白组学云分析平台,集成12大分析模块、50+分析内容,覆盖从表达分析、功能注释到通路可视化等全流程研究需求。项目结果在手,如何快速锁定关键信息?本期为您详解「全局模块」功能,助力高效分析,事半功倍。
1、项目概括
该模块主要是对本项目相关信息进行展示,包括项目信息、样本信息、分析软件、实验报告等。
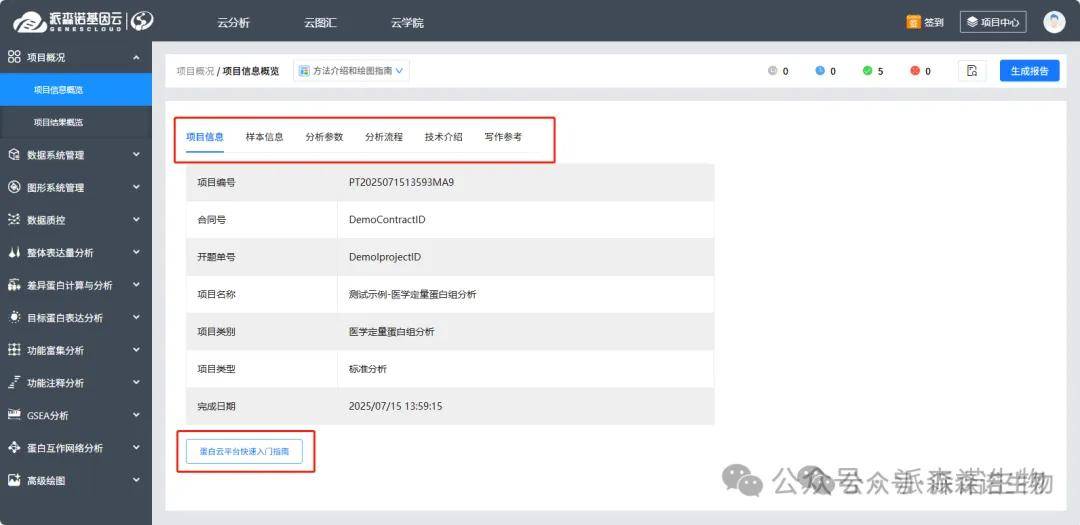
1)项目信息:展示项目编号、合同号、名称及类别等内容,便于查阅和核对项目数据。
2)样本信息:展示命名规范的原始样本名称、分组名称,及样本来源物种信息。
3)分析参数:详细列明各分析模块所对应软件版本号,便于深入了解软件详情和算法依据。
4)分析流程:该项目整体的分析逻辑和分析内容概括。
5)技术介绍:概述了该技术的实验原理与实验流程。
6)写作参考:包含实验报告、原始数据上传指南文档,用于论文写作材料的参考。
2、项目结果概览
该模块主要展示项目鉴定蛋白数、蛋白功能注释率、差异蛋白数等整体概括。
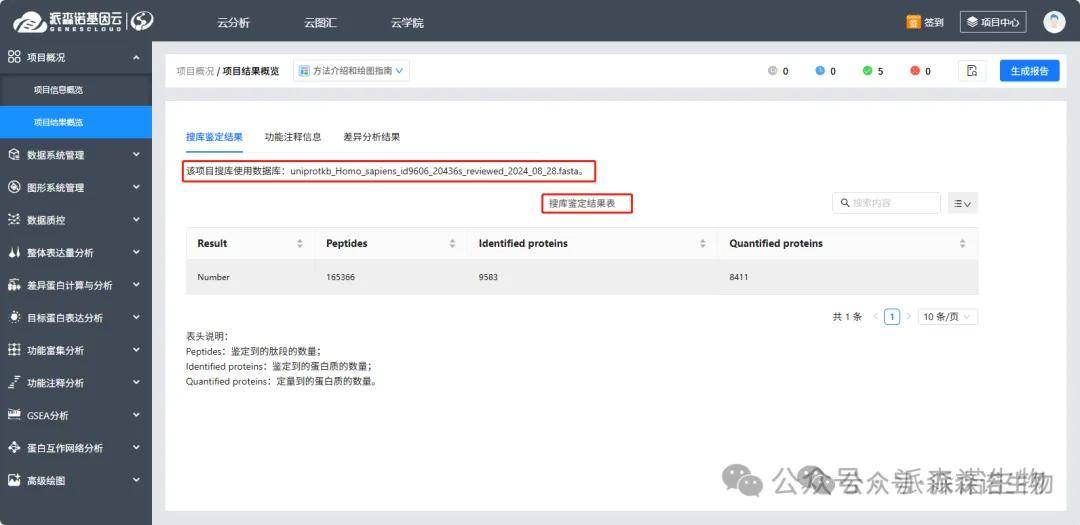
1)搜库鉴定结果:包含搜库定性定量所用数据库序列,及原始鉴定到的肽段和蛋白数。
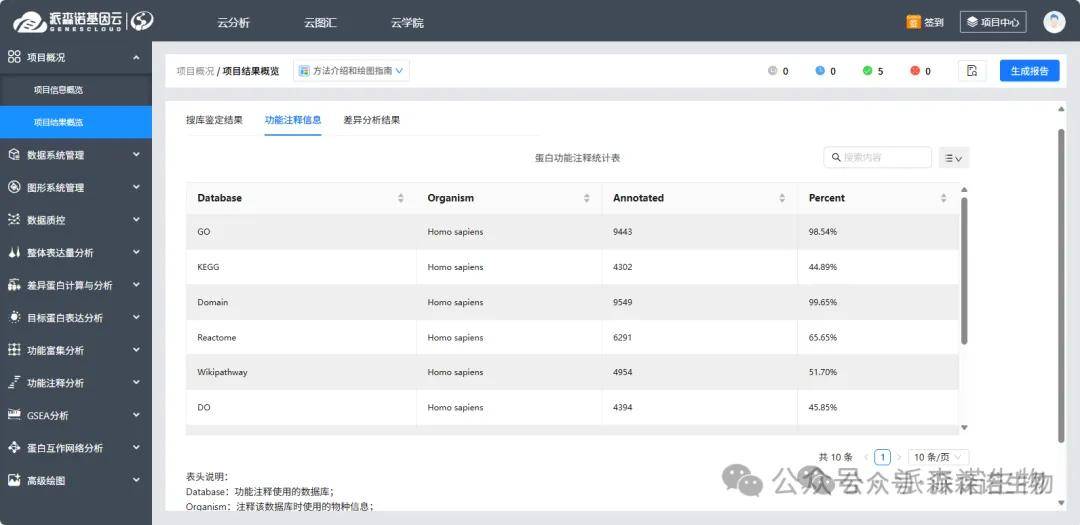
2)功能注释信息:GO、KEGG、结构域、亚细胞定位等功能注释率统计,即该项目中有多少蛋白能够进行该功能注释。
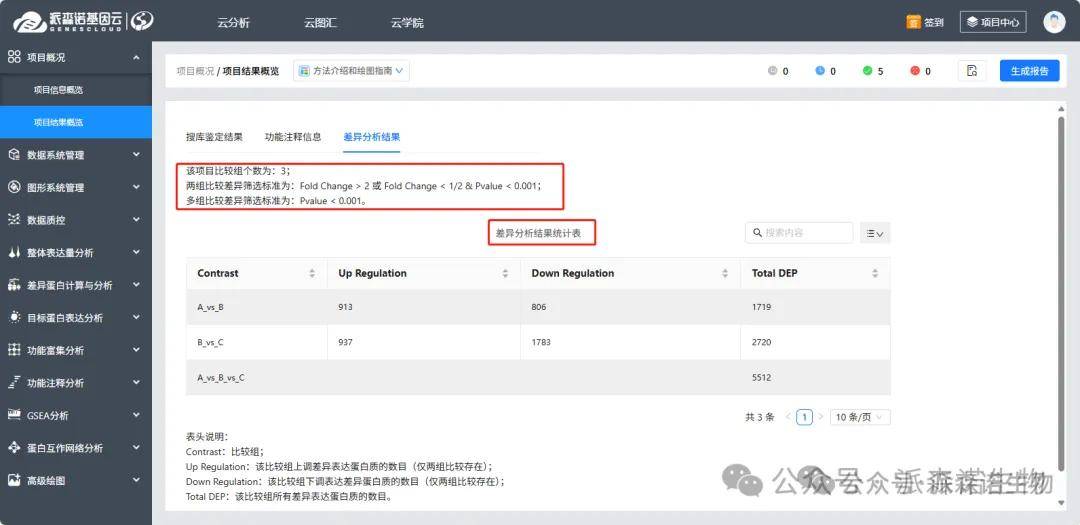
3)差异分析结果:展示了该项目默认的差异标准,和基于该差异标准筛选到的上、下调差异蛋白数量统计。
3、数据质控
该模块主要从单个样本蛋白鉴定数、蛋白理化性质、iRT、QC等初步评估检测流程的稳定性与数据的可靠性。
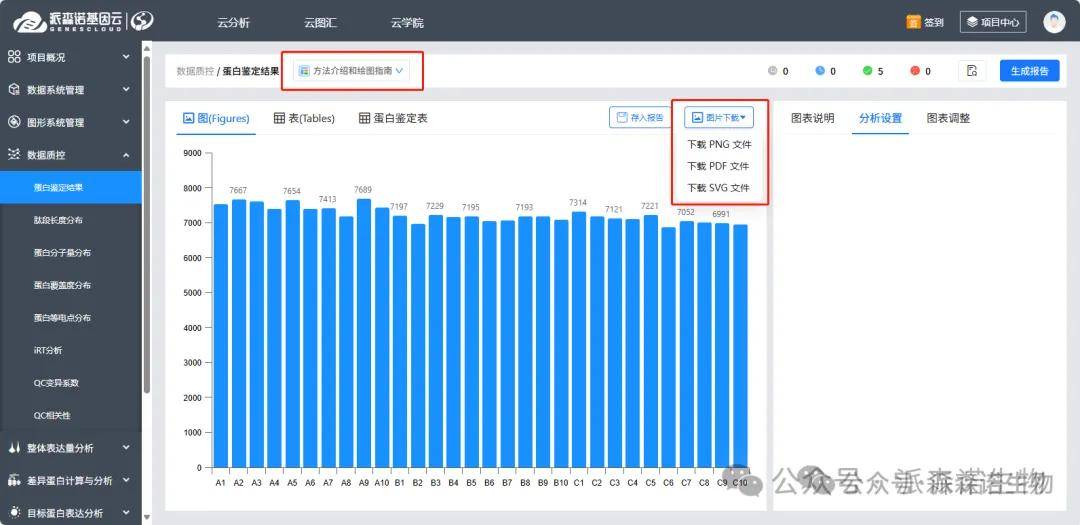
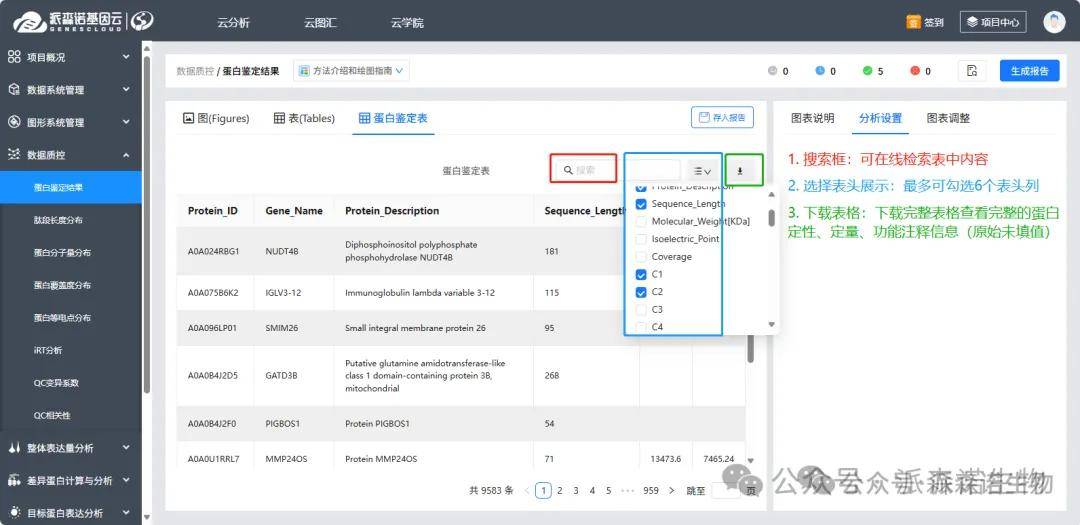
1)蛋白鉴定结果:“图”为鉴定统计柱状图,“表”为鉴定统计,“蛋白鉴定表”可选择在线展示部分表头内容,进行在线检索蛋白ID、蛋白名称、基因名称等信息,详细的鉴定数据可进行表格的下载,这里的鉴定结果表是未进行过滤、填空值的原始鉴定表。
2)肽段长度、蛋白分子量、蛋白覆盖度、蛋白等电点:针对所有鉴定蛋白的理化性质指标进行统计分析,了解蛋白主要的分布范围。
3)iRT分析:该项目≥6个样本时,会通过内标质控提取、上机等整体流程的可靠性与稳定性,每个内标肽越平稳,说明该项目检测稳定性越高,数据可靠性越高。
4)QC变异系数、QC相关性:该项目≥12个样本时,会通过混合溶液QC进行质控,一般要求QC变异系数<0.3的蛋白占比>70%,QC相关性>0.85,说明数据可靠性较高。
4、整体表达量分析
该模块主要对蛋白表达量水平进行整体把控,先对原始搜库结果进行数据预处理,进而根据每个样本的表达量分布,判断样本之间的相对距离远近(组内重复性和组间分离度)。
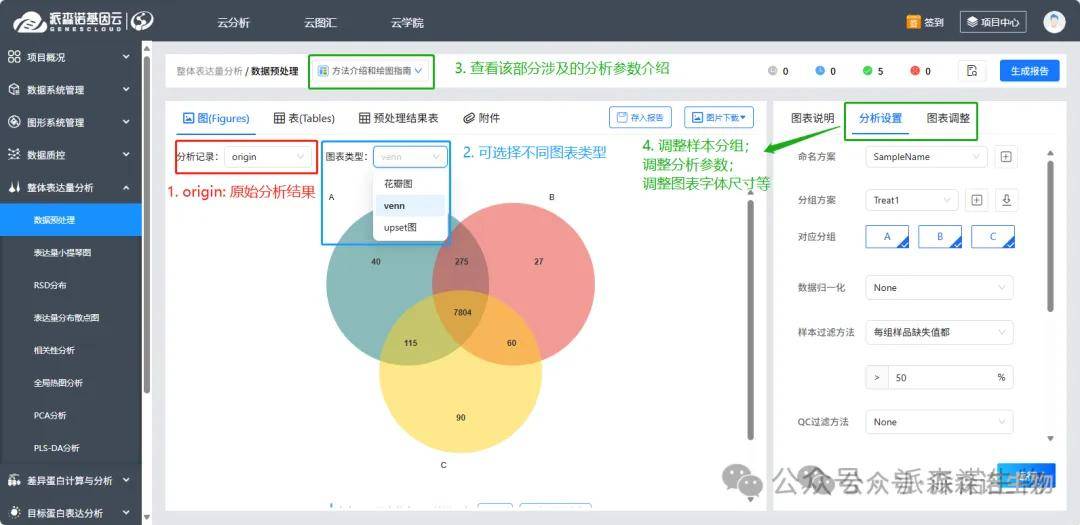
1)数据预处理:①先对搜库原始表分别进行过滤、缺失值填充处理,得到“预处理结果表”,进而用于后续分析,可在线查看、检索与图表下载。②分析记录为“origin”的即为该项目默认参数下进行预处理的结果,如调整运行重分析,新的分析结果可通过“分析记录”切换来展示。③右侧“分析设置”中为详细对应的参数,老师可根据需求进行调整,再“运行”分析(默认参数均为相对较优方式,建议不作调整);“图表说明”为图表的意思解读;“图表调整”可进行图中文字、尺寸、配色等调整。④上方“分析介绍”对该部分的处理过程进行了详细的介绍说明。⑤“附件”下为未处理的原始表和预处理中间文件。
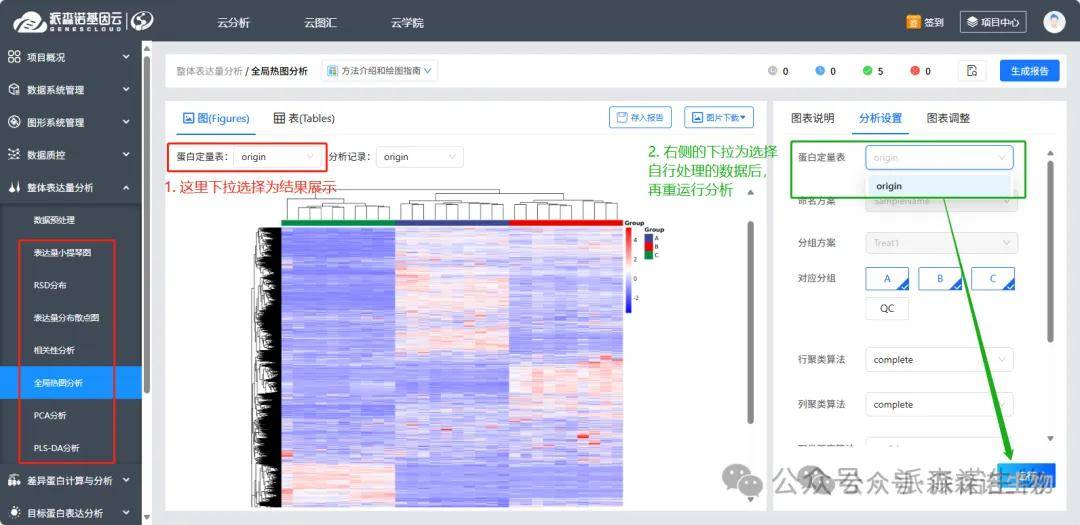
2)表达量小提琴图等:“蛋白定量表”对应的“origin”即为前述的“预处理结果表”。如果“数据预处理”部分有调整参数重分析,会生成一个新的蛋白定量表,就需要在右侧的“分析设置--蛋白定量表-选择新的蛋白表-运行”进行该部分的重绘图。这些绘图主要用到蛋白的定量数据,不同的分析图会对定量数据进行不同的标准化处理,详见右侧“分析设置”下的各个参数,不同分析图和表头的意思详见“图表说明”。
5、差异蛋白计算与分析
该模块主要针对比较组方案,进行差异蛋白的分析筛选,分析图分别以差异蛋白数量统计柱状图、火山图、单个差异蛋白显著性柱状图等展示,对应的表均可在线查看、检索、选择及下载。
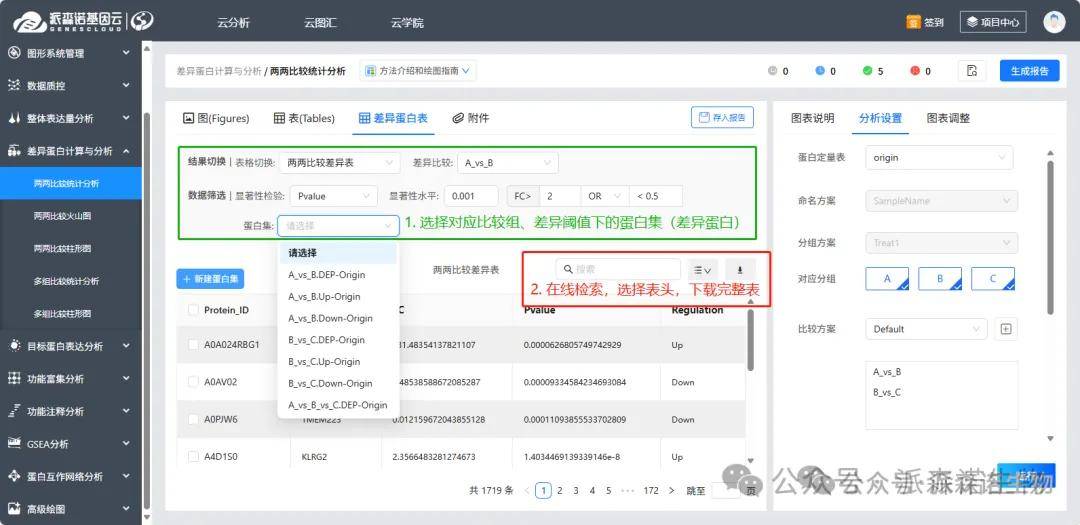
1)两两比较统计分析:在“分析设置”下对应的差异标准进行差异蛋白的筛选,获得差异蛋白信息:DEP为差异表达的蛋白,Up为上调的差异蛋白,Down为下调的差异蛋白,DEP=Up+Down,all.DEP是所有比较组的差异蛋白的并集;带“Origin”字样的为默认分析参数的筛选结果。在线表格勾选蛋白可自定义创建目标“蛋白集”,用于指定的可视化作图。
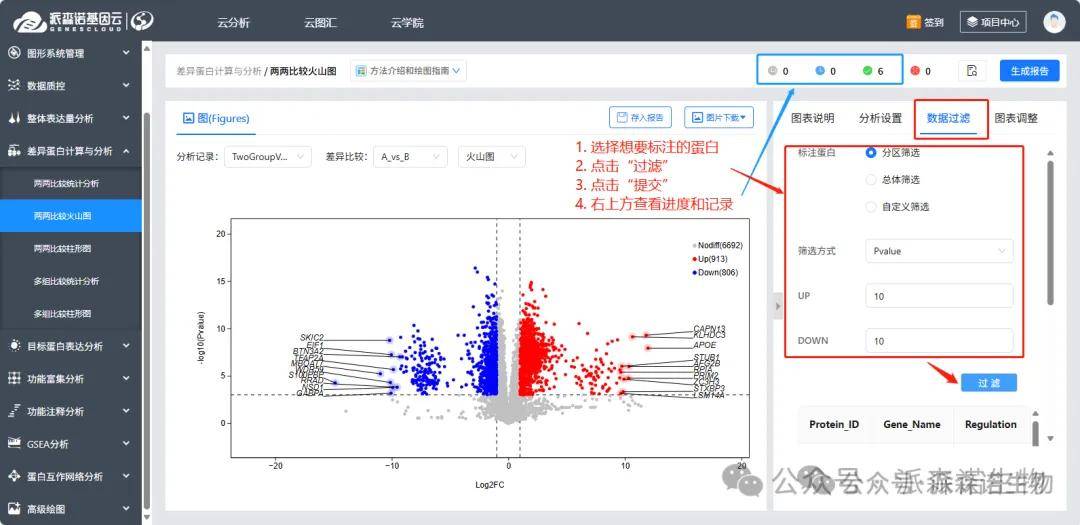
2)两两比较火山图:根据需求标注想要标注的蛋白ID或者基因名称。右侧“数据过滤”①分区筛选:上、下调蛋白分别筛选;②总体筛选:全部差异蛋白进行筛选;③自定义筛选:粘贴蛋白ID到文字框;点击“过滤”,下方为预览表,再点击“提交”即可重新绘图,右上方分析进程变绿后表示分析完成,然后“分析记录”下拉选择新的分析结果查看、下载即可。
6、目标蛋白表达分析
该模块默认用前面筛选的差异蛋白进行聚类热图、趋势图(大于3个组)、维恩分析(大于2个比较组)、雷达图的可视化,展示差异蛋白在组间的上下调变化趋势。
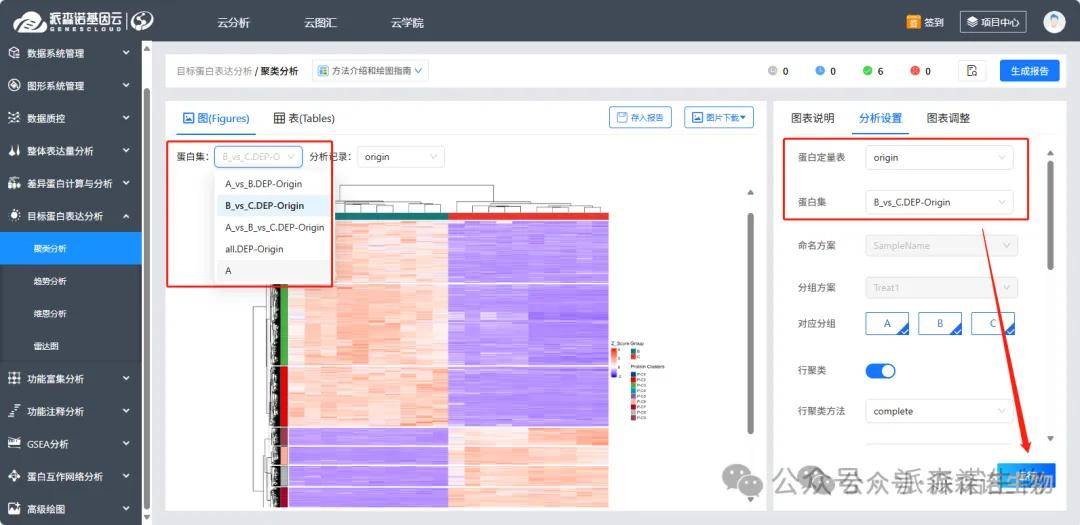
1)聚类分析:选择不同“蛋白集”查看各个比较组的差异蛋白在组间的上下调变化趋势。如果调整参数重新筛选差异蛋白,或者新建目标蛋白集,在“分析设置--蛋白集”选择新的蛋白集,点击“运行”即可获得新的分析记录。
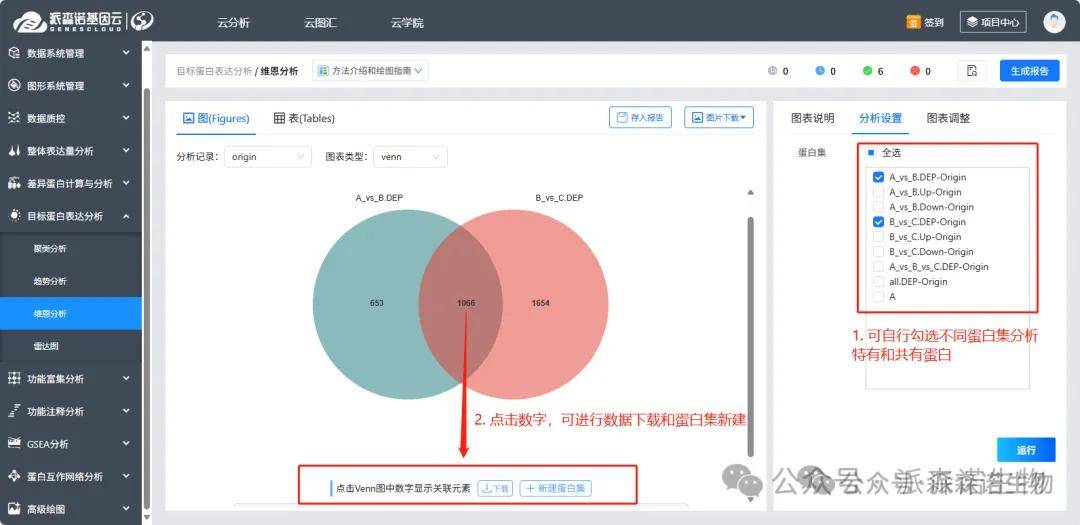
2)维恩分析(要求至少2个比较组):展示不同比较组方案中特有和共有差异蛋白,右侧可自行勾选蛋白集进行绘图分析,点击图中数字可下载查看,以及新建感兴趣的“蛋白集”,用于指定模块的分析。
7、功能富集分析与功能注释分析
该模块主要对每个比较方案下的差异蛋白进行GO/KEGG/Domain/Reactome/WikiPathway以及疾病特有的DO/DisGeNET等多个数据库功能通路进行富集分析,辅助挖掘潜在的功能机制,以及转录因子、激酶注释,亚细胞定位和细胞外基质分析。
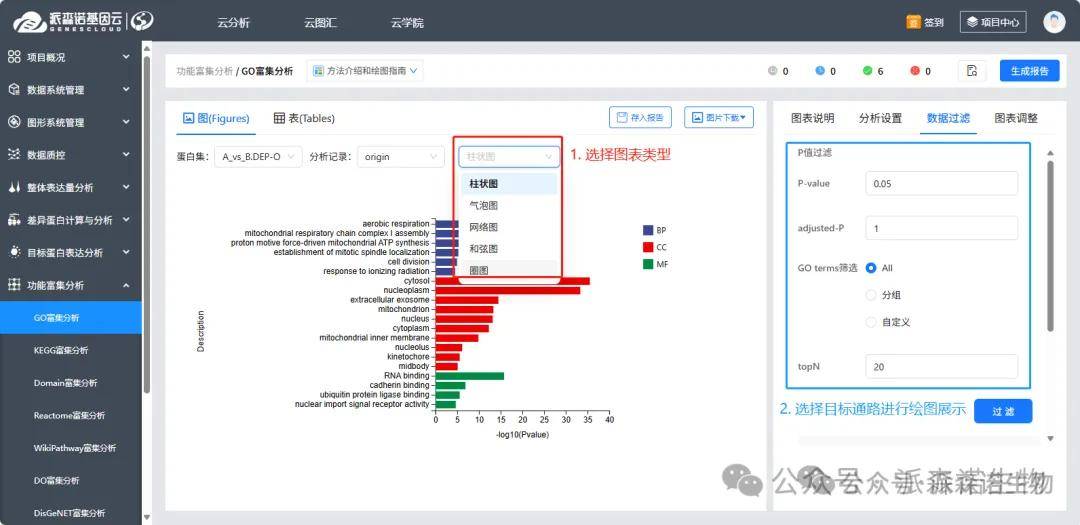
1)富集分析:具有多种图表类型,老师根据需求选择即可。右侧“数据过滤”可进行目标通路的筛选及绘图。①All:从所有富集到的通路中筛选;②分组:按照通路类型进行筛选;③自定义:根据自己需求,粘贴目标通路进行筛选。选择后“点击过滤--勾选通路--点击提交”,即可对目标通路进行绘图,分析完成后可从分析记录中查看新的结果图。
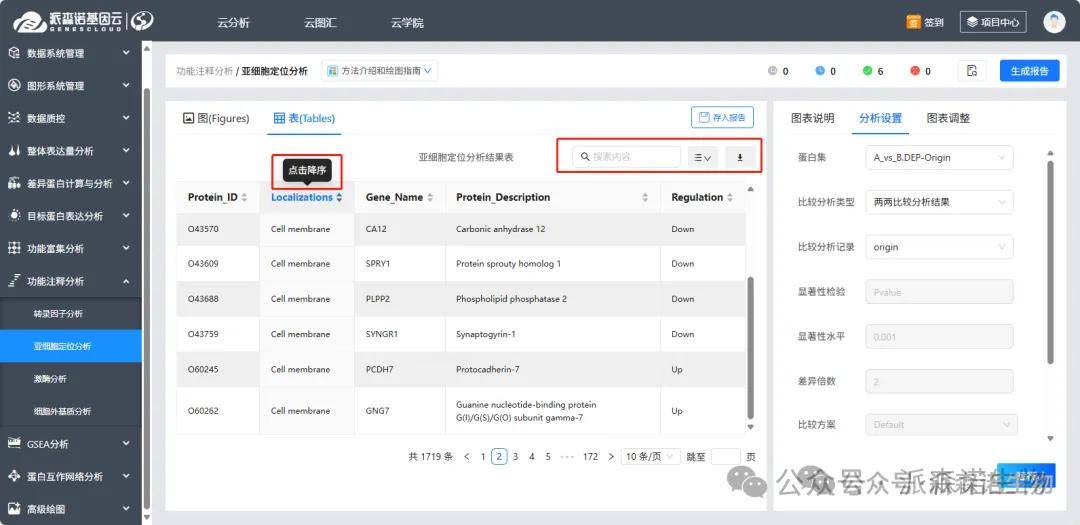
2)注释分析:默认针对差异蛋白进行注释统计分析,“表”模块可以在线检索、筛选感兴趣的内容,也可以下载线下excel进行查看、筛选处理等。
8、GSEA分析
该模块用所有蛋白按照在两组样本中的差异表达程度进行排序,然后采用统计学方法检验预先设定的蛋白集合是否在排序表的顶端或底端富集。判断该通路在两组中可能处于激活还是抑制状态。
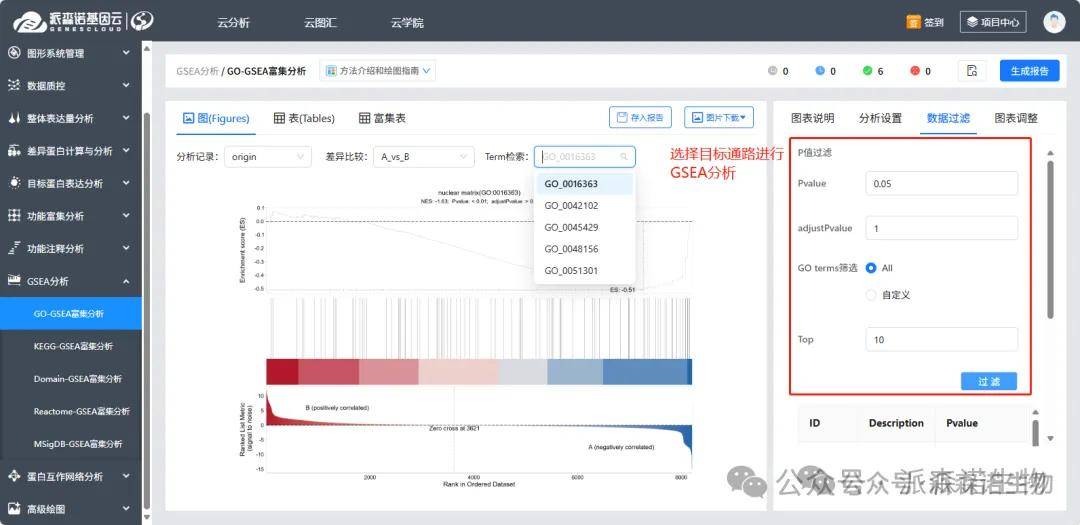
1)通路-GSEA分析:因该部分计算量较大,每种通路数据库的GSEA分析默认只展示5条通路,老师可根据需求在右侧的“数据过滤”中选择目标通路,点击“过滤--提交”即可对目标通路进行绘图,分析完成后可从分析记录中查看新的结果图。
2)GSEA分析的理解:①ES为正,表示该通路在处理组中被激活,在对照组中被抑制;ES为负,表示该通路在处理组中被抑制,在对照组中被激活;②一般认为|NES|>1,p value<0.05,adjustPvalue<0.25的通路是显著富集的,NES绝对值越大表示富集程度越高。
9、蛋白互作网络分析
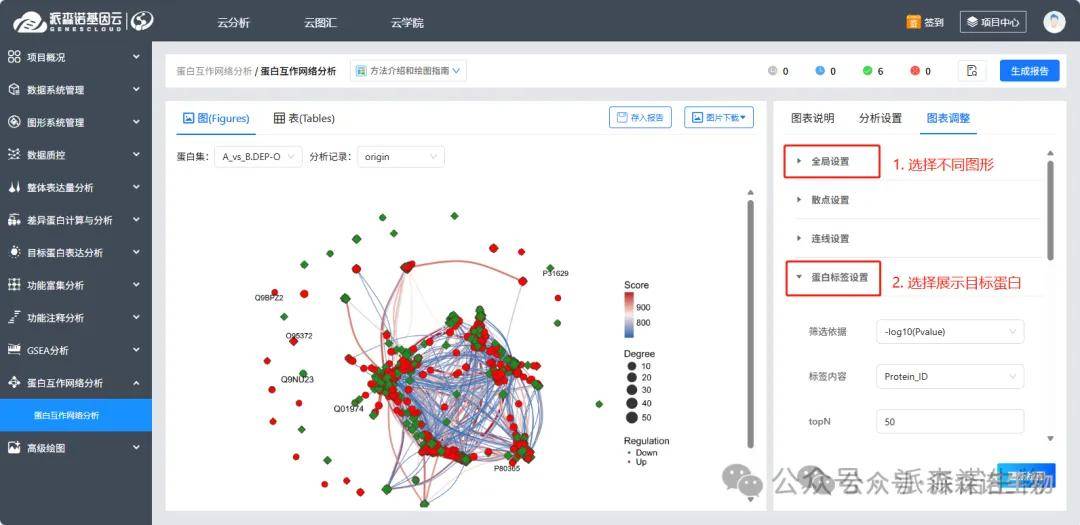
该模块是针对筛选到的差异蛋白进行蛋白互作分析,旨在筛选相互作用强的蛋白,这些可能是通过相互作用发挥一些功能。“图表调整--全局算法”可选择不同的图形展示形式,“图表调整--蛋白标签设置”可选择目标蛋白进行展示。
派森诺定量蛋白组云平台,从项目概览奠定数据基础,以数据管理深化分析洞察,凭严格质控保障结果可靠,为蛋白组学研究提供全流程解决方案。诚邀您体验云平台的智能化与便捷操作,并持续关注后续系列解读,发掘蛋白组学数据的深层价值,让每份数据都成为驱动科研突破的关键力量。




















