

文章亮点
构建了一个包含29个葡萄基因组全基因组图谱,这是目前最大的葡萄基因组数据集之一;
通过GWAS分析发现了许多与果实品质相关的基因,这有助于改善葡萄的品种选育和提高产量;
研究者还提出了一些新的策略,例如针对SV的遗传分析和多目标育种等,这些策略有望进一步提高葡萄育种的效率和质量。
文章信息
英文题目:Grapevine pangenome facilitates trait genetics and genomic breeding中文题目:葡萄泛基因组助力性状遗传学与基因组育种研究
发表期刊: nature genetics
影响因子:29.0/Q1
发表时间:2024年10月1日
涉及组学:泛基因组、GWAS、GS。
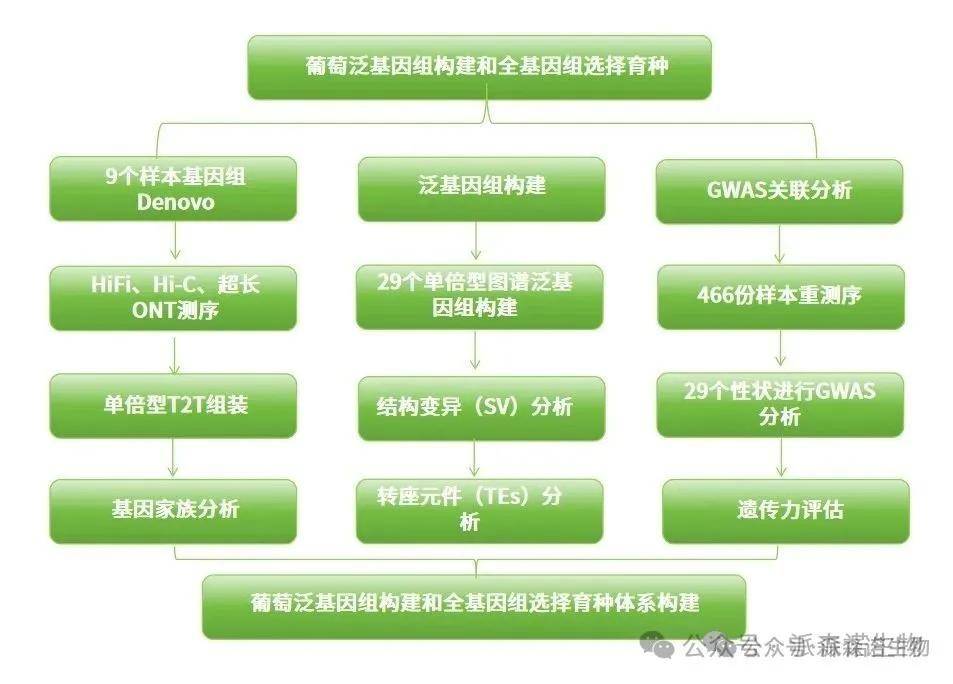
技术路线
摘 要
本研究旨在通过构建葡萄基因组参考图谱(Grapepan v.1.0)和对466个葡萄品种进行重测序数据的分析,探索复杂农艺性状的遗传基础,并为育种提供支持。研究人员使用SNP和结构变异(SVs)建立了一个变异地图,并将SVs整合到全基因组关联研究中,共发现了29个农艺性状的148个数量性状位点,其中12个性状显著受SVs影响。此外,还发现了一些代谢和果实发育等不同类型的性状之间的遗传相关性。研究结果表明,包括SVs在内的全基因组关联研究可以提高遗传可解释性的平均值,并且在多性状选择模型下,可以通过基因组预测方法来实现多个性状的育种。这项研究为葡萄藤的育种提供了新的思路和工具。
前 言
葡萄基因组具有高度的杂合性,部分原因是由于克隆繁殖过程中遗传变异的积累。这种繁殖方式已持续数千年。例如,二倍体霞多丽(Chardonnay)和赤霞珠(Cabernet Sauvignon)的基因组中包含超过10%的杂合位点,包括单核苷酸多态性(SNPs)、插入缺失(InDels)和结构变异(SVs)10,24,25。尽管常用的参考基因组PN40024经过9代自交后已高度纯合,但与杂合品种相比,它仍缺失了超过10%的基因。在不同品种之间,只有约7%的基因是共享的,而每个品种特有的基因则占约8%。葡萄基因组的高度变异性表明,有必要构建一个泛基因组参考,以纳入存在-缺失变异,提高对基因组变异(包括结构变异)的检测能力,并减少参考偏差。
研究内容
1.构建首个驯化葡萄图形泛参考基因组(Grapepan v.1.0)
研究人员对9个具有代表性的二倍体样品进行了HiFi测序、Hi-C测序和超长纳米孔测序,其中包括1个亚洲特有野生种Vitis retordii的品系以及8个葡萄栽培品种(7个鲜食葡萄和1个酿酒葡萄)。这9个样品产生了18个单倍型,经过填补缺口后达到了T2T水平的组装。基因组大小范围为479.15至539.30 Mb。通过高连续性(>99.9%)、最小切换错误率(<0.05%)和低Hamming错误率(<2.83%)(图1a,b),证实了单倍型组装的质量。使用通用单拷贝同源基因评估基准测试表明,这些单倍型的平均完整性为98.4%。我们使用相同的流程对所有单倍型进行注释,以确保结果的一致性。在18个单倍型中,编码蛋白基因的数量范围为34,536至38,526,每个单倍型的TE序列长度范围为263.86 Mb(54.68%)至312.10 Mb(59.03%)。此外,我们在所有组装结果中鉴定了着丝粒和端粒序列(图1c)。与之前的研究结果一致27,着丝粒的主要重复单元长度为107 bp。总体而言,这18个组装的单倍型及其注释代表了迄今为止所生成的高质量葡萄基因组数据集之一。
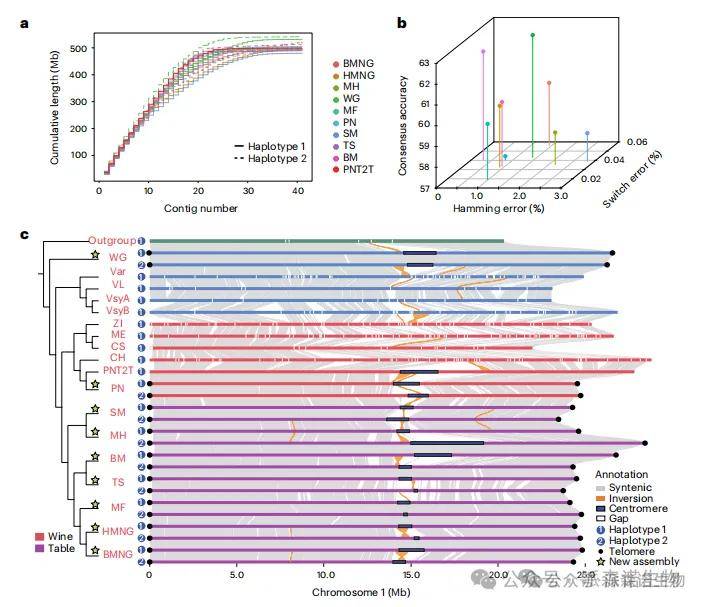 图|T2T基因组组装及Grapepan v.1.0的构建
图|T2T基因组组装及Grapepan v.1.0的构建
2.复杂农艺性状的GWAS及SV的重要性
为了研究结构变异对数量性状的贡献,我们对324个新测序的葡萄品种进行了为期两年(2016年和2017年)的表型调查,共涉及29个性状(图2a)。这324个栽培品种包括106个酿酒葡萄、108个鲜食I类葡萄和110个鲜食II类葡萄。这29个性状包括五个表型类别:果穗(6个)、内容物(8个)、浆果性状(8个)、果实大小(4个)和果皮(3个)(图2a)。以往的研究已利用约6,000个SNP位点对与果实组成和尺寸相关的表型性状进行过GWAS分析36。本研究更大的样本量和全基因组范围内的变异数据,有助于同时对多个性状进行基因组选择。首先,研究人员分析了两年间数量性状之间的相关性,并采用主成分分析(PCA)对表型进行映射(图2b)。在浆果内容物类别中,果糖(Fru)、葡萄糖(Glu)和可溶性固形物含量(SSC)等性状两两之间存在显著的正相关关系(P<<0.001)。此外,不同类别之间的性状也存在相关性。例如,内容物类别的可滴定酸(TAC)与果实大小的四个测量指标之间存在显著的负相关关系:浆果重量(BeWe)、浆果体积(BV)、浆果长度(BL)和浆果宽度(BeWi)。这些相关性可能源于这样一个事实:在果实转色期(veraison)时,酸的合成通常会停止,而这种停止会导致果实继续成熟和膨大过程中酸浓度的稀释。表型之间的这些相关性可能导致一些候选的GWAS位点重叠,并对具有多种理想性状的优质葡萄品种的基因组选择产生潜在影响。
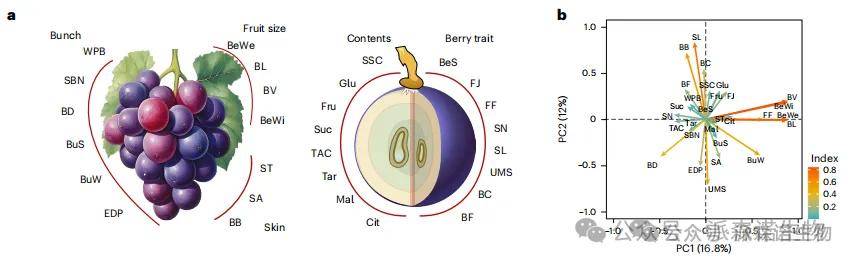 图2 | 不同葡萄群体间29项农艺性状的相关性分析。
图2 | 不同葡萄群体间29项农艺性状的相关性分析。
以往GWAS研究难以同时处理大量品种、多性状及高分辨率数据,且葡萄研究中从未纳入结构变异(SVs)。我们利用Grapepan v.1.0平台对SNP和SVs进行GWAS分析,发现148个基因座与农艺性状相关(136个由SNP检测,12个由SVs捕获),覆盖27.61 Mb基因组区域(~5.58%)。其中26个位点(~17.57%)与已知功能位点重叠。例如,18号染色体上AGL11基因(调控种子发育)与无籽性状相关;BL1位点95bp缺失(PVE 6.31%)与果实长度相关;SN6基因座1.1 kb缺失(PVE 6.08%)与光裂合酶基因相关;Suc1基因座139 bp插入(PVE 6.60%)影响蔗糖含量。PNT2T参考基因组分析显示,91.18%的位点与泛基因组SNP结果一致。
此外,SSC7和BeWi9位点的显著SNP(P值6.05%/5.91%)与浆果宽度相关。局部系统发育树显示栽培葡萄存在选择性清除。SNP 17_6489512和17_6484258分别影响可溶性固形物和浆果宽度。该区域注释出NEPS和NRT1基因家族,其中Vitvi031750和Vitvi031760高表达,Vitvi031756在酿酒葡萄中表达更高。
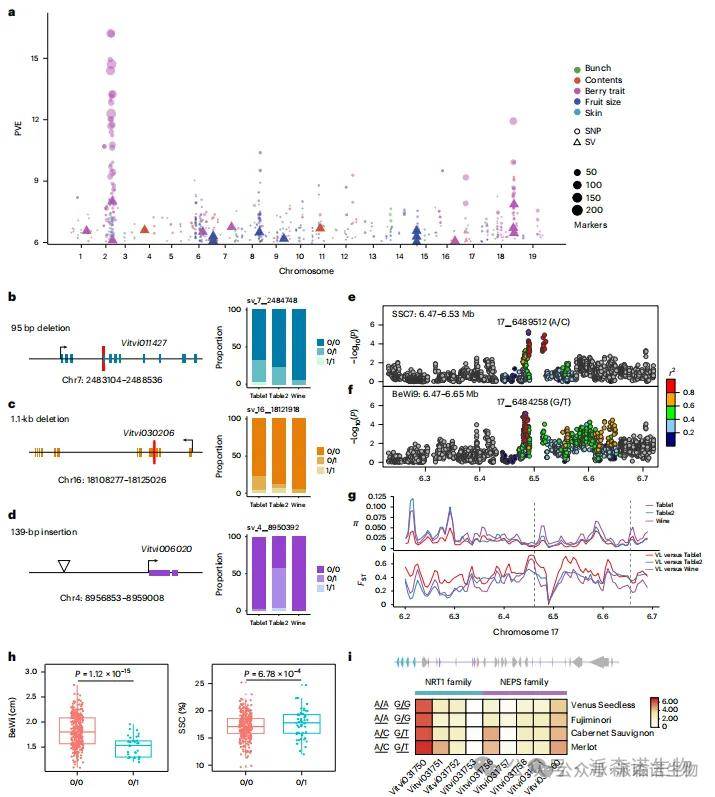 图3|与农艺性状相关的候选基因座及其人工选择的基因组足迹
图3|与农艺性状相关的候选基因座及其人工选择的基因组足迹
4.葡萄农艺性状的分歧选择
通过XP-EHH分析发现,鲜食葡萄组间21.45 Mb(4.4%)基因组区域存在显著分化(P < 0.05),FST分析前5%异常值呈现相似模式。GSEA显示分化区域富集于激素和胁迫响应相关通路。六个GWAS候选位点(BC4、SA1、BeS2、BuW5、FF6、Tar4)位于分化区域,分别调控果实颜色、涩味、形状、穗重、硬度和酒石酸含量。其中,BC4位点含MYB基因簇,FF6位点(PVE 7.35%)使鲜食组果肉硬度显著提升11.7%(P < 0.01)。
酿酒与鲜食葡萄比较发现21.35 Mb分化区域(P < 0.05),GSEA提示糖胺、谷胱甘肽代谢及毒素分解通路富集。45个GWAS位点与果实大小相关,32个位点关联风味代谢物(如总酸、单宁、糖类)。分化选择显著影响BV12、BeWe6等果实大小位点及TAC3、SSC7等代谢位点,表明农艺性状分化与育种目标密切相关。
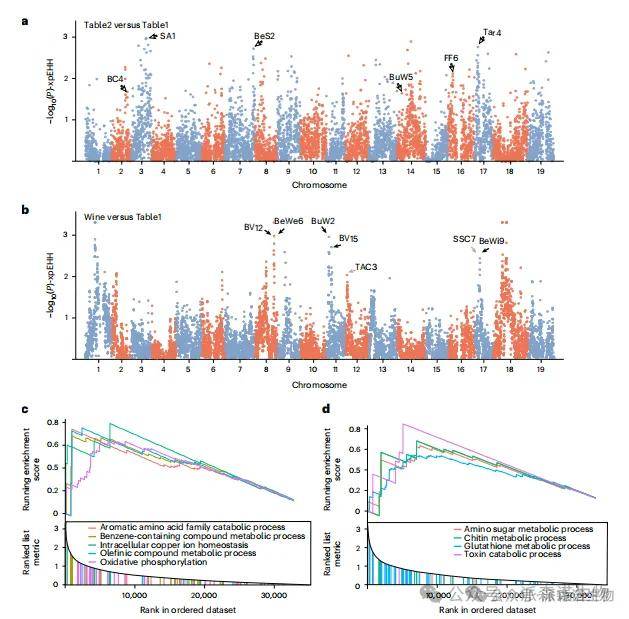 图4|亚群间农艺性状的差异选择
图4|亚群间农艺性状的差异选择
5.SVs提高了葡萄性状的遗传力估计
本研究发现,大多数结构变异(SV)与单核苷酸多态性(SNP)无连锁,且部分GWAS信号区域仅由SV驱动,表明SV对遗传力缺失具有独特贡献。通过LDAK模型分析显示,SV对性状遗传力的贡献(0.5%-86.1%)显著高于SNP(0.01%-52.3%),其中定量性状主要受SNP多基因效应调控,而定性性状更依赖SV大效应。典型案例如BeWi表型(SV解释74.6% vs SNP 0.5%)、SSC性状(SV 35.8% vs SNP 0.6%)以及BL性状(SV引入使遗传力从20.9%提升至64.9%)。特别值得注意的是,Chr7上5.6 kb缺失(SSC2)通过调控异淀粉酶基因Vitvi011368显著影响SSC表型。在育种应用方面,整合SV与SNP的多性状分析显示20.7%的性状存在显著遗传相关(rG=0.41-0.97),基于多基因评分(PGS)的预测准确率平均超过50%,较传统方法提升16%以上,其中BL性状预测准确率高达79.53%。这些发现为葡萄分子设计育种提供了新的理论依据和技术支撑。
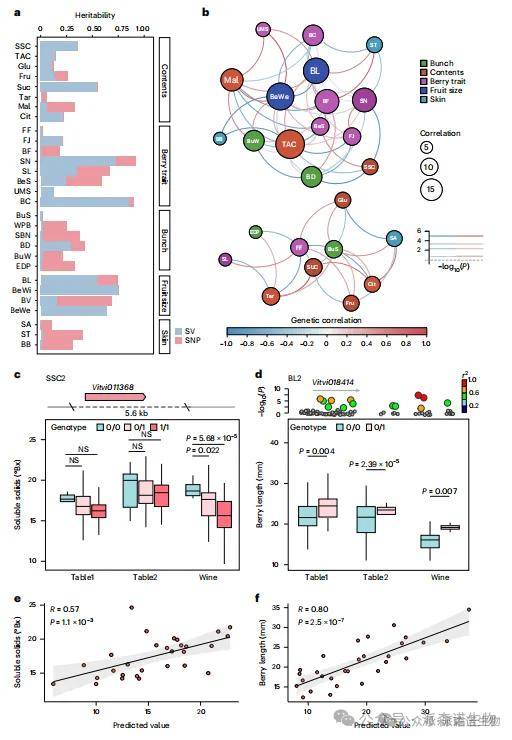 图5|缺失的遗传力、遗传相关性和农艺性状的基因组预测
图5|缺失的遗传力、遗传相关性和农艺性状的基因组预测
总 结
研究人员成功地构建了一个包含29个完整基因家族的葡萄基因组学全基因组图谱,并发现其中许多基因与果实品质相关。通过对这些基因进行进一步的功能分析,研究人员得出了一些关于葡萄生长发育的重要结论,这对未来的葡萄育种和生产具有重要的指导意义。同时,该研究也证明了全基因组测序技术和高精度组装技术在植物基因组学研究中的重要性和应用前景。
参考文献:Liu Z, Wang N, Su Y, et al. Grapevine pangenome facilitates trait genetics and genomic breeding. Nat Genet. 2024 Dec;56(12):2804-2814. doi: 10.1038/s41588-024-01967-5. Epub 2024 Nov 4.




















