
文章题目:Metagenome-informed metaproteomics of the human gut microbiome, host, and dietary exposome uncovers signatures of health and inflammatory bowel disease
中文标题:人类肠道微生物组、宿主及饮食暴露组的宏基因组辅助宏蛋白质组学研究揭示健康与炎症性肠病的特征标志
发表期刊:Cell
影响因子:42.5
发表时间:2025.01
样本类型:人、小鼠粪便
组学技术:宏基因组测序、宏蛋白组学
研究背景
肠道微生物组、宿主和饮食的复杂相互作用是维持健康或引发疾病的关键。然而,传统研究方法(如宏基因组测序)主要关注微生物的基因组成,无法直接反映其功能活性。蛋白质作为生命活动的直接执行者,其表达水平更能体现微生物和宿主的动态功能状态。炎症性肠病(IBD)等肠道疾病与微生物组失调密切相关,但现有诊断标志物(如钙卫蛋白)敏感性和特异性有限,且缺乏对饮食暴露的客观评估方法。研究开发了一种结合宏基因组信息的宏蛋白质组学方法MIM(metagenome-informed metaproteomics),该方法能够同时量化来自饮食、宿主和微生物的蛋白质,以更好地解析宿主-微生物组-饮食相互作用,并为炎症性肠病(IBD)等肠道微生物组相关疾病的个性化诊疗开辟了新的可能性。
研究思路
1.肠道微生物组与宿主健康的直接功能联系
通过对小鼠和人类的研究,科学家们发现肠道微生物组的功能变化与宿主健康之间存在直接的联系。在炎症性肠病(IBD)患者中,不仅微生物组的组成发生了变化,其功能也出现了特定的“功能失调”,这种失调表现为某些细菌对炎症信号的反应受到抑制。
2.饮食、宿主和微生物组的相互作用
通过分析健康个体和IBD患者的粪便样本,科学家们能够准确地描绘出饮食暴露的情况。他们发现,在IBD患者中,饮食模式与健康个体存在显著差异,这种差异可能与疾病的发生和发展有关。
3.早期疾病标志物的发现
通过对IBD患者和健康对照组的比较分析,研究团队发现了一系列潜在的疾病标志物,这些标志物不仅包括宿主蛋白,还包括微生物蛋白。
研究内容
1. MIM宏蛋白质组工作流程和性能测试
“宏基因组指导下的宏蛋白质组学”(Metagenome-Informed Metaproteomics,MIM)方法主要是利用每次实验样本的宏基因组测序结果构建个体化参考数据库,结合质谱进行蛋白质组检测和定量的分析方法,以此实现对宿主、肠道微生物及膳食来源蛋白的同步、种水平定量分析。简单说,就是先用宏基因组获得菌群信息,再用这些菌的基因信息做参考,结合质谱检测蛋白,这样就能精准知道粪便里宿主、细菌和吃进去的食物都有哪些蛋白被表达出来,从而分析它们之间的相互作用。在健康SPF小鼠中,MIM平均检测到631个宿主蛋白、2656个细菌蛋白和23个膳食蛋白;在人类样本中,分别为363、3746和58个。通过与其他宏蛋白质组学和宏基因组方法的体外、计算模拟及体内对比,MIM在灵敏度、特异性和分类精度上表现更优,能够识别>90%的已知细菌蛋白,且准确度高于97%。
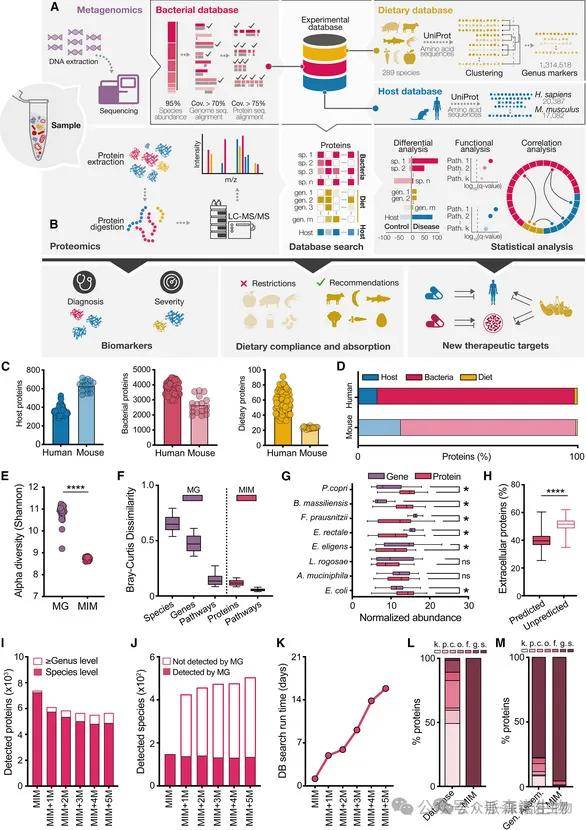
基于宏基因组信息的宏蛋白质组学(MIM)在微生物组研究中的应用
2. MIM 评估人类胃肠道(GIT)中的功能性微生物组-宿主相互作用
通过MIM方法分析了健康人群粪便样本的微生物蛋白质组,与宏基因组相比,MIM检测到更少但功能活跃的蛋白,且样本间变异度更低,提示其更能反映功能状态。MIM发现多数细菌的蛋白丰度与其基因丰度不一致,尤其是分泌蛋白,说明基因难以预测实际蛋白表达。此外,MIM将97%的粪便微生物蛋白精确定位至种水平,而非目标物种的随机蛋白会严重降低分析准确性和效率。在复杂菌群背景下,MIM展现出高于参考数据库和基因组拼接方法的物种解析力。进一步研究了治疗前健康个体和广谱抗生素处理个体从胃到粪便的多部位样本。MIM揭示健康肠道共生菌蛋白在不同部位的特异性表达,例如幽门螺杆菌在胃中特异表达致病岛蛋白,而链球菌糖类代谢蛋白在胃中丰度高于粪便,仅由MIM检测到。抗生素处理导致条件厌氧菌如大肠杆菌和肠球菌蛋白上升,伴随蛋白和种类梯度的消失,并产生个体化的抗药性蛋白谱,这些多无法通过粪便检测得到。
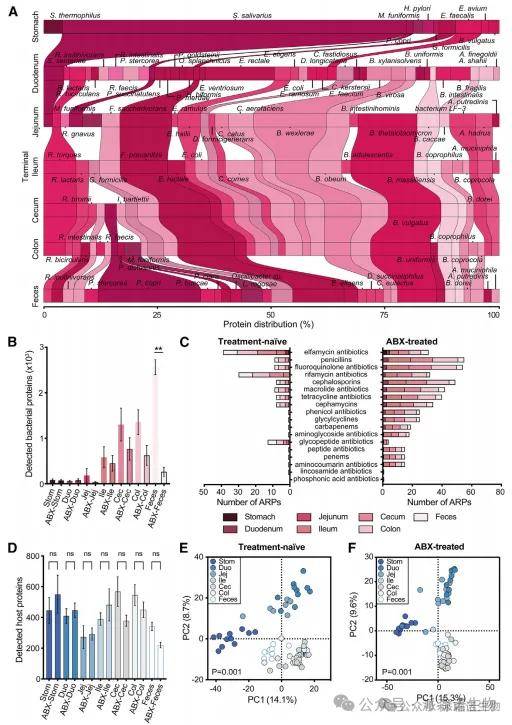
健康与抗生素干扰下的人类宿主-微生物组MIM景观
3.饮食暴露组的蛋白质组定量分析
通过MIM分析,研究不仅能够准确识别饮食来源的蛋白质,为每项分析提供饮食暴露组的定量评估,还展示了其在不同实验设置下的应用潜力。以控制饮食的小鼠为对象利用MIM分析不同饮食小鼠的粪便样本宏蛋白质组。纯酪蛋白饮食小鼠中专一性的检出了α-酪蛋白,氨基酸饮食小鼠检测不到食物来源蛋白信号,豌豆和大米喂食的小鼠中专一性检出了豌豆和水稻蛋白,MIM宏蛋白质组也能准确区分麸质饮食和非麸质饮食的小鼠。作者在人群样本中同样利用MIM宏蛋白质组定量了膳食暴露组。基于MIM的宏蛋白质组营养评估可以准确捕捉基于人群由饮食习惯文化差异驱动的食物暴露差异。来自以色列队列(n=54)和德国人队列(n=100)的基于MIM的饮食暴露确定队列检测到两国居民粪便中对含小麦食物的膳食暴露水平相似。
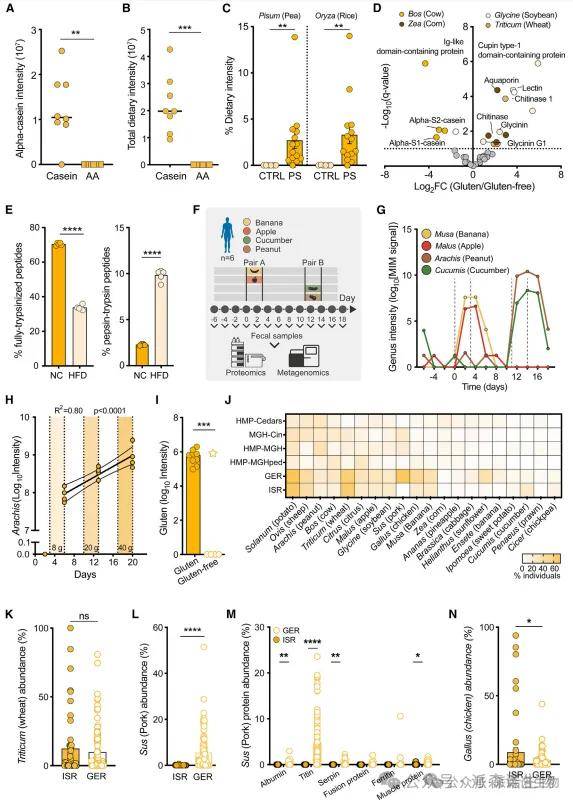
MIM可准确识别小鼠和人类的饮食暴露情况
4. 小鼠肠道自体炎症过程中宿主-共生菌-饮食交叉调控的功能阐明
利用 MIM 来解析在诱导小鼠急性葡聚糖硫酸钠炎症性肠病(IBD)结肠炎模型过程中发生的复杂整体生物相互作用。在 DSS 补充开始后仅 3 天,并且在整个疾病过程中,无论是通过宏基因组还是 MIM 评估,粪便微生物组都与基线相比发生了显著变化。值得注意的是,MIM 识别出这种急性炎症环境中的两种不同的失调模式:一种是“组成性失调”,表现为共生菌丰度的变化,特征为物种特异性蛋白的单向表达变化。这些变化可以通过宏基因组检测到(粉色)或仅通过 MIM 检测到(红色)。此外,还观察到一种“功能性失调”,它表现为某些物种中一组蛋白质的变化,大多数“管家蛋白”和相关的宏基因组信号保持不变(深红色)。这种与小鼠肠道自体炎症相关的功能性失调,特别表现为多种细菌蛋白的减少,主要来自 毛螺菌属(Lachnospiraceae) 物种。
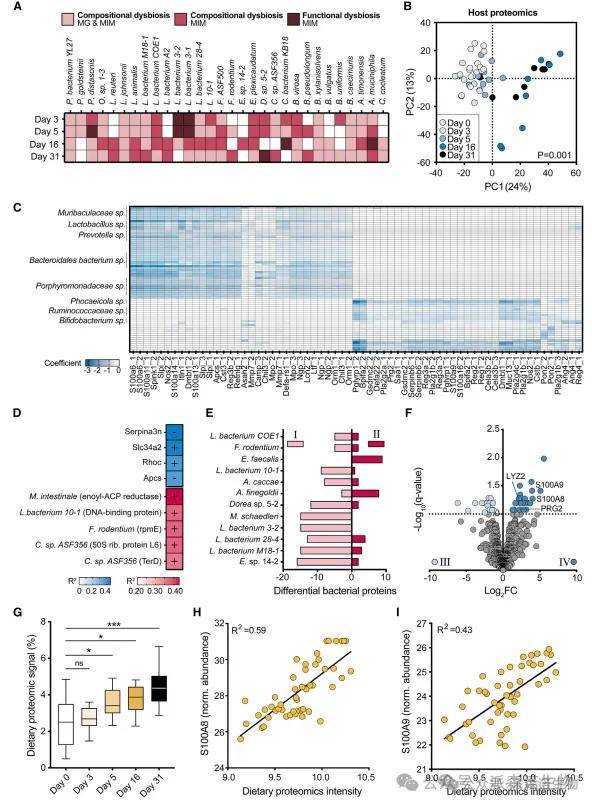
小鼠患肠道炎症过程中宿主-微生物组饮食 MIM 变化
5.MIM揭示了人类IBD中宿主-微生物群功能程序的改变
首先评估了以色列一组参与者的粪便MIM谱,这些参与者包括一组新发病的儿童克罗恩病患者(ISR-pCD,n = 26)和与其年龄及性别匹配的健康对照组(ISR-HC,n = 28)。ISR-pCD患者的粪便细菌MIM谱与ISR-HC组显著不同,其中包括多个细菌代谢途径的失调,这些途径在ISR-pCD患者中被发现发生了变化,例如普通拟杆菌(Bacteroides vulgatus)中的糖基降解和鞘氨醇脂代谢。有趣的是,几种细菌蛋白与临床参数显著相关,包括C反应蛋白(C-reactive protein, CRP)、钙卫蛋白和儿童克罗恩病活动指数(PCDAI)。此外,粪便中ISR-pCD宿主的MIM特征与ISR-HC组明显不同,并且与免疫系统,特别是中性粒细胞功能和细菌防御相关。
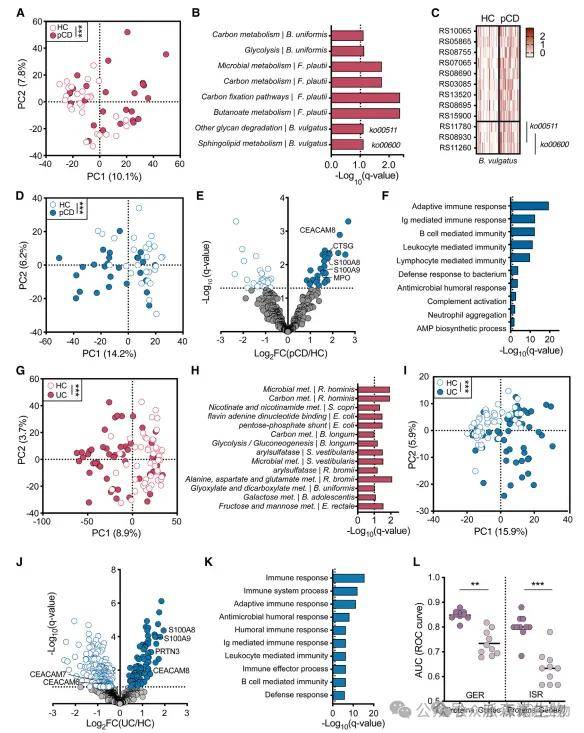
人类IBD中的宿主-微生物群膳食肠道MIM谱
6.粪便饮食蛋白质组评估揭示人类IBD中的与炎症相关的蛋白质吸收不良
在新发ISR-pCD患儿中,开始实施专一性肠内营养(EEN)后,粪便中膳食蛋白显著减少,个体间差异明显,且粪便膳食蛋白数量与炎症标志物S100A9呈正相关,表明MIM可同时评估饮食依从性与炎症状态。进一步分析HMP-Cin(Cincinnati Medical Center的 HMP队列)和HMP-MGH(Massachusetts General Hospital的HMP 队列)(人类微生物组计划(Human Microbiome Project, HMP)两个IBD队列,发现仅在HMP-Cin中,IBD患者粪便膳食蛋白总量显著升高,尤其是伴有上消化道受累的Crohn患者,提示MIM可作为非侵入性手段推测小肠吸收功能异常,具有潜在临床价值。
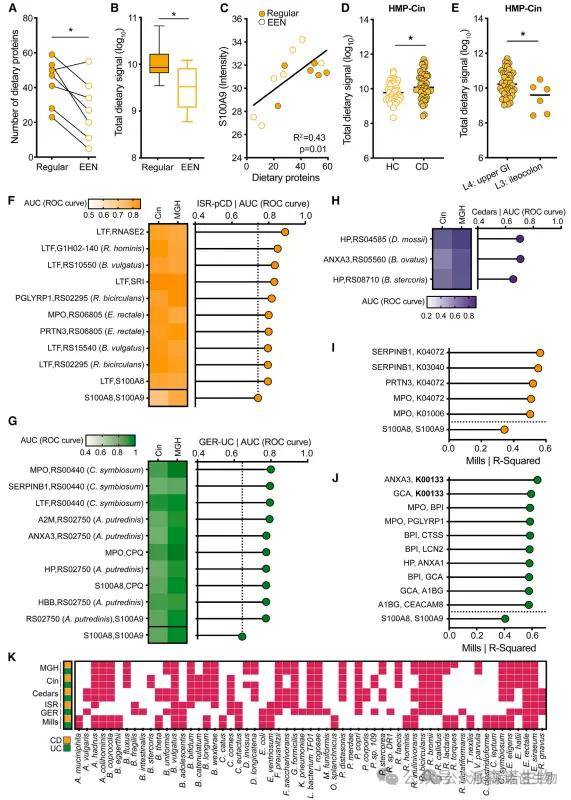
基于MIM的非侵入性炎症性肠病生物标志物
研究结论
该研究采用了基于宏基因组的宏蛋白质组学(metagenome-informed metaproteomics, MIM)方法,在小鼠和人类模型中,以非侵入性方式探索了共生菌和病原菌定植、营养调控以及抗生素干扰期间的物种水平的微生物群-宿主相互作用。同时,粪便MIM精确表征了多种临床和饮食环境中的营养暴露情况。在小鼠自身炎症模型和人类炎症性肠病(inflammatory bowel disease, IBD)研究中,MIM鉴定出“组成性菌群失调(compositional dysbiosis)”及随之而来的物种特异性“功能性菌群失调(functional dysbiosis)”,该失调由共生菌对炎症宿主信号的抑制性响应所驱动。更重要的是,同时进行的粪便营养MIM分析可用于确定IBD相关的饮食模式、饮食治疗依从性以及小肠消化异常。整体而言,平行化的饮食-细菌-宿主MIM分析能够从功能角度揭示跨界互作网络如何塑造胃肠生态,并为与微生物群相关的疾病提供个性化的诊断和治疗见解。





















