

微生物的世界不再神秘,宏蛋白组学正以其独特视角解读食品中的蛋白质动态,为食品安全、品质控制和营养健康带来革命性突破。
在过去,食品科学大多依赖传统微生物培养和化学分析方法,难以全面洞察复杂食品基质中的微生物功能活性及其对健康的影响。如今,宏蛋白组学能够直接鉴定和量化食品生态系统中的蛋白质组成,揭示微生物的实际功能活动。
通过整合宏基因组学与蛋白质组学信息,宏蛋白组学不仅告诉我们食品中“有什么”微生物,更能解答这些微生物“在做什么”的关键问题,成为食品科学研究中不可或缺的强大工具。
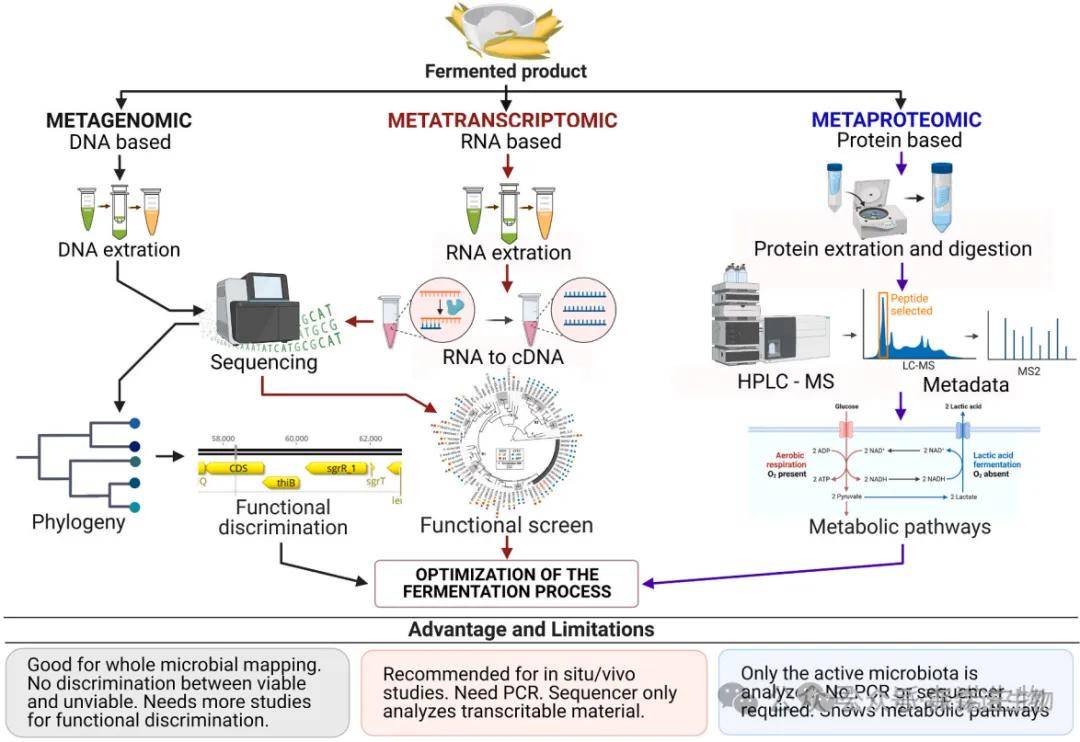
主要组学技术的示意图以及使用它们表征微生物群和优化发酵过程的可能性
(https://doi.org/10.1016/j.tifs.2022.09.017)
针对食品领域热点的典型分析方案
1. 发酵过程监控
研究思路:
解析发酵过程中微生物群落结构、功能动态变化及风味形成机制。
数据挖掘与分析方案:
时间序列分析:将不同时间点的样品作为一组,进行差异分析和趋势分析(聚类)。
关联分析:将特定酶(如酯酶、淀粉酶)的丰度与关键风味物质(代谢组数据)的含量进行相关性分析(如Spearman相关)。
功能聚焦:重点注释CAZy(碳水化合物代谢)、氨基酸代谢(KEGG)、次级代谢产物合成通路。
2. 食品安全与溯源
研究思路:
快速鉴别致病菌、腐败菌及其活性毒力因子,追溯污染源。
数据挖掘与分析方案:
标志物筛选:通过差异分析,筛选在污染样本中显著高丰度的物种特异性蛋白和毒力因子(如毒素、黏附素)。
机器学习:使用随机森林等算法,基于蛋白丰度 profile 构建分类模型,预测样品状态(如新鲜/腐败)。
3. 宿主-微生物互作
研究思路:
研究益生菌/发酵食品如何通过调节肠道菌群影响健康。
数据挖掘与分析方案:
整合分析:(核心)将食用食品后的动物/人肠道宏蛋白组数据与宿主蛋白(如免疫标志物、肠道屏障蛋白)表达量进行关联。
通路富集分析:特别关注微生物的短链脂肪酸合成通路、宿主炎症反应通路(如NF-κB信号通路)的富集情况。
核心数据挖掘步骤与思路
1. 数据预处理与质控
这是所有分析的基础,确保数据质量。
思路:对原始质谱数据进行校准、去噪,并评估数据质量。
2. 蛋白质鉴定与定量
思路:将质谱图谱与蛋白质数据库进行匹配,鉴定肽段和蛋白质,并进行相对或绝对定量。
数据库选择(关键!):
使用来自同一样本的宏基因组测序数据组装、预测的基因集作为搜索数据库。这最大程度减少了数据库偏差。如没有宏基因组数据,也可以混合一个样本进行宏基因组测序做为数据库,建议测序数据量20G-30G,可确保数据库的完整性。
3.物种与功能注释
思路:回答“谁在那里?”(物种)和“他们在做什么?”(功能)的问题。
物种溯源:
使用Unipept (https://unipept.ugent.be) 工具,通过鉴定到的肽段序列(通常是种属特异性肽段)回溯到最低分类等级(种/属)。
输出:各样本在门、纲、目、科、属、种水平的物种丰度表格。
功能注释 (Functional Annotation):
工具:将鉴定到的蛋白质序列与各类功能数据库进行比对。
常用数据库:
GO (Gene Ontology):描述蛋白质的生物学过程(BP)、分子功能(MF)和细胞组分(CC)。
KEGG (Kyoto Encyclopedia of Genes and Genomes):映射到代谢通路、酶促反应。
COG (Clusters of Orthologous Groups) :蛋白功能分类。
CAZy (Carbohydrate-Active Enzymes):针对碳水化合物活性酶,在食品发酵、肠道微生物研究中非常重要。
CARD (Comprehensive Antibiotic Resistance Database):针对抗生素抗性基因分析。
VFDB (Virulence Factors of Pathogenic Bacteria):致病菌毒力因子注释数据库。
4. 差异丰度分析
思路:比较不同组别(如:发酵第1天 vs 第7天、健康样品 vs 污染样品)之间蛋白质/物种/功能的丰度差异,寻找标志物。
统计检验:为了分析不同组间具有表达差异的蛋白质,对实验数据进一步进行差异筛选,其中 Fold change 用来评估某一蛋白在样品间的表达水平变化倍数;经计算的 P value 展现样品间差异的显著程度。该项目差异筛选条件为 Fold change > 1.5 倍且 P value < 0.05。
5. 高级整合与可视化分析
思路:挖掘数据中更深层次的模式、关联和网络关系。
多组学整合:
与宏基因组整合:比较宏蛋白组(表达层面)和宏基因组(潜能层面)的差异,揭示哪些功能基因被 actively expressed。例如,基因组里有某个毒素基因,但蛋白组未检测到,则风险较低。
与代谢组整合:将显著变化的代谢物与差异表达的酶/通路进行关联,构建“酶-代谢物”网络,解释代谢表型的驱动因素。
网络分析 (Network Analysis):
共现网络 (Co-occurrence Network):分析不同物种或蛋白质之间的共生或互斥关系。
物种-功能关联网络:将物种信息与它们所执行的功能联系起来,揭示功能冗余或特定功能的关键贡献者。
派森诺生物全新的宏蛋白组学分析内容包含五大分析模块、四十二项分析内容。除了常规的质控分析、差异蛋白及功能分析,重点是增加了物种和微生物功能相关的分析内容,从蛋白的维度解析微生物的多样性和功能。
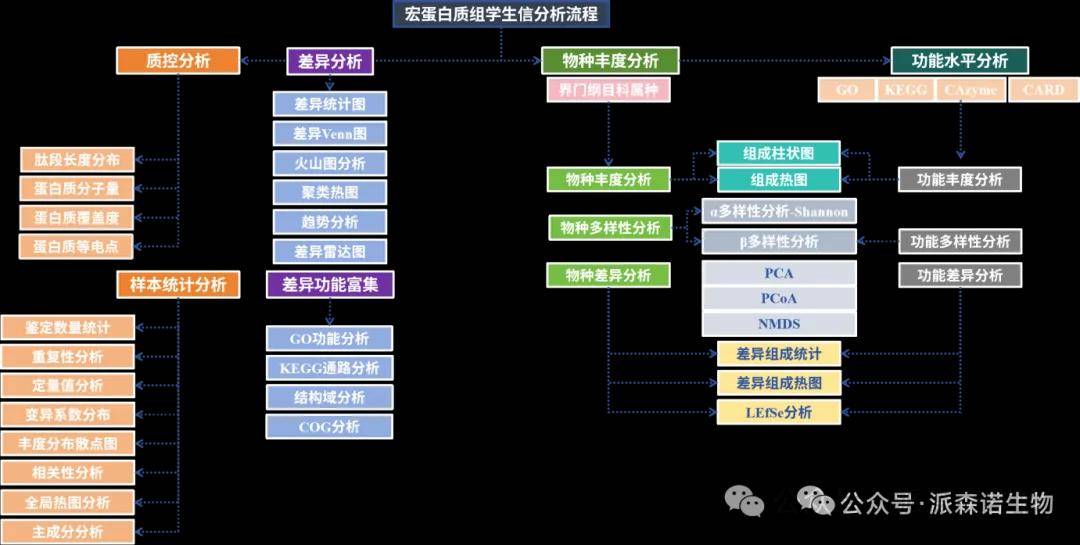
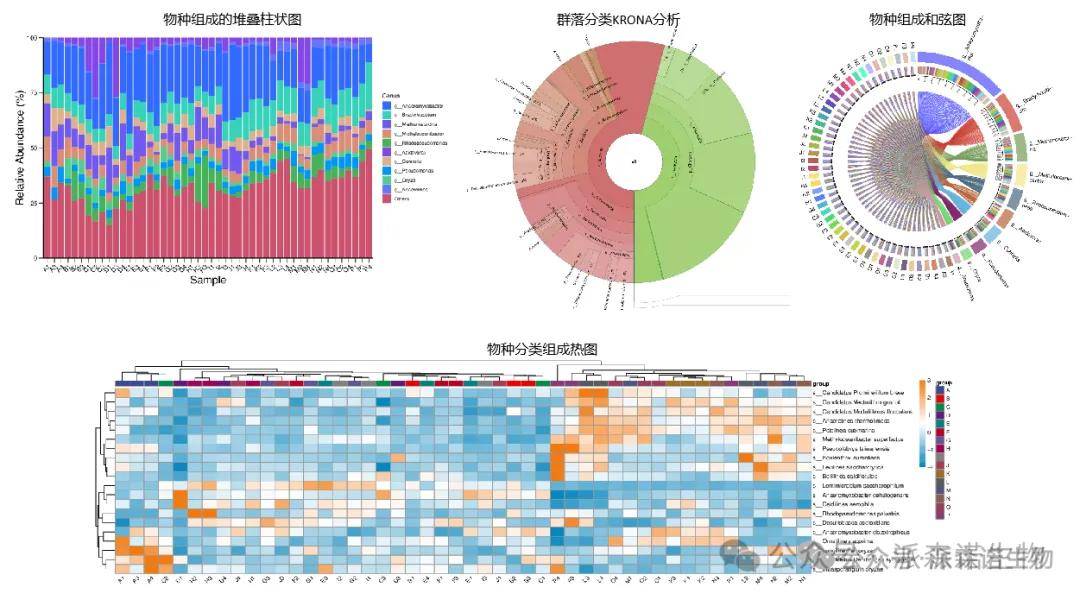
宏蛋白组分析报告继承了前面常规蛋白组升级的内容,不仅框架更加合理,可读性、便捷性等各方面都有了提升,与常规蛋白组的不同之处,就是微生物物种注释分析的升级,我们采用了全新的物种注释算法,使得基于宏蛋白的物种注释更加的科学、准确,同时增加了很多优美的图片展示,包括物种组成柱形图、和弦图、热图、群落KRONA分析等多种形式。
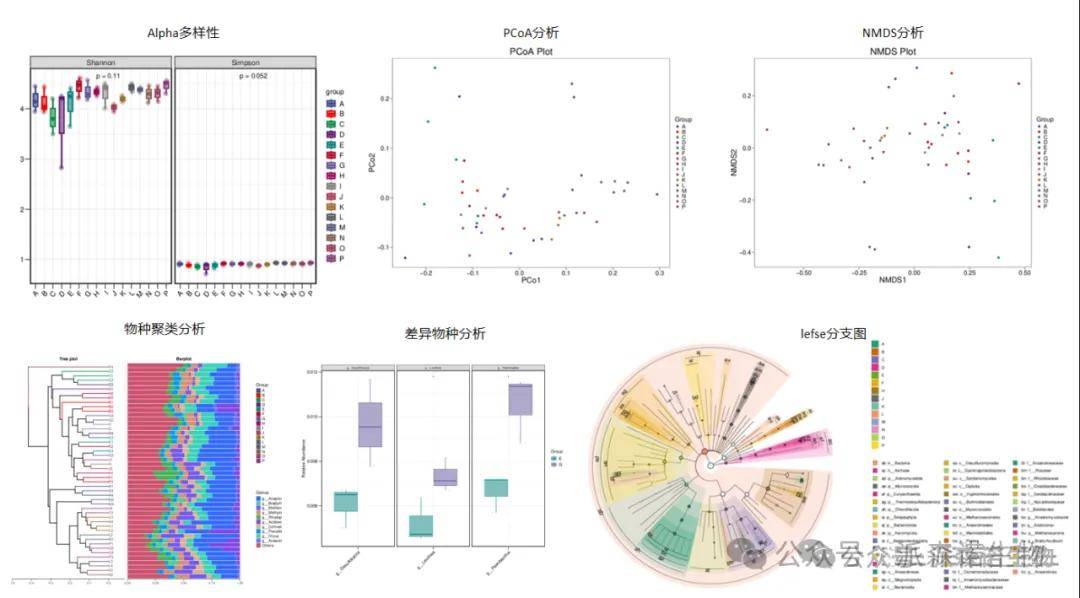
宏蛋白组的功能分析不仅涵盖了常规的GO、KEGG和结构域注释,还特别新增了与微生物密切相关的功能分析模块,例如抗生素抗性基因分析(CARD)、糖基水解酶分析(CAZy)以及毒力因子分析(VFD)等。这些新增分析内容显著拓展了宏蛋白组研究视角,为深入探索微生物群落的抗性机制、代谢潜能和毒力特征提供了更全面的数据支持。
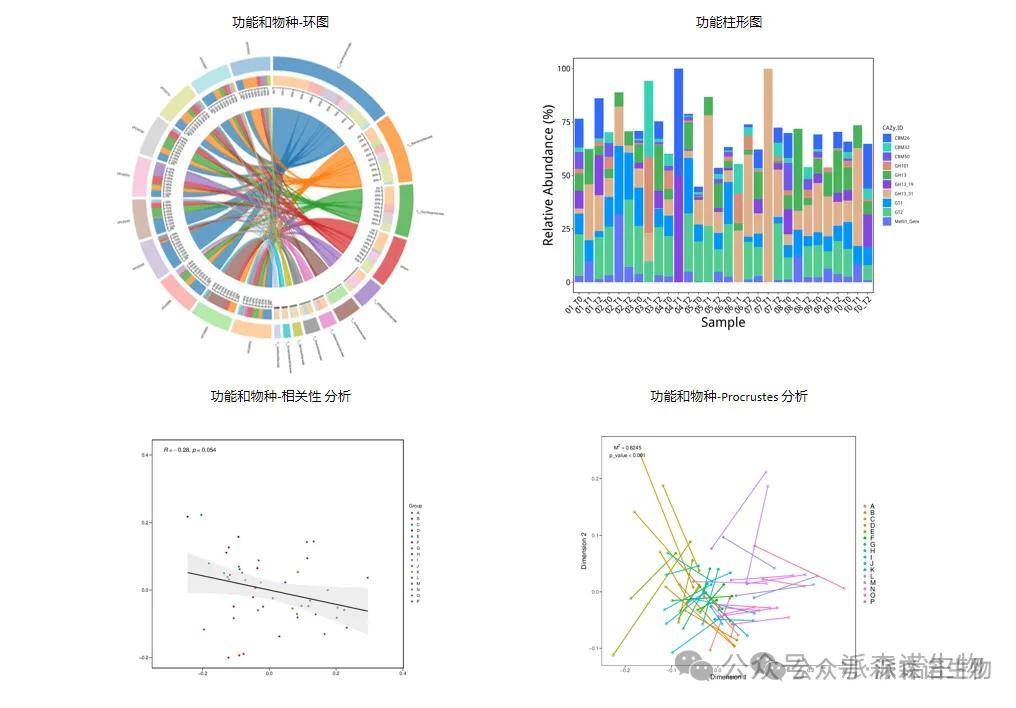
同时也增加了一些新的图形和展现形式,基于微生物不同分类水平、不同功能注释模块等,提供了更加多样化的分析内容。
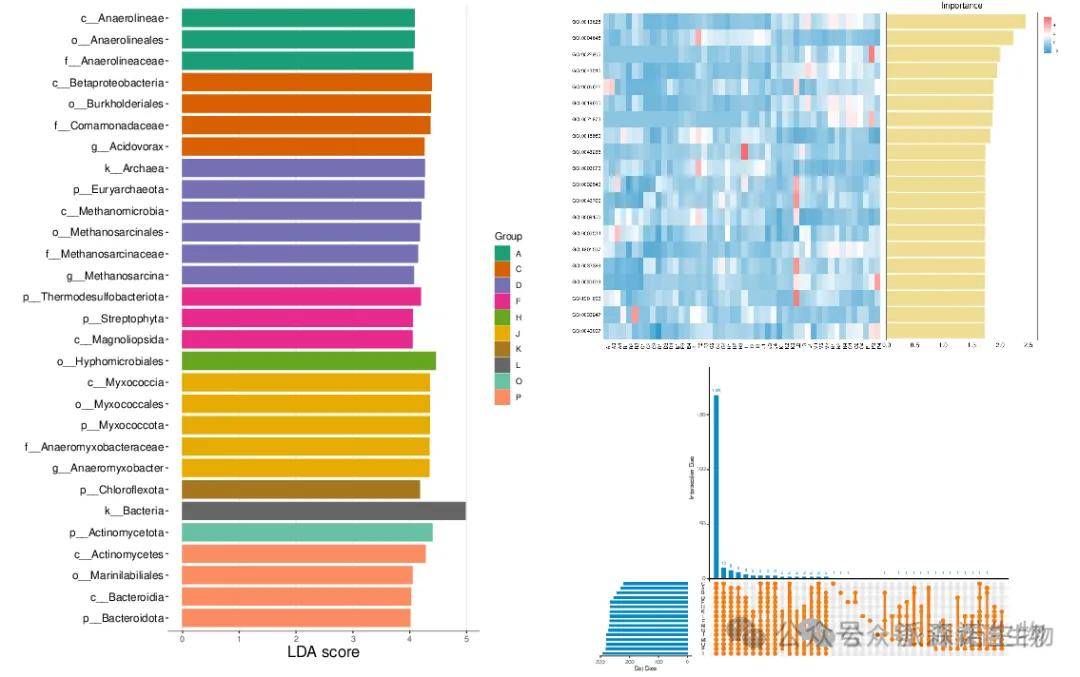
总 结
宏蛋白组的数据挖掘是一个假设驱动的过程。从简单的物种功能列表,到复杂的多组学网络,其深度取决于你的科学问题。始终牢记:生物学重复至关重要,它是所有统计学意义的基础。对于食品这种复杂基质,基于宏基因组的定制数据库是获得可靠结果的黄金标准。




















