
2025-09-16

文章亮点
研究采用了灵活的稀疏部分二元交叉设计,能够有效地提高杂交育种效率。
利用了基因组关联分析、基因组选择和亲本系通用性评价等多种方法,全面评估了杂交系的遗传特性。
成功筛选出多个具有高产潜力的杂交系,为玉米杂交育种提供了新思路和技术手段。
文章信息
英文题目:SPDC-HG: An accelerator of genomic hybrid breeding in maize
中文题目:SPDC-HG:玉米基因组杂交育种的加速器
发表期刊: Plant Biotechnology Journal
影响因子:10.5/Q1
发表时间:2025年2月4日
涉及组学:GWAS、GS。
技术路线
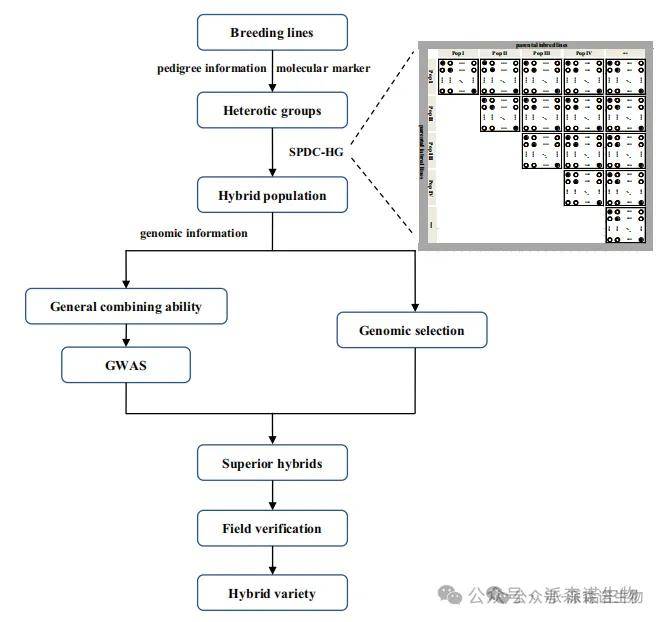
摘 要
将多种现代育种技术应用于玉米研究始终面临挑战。本研究通过采用由266个不同异源组系培育的945个玉米杂交种组成的灵活稀疏部分双交设计,旨在解决这一难题。研究整合了全基因组关联分析、亲本系基因组选择及亲本系基因组评估,加速单交杂交种的育成进程。研究发现7-25个稳定单核苷酸多态性(SNP)与九项产量相关性状的综合结合能力(GCAs)存在显著关联。基于玉米基因组数据库(maizeGDB)和美国国家生物技术信息中心(NCBI)数据库,通过至少三种统计方法检测到的显著SNP筛选出264个候选基因,并进行功能注释。基于这些GCA SNP构建的标记集显著提升了杂交种在所有性状上的预测准确性。参与前100名和后100名杂交种培育的亲本系,其GCA估计值始终稳定排在表型排名的前100和后100位,验证了预测的准确性。此外,通过贝叶斯贝斯(BayesB)、广义双交系育种预测模型(GBLUP)和岭回归(LASSO)筛选出的前100名杂交组合,在田间验证中平均穗重较后100名杂交组合105.4–108.6%提升,显示出显著的选择增益。值得注意的是,在百大杂交种中,A017/A037和A037/A169这两个品种,各自包含六个优良基因型,分别被中国国家作物品种审定委员会注册为苏育161和通育1701。这些成果不仅彰显了基因组选育技术的有效性,更为玉米基因组杂交育种的发展提供了重要参考价值。
前言
玉米是粮食、动物饲料和生物能源生产的重要作物,目前其全球产量已稳居谷物首位。杂交育种仍然是提高玉米产量潜力的最有效策略。目前的GCA研究仍受限于测验材料数量不足,从而影响了GCA估算的准确性。因此,开发一种全新的方案以精准评估和解析GCA,已成为当务之急。此外,如何确定所选自交系之间的最佳杂交组合,也是杂交育种过程中面临的另一大挑战。由于实际操作中无法在田间逐一评估所有可能的杂交组合,这一难题亟待解决。
资源有限的情况下,基因组选择(GS)是一种极具前景的解决方案,能够有效应对这些挑战。它利用全基因组标记,在表型测定之前即可预测个体的遗传价值。尤其在杂交育种中,GS优势明显:通过分析亲本基因型,可推断出杂交后代的基因型,从而显著加快育种进程、降低育种成本。在玉米育种中,同时整合多种现代育种技术一直是一项巨大挑战。为此,本研究提出了一种全新的遗传交配设计——基于稀疏部分双列杂交(SPDC)的不同高世代(HG)群体间的交配方案,并简称为SPDC-HG。依据这一设计,研究人员选取了来自五个不同高世代的266个自交系,共组配出945个杂交种,即SPDC-HG群体。借助该群体,研究人员开展了全基因组关联分析(GWAS)、基因组选择(GS)以及亲本自交系的基因组评估联合分析,旨在加速育种进程,培育出更优的杂交种。
研究内容
1.SPDC-HG群体的表现
利用Admixture分析、邻接法构建的系统发育树以及系谱信息,对266份自交系的群体结构进行了研究(图1a,b)。经综合分析,这266份自交系被划分为5个高度分化类群(HG)。群体结构分析表明,所选的266个自交系具有广泛的遗传基础。此外,研究人员采用主成分分析(PCA)展示了5个杂优群(HG)的划分结果(图1c)。前三个主成分有效地将这5个杂优群区分开来,进一步验证了其分类的可靠性。基于不同杂优群间的稀疏部分双列杂交,构建了一个SPDC-HG群体(图1d)。然而,由于这些自交系受到复杂环境因素及花期不一致的影响,并未完全按照SPDC-HG设计完成全部杂交。尽管如此,研究人员仍成功获得了945个杂交组合,作为训练集使用,其中不包括正反交配的组合。这些杂交组合中,有772个是由来自不同杂优群的两个亲本杂交而成,另有173个则是同一杂优群内亲本间的杂交(图1e)。
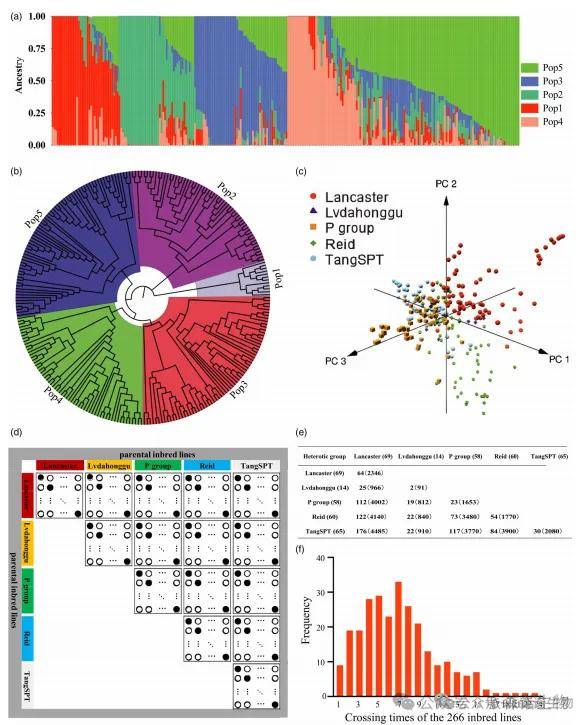
图1 SPDC-HG种群的发展
2.玉米杂交品种的GS和田间验证
基于266个自交系训练群体,研究者采用交叉验证的方式评估了BayesB、GBLUP和LASSO三种GS方法对穗重(EW)、穗粒重(EGW)、穗行数(ERN)、行粒数(KNR)、穗粗(ED)、穗长(EL)、轴粗(CD)、株高(PH)和穗位高(EH)共9个性状的预测效果。结果显示,不同GS方法间的预测准确性差异较小,而不同性状间的预测准确性则存在显著差异,其预测准确性的范围为0.473~0.795。研究人员进一步对266个亲本3,5245个潜在杂交种进行了穗重预测,并筛选出穗重最高的100个(top100)和最低的100个(bottom100)杂交组合进行田间验证。BayesB、GBLUP和LASSO三种方法田间验证的top100杂交种比bottom100杂交种的穗重分别高出108.3%、105.4%和108.6%(图2)。此外,在田间验证的top100杂交组合中,有21个组合的穗重超过对照品种苏玉29,其中17个组合的穗重超过对照品种5%以上。
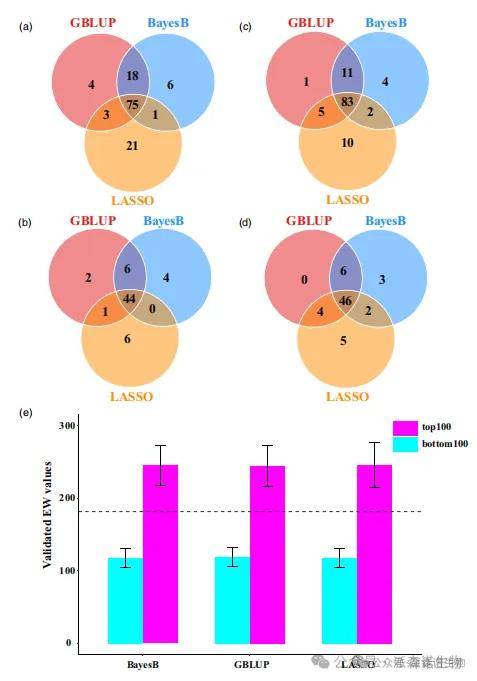
图2 | 田间试验验证的三种GS方法的优选增益。
3.GCA的准确估计
随着分子生物学技术的发展,育种专家可通过基因型-性状关联分析(GP)精准估算基因型-性状相关系数(GCA)。本研究基于SPDC-HG群体基因组数据,采用线性混合模型对可能杂交组合的表型进行预测,成功计算出266个近交系的精确GCA值。各性状GCA值均呈正态分布,均值接近零且变幅较宽,群体间差异显著(P<0.05)(图3)。
P组在GCAEW、GCAEGW、GCAERN、GCAKNR和GCAPH等性状上表现突出,显示出优异的产量和穗型育种潜力。Reid组在GCAEW、GCAEGW、GCAERN和GCAKNR上也表现较好,其中ED和CD的GCA值最高,但GCAPH和GCAEH为负值,表明该组在增产的同时可降低株高和穗位高。Lancaster组的GCAEL最高、GCAEH最低,其余性状GCA值中等。Lvdahonggu组在GCAED和GCAEH上最优,但其他性状GCA值较低或为负,显示其虽能改良株高和穗位高,却缺乏增产潜力。TangSPT组的GCAEW和GCAEGW最低。
为评估GCA预测准确性,采用BayesB、GBLUP和LASSO对九个性状进行交叉验证。结果显示,GCACD的预测准确率最高(0.827、0.833、0.778),GCAKNR最低(0.644、0.658、0.522)。GBLUP在所有性状中表现最优,其中对GCAKNR的预测显著优于BayesB和LASSO。其余性状上,GBLUP与BayesB准确率接近,且均高于LASSO。例如在GCAEW中,GBLUP和BayesB分别为0.734和0.730,比LASSO高7.5%以上。
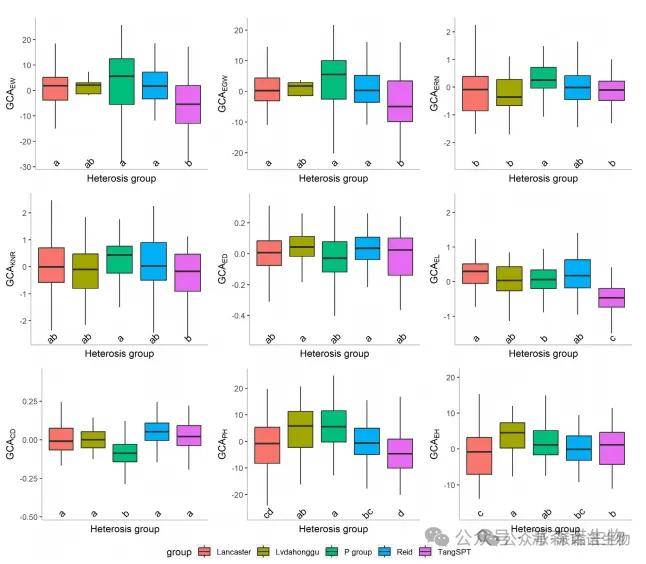
图3|根据不同杂交群对9个产量相关性状进行分类的GCA表型分布。
4.产量相关性状的GWAS及其对应的GCA
本研究采用五种多基因座分析方法(包括Blink、FarmCPU、FASTmrMLM、FASTmrEMMA和ISIS EM-BLASSO)对产量相关性状及其对应的基因组协同分析(GCA)值进行全基因组关联研究(GWAS),共鉴定出630个显著SNP(图4)。此外,产量相关性状共鉴定出299个显著SNP(图4),其中EW有35个,EGW有27个,ERN有39个,KNR有26个,ED有44个,EL有36个,CD有45个,PH有40个,EH有34个。为降低统计方法对关联分析的影响,本研究将至少通过两种不同统计方法鉴定的SNP视为稳定SNP。结果显示:GCAEW、GCAEGW、GCAERN、GCAKNR、GCAED、GCAEL、GCACD、GCAPH和GCAEH分别有7、16、13、14、14、10、25、13和15个稳定SNP显著相关。此外,EW、EGW、ERN、KNR、ED、EL、CD、PH和EH分别有6、5、9、1、4、5、16、3和6个稳定SNP显著相关。
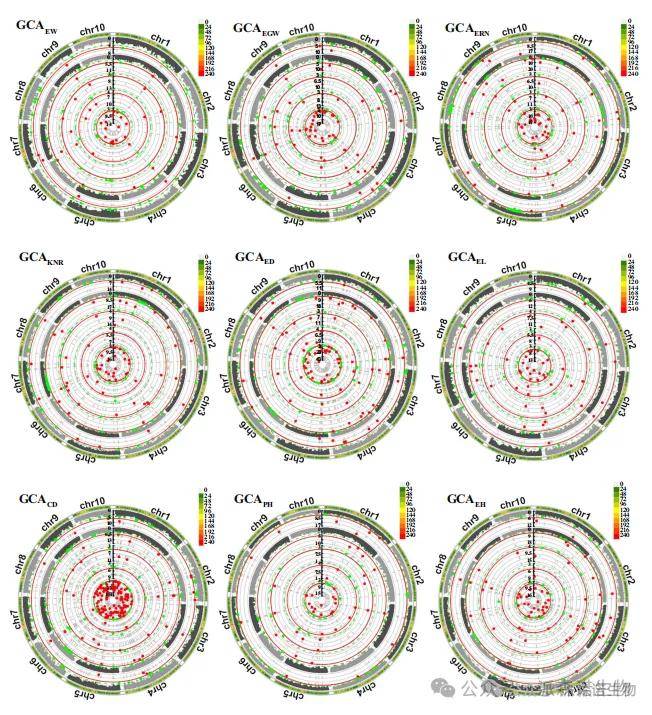
图4|使用五种方法获得的九个产量相关性状的GCA曼哈顿图。
为了进一步分析这些相关SNP在杂交预测中的作用,研究人员从266个自交系中选取了187个构建新的杂交群体。共成功杂交259个,以验证九组标记集的预测准确性,这组标记集包含多种类型的SNP(图5)。全SNP标记集包含所有SNP标记。GCA_SNP1包括通过至少一种统计方法检测到的显著GCA SNP,而随机GCA_SNP1是与GCA_SNP1规模相当的随机选择SNP集合。GCA_SNP2由至少两种统计方法检测到的显著GCA SNP组成,随机GCA_SNP2则是相同规模的随机选择SNP集合。Trait_SNP1包括通过至少一种统计方法检测到的性状本身显著SNP,而随机Trait_SNP1是与Trait_SNP1数量匹配的随机选择SNP集合。Trait_SNP2包含通过至少两种方法检测到的性状本身显著SNP,而随机Trait_SNP2是相同规模的随机选择SNP集合。结果表明,使用这些标记集评估的九个产量相关性状在杂交体中的预测准确性呈现出相似趋势。GCA_SNP2和GCA_SNP1对九个性状的预测准确性显著高于其他标记集,而随机Trait_SNP2的预测准确性最低。例如,EW的预测准确率GCA_SNP1和GCA_SNP2分别为0.733和0.744,相比所有SNP分别提升了8.4%和10.1%。相比之下,与所有SNP相比,随机GCA_SNP1和随机GCA_SNP2的预测准确率分别小幅下降了0.6%和4.4%。Trait_SNP1和Trait_SNP2的预测准确率分别为0.696和0.685,相比所有SNP分别提高了3.0%和1.3%。相反,随机Trait_SNP1和随机Trait_SNP2的预测准确率相比所有SNP分别下降了1.3%和10.4%。
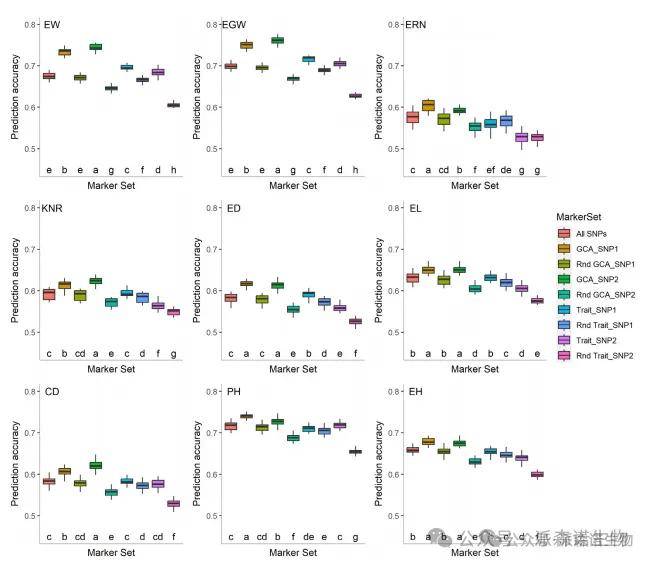
图5|利用9个不同的SNP标记数据集预测玉米杂交种中9个产量相关性状的准确性。
5.EW与相应GCA之间的关系
如图6b所示,位于前100名和后100名杂交种中的自交系分别用红色和金色垂直线表示。前100名和后100名杂交种中的自交系估计的GCAEW值始终分别位于顶端和底端,这表明GCA的估算效果相当显著。准确估算GCA至关重要,因为高GCA值的杂交亲本能显著提高培育出优质杂交种的概率。反之,低GCA值的亲本则容易导致杂交种质量欠佳。此外,前100名杂交种涉及50个亲本自交系,其中包括DH382、H2671、A489和S181等广受欢迎品种的亲本。最后,对前100名和后100名杂交种中与GCAEW显著SNP位点相关的优质基因型积累分析显示,通过遗传选择(GS)筛选的后100名杂交种几乎没有有利基因型的积累,而前100名杂交种则能广泛聚集优质基因型(图6a)。例如,前100名中的杂交种A345/A351、A017/A037、A037/A169和A345/A438,每个都含有六个优质基因型。值得注意的是,A017/A037和A037/A169分别被国家作物品种审定委员会注册为苏育161和通育1701。方差分析显示,具有不同数量优良基因型的杂交种其环境育性(EW)存在显著差异(P < 0.01),且随着优良基因型数量的增加呈现出上升趋势(图6c)。
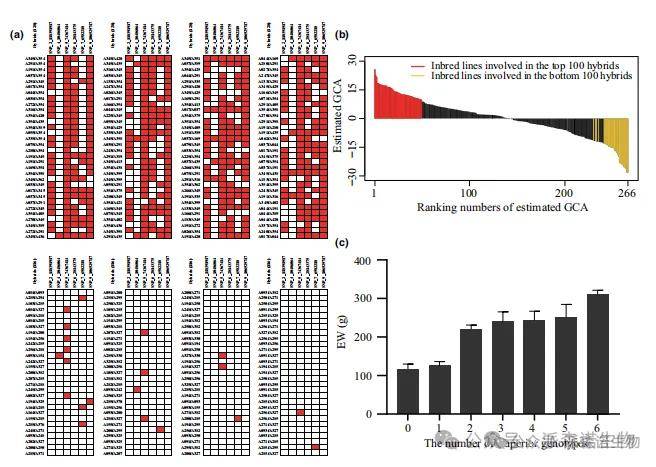
图6|优势基因型的性能。
总 结
通过应用灵活的稀疏部分双列杂交设计,结合全基因组关联分析、基因组选择和亲本单倍型评估等技术手段,可以有效提高玉米杂交育种过程中的基因型鉴定和预测准确率。实验结果表明,该方法能够显著提高杂交组合的产量表现,特别是在穗重和穗粒重这两个重要性状方面,一些特定的杂交模式表现出了更高的平均值和更强的杂种优势。因此,这种方法对于加速玉米杂交育种进程,优化杂交组合的选择具有重要意义。
参考文献:Zhang Z, Wang X, Zhang Y, et al. SPDC-HG: An accelerator of genomic hybrid breeding in maize. Plant Biotechnol J. 2025 May;23(5):1847-1861. doi: 10.1111/pbi.70011.