
1、什么叫做染色质可及性
染色质可及性,也被称为染色质开放程度,是反映染色质转录活性的重要指标,对基因表达调控、表观遗传学、生长发育以及疾病研究都具有重要意义。染色质可及性由核小体及其他结合因子在染色质上的占据情况和拓扑结构决定。细胞中的染色质结构处于动态变化中,受到细胞类型、生理条件和外界刺激的影响,表现出不同的结构和状态,这些变化正是染色质可及性的体现。
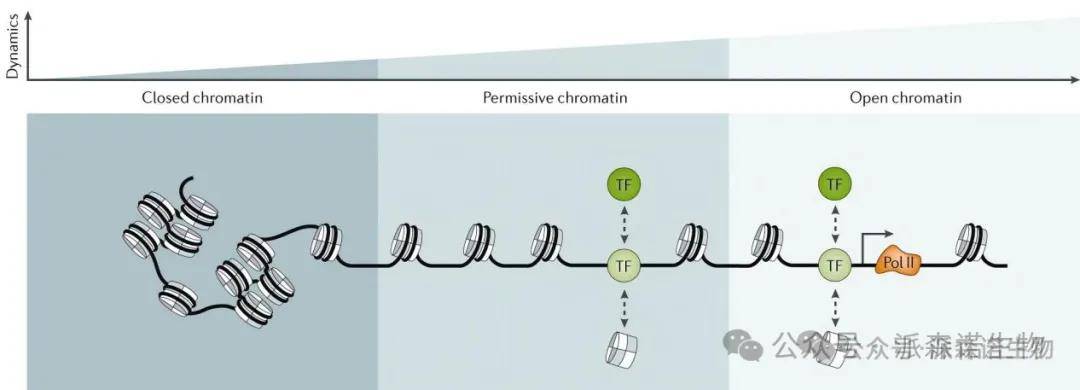
基因组中染色质状态(Klemm et al., 2019)
2、ATAC-seq检测染色质可及性的原理
ATAC-seq(Assay for Transposase-Accessible Chromatin using sequencing)是一种高通量测序技术,用于分析染色质的开放区域。该方法利用改造的Tn5转座酶,靶向插入基因组中开放的染色质区域,即未被核小体紧密包裹的DNA,同时实现DNA片段化和测序接头的标记。经转座酶处理的DNA片段通过PCR扩增构建测序文库并进行高通量测序。ATAC-seq能够绘制全基因组染色质可及性图谱,揭示细胞类型特异的表观遗传调控机制,广泛应用于发育生物学、疾病研究及药物靶点筛选等领域。
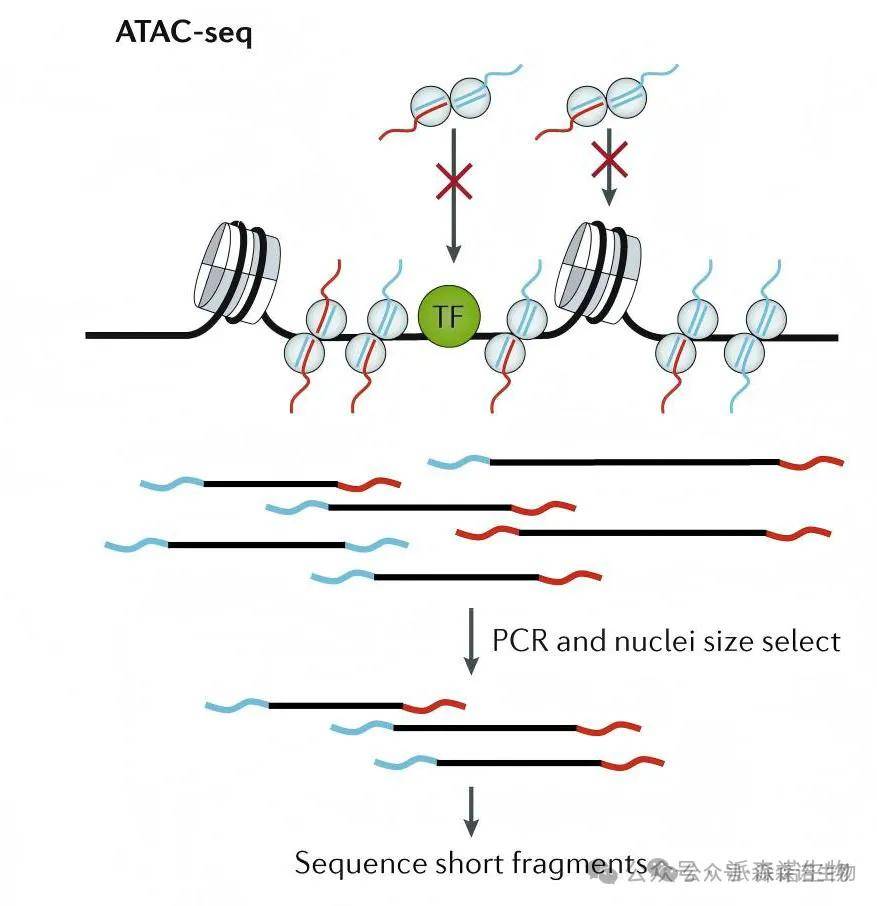
ATAC-seq技术原理
3、技术优势
高灵敏度:ATAC-seq可检测微量基因组中可及染色质区域,确保在样本资源有限时获得有效结果。
低样本需求:所需细胞数较少,特别适合稀有细胞样本,优于其他染色质可及性测序技术。
高通量:利用高通量测序,ATAC-seq可同时分析数百万个片段,提供全面的基因组数据。
直接检测:通过标记开放染色质区域,ATAC-seq直接反映基因组可及性,无需额外步骤。
流程快速:相较于传统方法,ATAC-seq实验流程简化,显著缩短样本处理时间。
低偏差:使用转座酶标记,使ATAC-seq相较于其他方法具有更少的偏差,更准确捕获染色质可及性。
高分辨率:提供较高的基因组分辨率,精确标记和定位开放染色质区域。
4、ATAC-seq与ChIP-seq的异同点
研究焦点:
ChIP-seq:在实验前明确选择特定转录因子,研究者需对某一表观遗传机制有初步假设,设计相应抗体进行ChIP实验,以验证该转录因子与DNA的相互作用。
ATAC-seq:不针对特定转录因子,而是在全基因组范围内评估染色质的开放程度,揭示潜在的蛋白质结合位点,而非验证已知相互作用。
实验目的:
ChIP-seq:旨在确认特定转录因子与DNA的结合,并解析其在特定生物过程中的作用。
ATAC-seq:用于筛选潜在的调控因子,以获取全基因组的开放染色质信息,为后续研究提供基础。
技术原理:
ChIP-seq:通过免疫沉淀技术捕获特定转录因子结合的DNA片段,随后进行测序。
ATAC-seq:利用转座酶直接标记开放染色质区域,获得全基因组的可及性图谱。
5、ATAC-seq应用范围
核小体定位
鉴定重要转录因子
识别启动子区域、潜在的增强子或沉默子
绘制染色质开放性图谱
寻找转录因子调控的靶基因
6、ATAC-seq应用实例

英文标题:Identification and functional characterization of conserved cis-regulatory elements responsible for early fruit development in cucurbit crops
中文标题:保守顺式调控元件的鉴定及功能特征研究:揭示葫芦科作物早期果实发育的调控机制
研究对象:葫芦科作物的果实
测序策略:ATAC-seq、RNA-seq
技术路线:
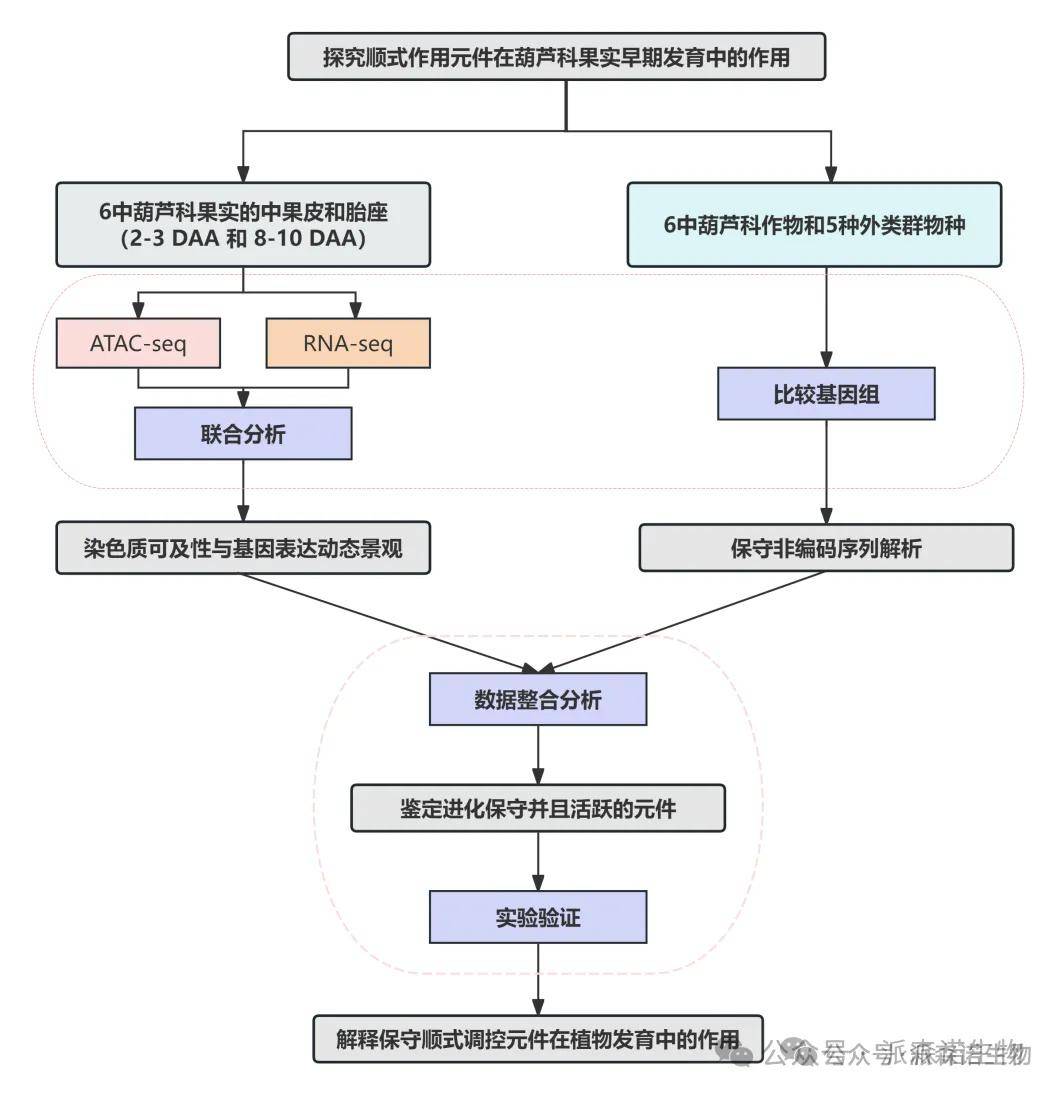
关键结果:
本研究探讨了保守顺式调控元件(CREs)在葫芦科作物早期果实发育中的作用及其进化历史。通过比较基因组学,鉴定出6种葫芦科作物和5种外类群物种中的392,438个保守非编码序列(CNSs),其中82,756个为葫芦科特有。
结合ATAC-seq和RNA-seq分析,定位了20,865至43,204个可及染色质区域(ACRs),这些区域与基因表达正相关,并表现出组织和阶段特异性。整合分析发现4,431个共线同源CNSs与ACRs重叠,其中1,687个为葫芦科特有,推测它们通过调控757个相邻基因参与早期果实发育。
CRISPR突变实验验证了靶向NAC1和EXT-like基因附近的葫芦科特有CNSs后,基因表达模式及果实形状、细胞尺寸显著变化,直接证明了这些CNSs的调控功能。本研究为葫芦科作物遗传改良提供了关键靶点,并揭示CREs在植物发育与进化中的重要性。
组学关键结果:
1.鉴定保守的非编码序列(CNSs)
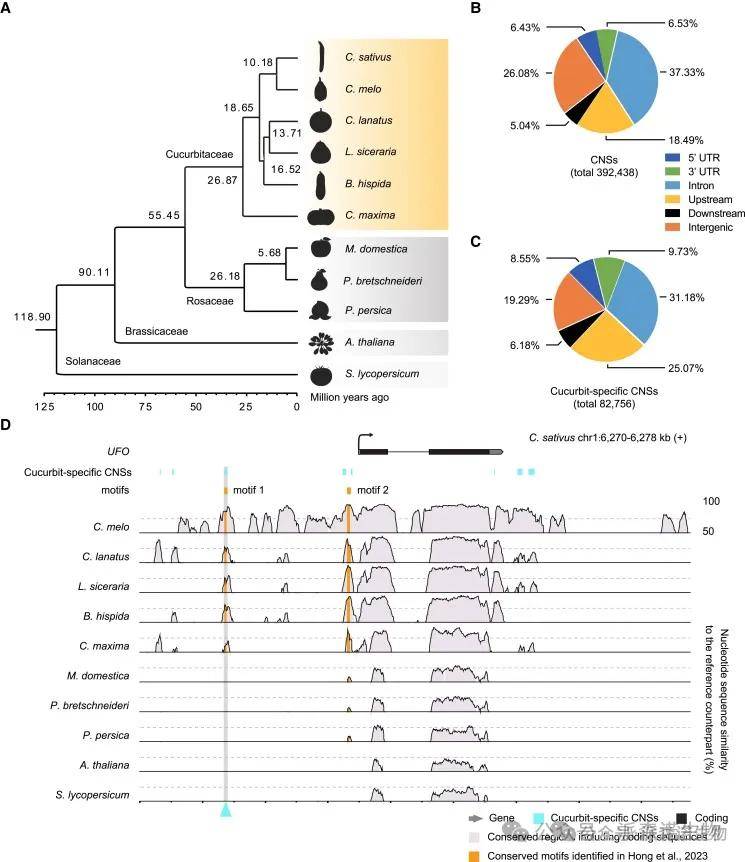
保守CNSs的鉴定与特征分析
2.葫芦科作物早期果实发育过程中可及染色质区域(ACRs)的鉴定
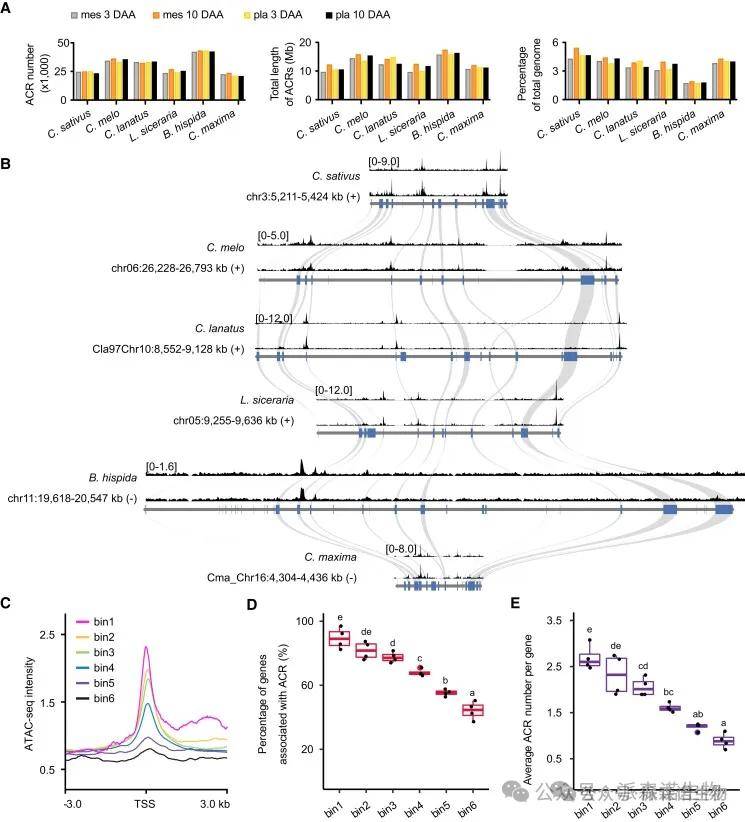
六种葫芦科作物两个组织在两个关键阶段的ACRs鉴定与特征分析
3.ACRs的进化保守性分析
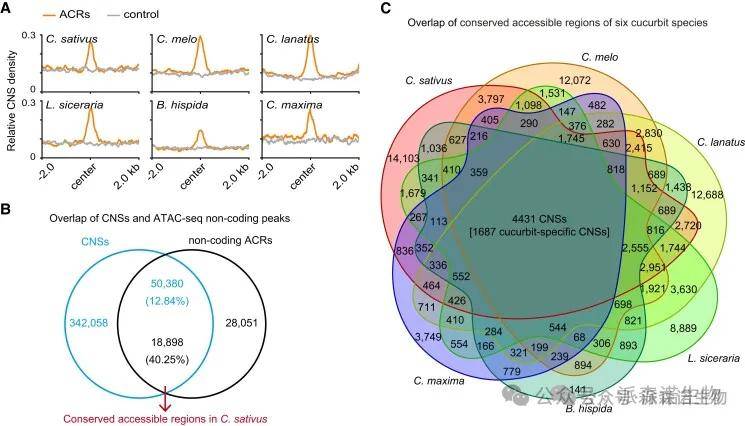
六种葫芦科作物中ACRs的保守性分析
4.染色质可及性保守的CNSs的功能验证
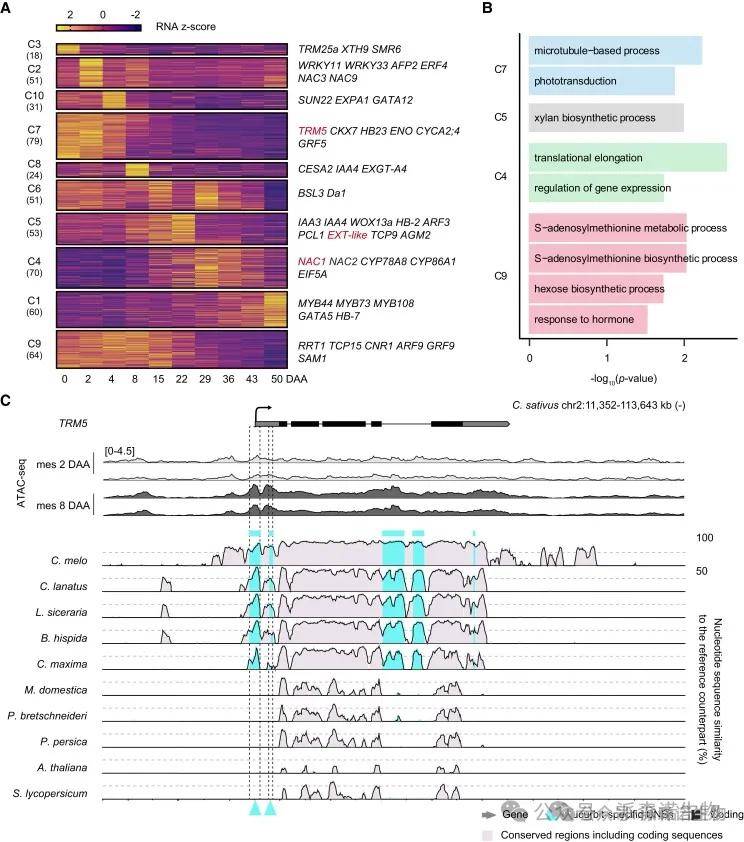
葫芦科特有与ACRs重叠的CNSs潜在靶基因特征分析
参考文献:
1.Klemm SL, Shipony Z, Greenleaf WJ. Chromatin accessibility and the regulatory epigenome. Nat Rev Genet. 2019 Apr;20(4):207-220.
2.Xin H, Liu X, Chai S, Yang X, Li H, Wang B, Xu Y, Lin S, Zhong X, Liu B, Lu Z, Zhang Z. Identification and functional characterization of conserved cis-regulatory elements responsible for early fruit development in cucurbit crops. Plant Cell. 2024 May 29;36(6):2272-2288.




















