
Highlights
构建基因调控网络,从“点”到“面”理解遗传架构——不再孤立地看一个个SNP,而是从系统生物学的角度,将微效的遗传变异整合起来,揭示它们如何协同扰动整个网络从而导致宏观表型的变化。
突破“连锁不平衡”困局,精准定位致病基因——通过多维数据的交叉验证,从一长串候选名单中精准锁定最可能的因果变异和其调控的靶基因。
多维数据的交叉验证和整合——派森诺擅长技术创新与多组学整合,通过GWAS+转录组+表观组学(WGBS/ATAC-seq/ChIP-seq)形成一个完整的证据链,极大地增强了研究发现的可信度和生物学意义。
01、简 介
传统GWAS研究只能找到与性状显著相关的基因组位点(SNPs),但这些位点绝大多数位于非编码区,其生物学功能是未知的。我们不知道它如何、通过谁、在何种条件下影响性状。
多组学联合分析的核心意义就在于,搭建一座从“统计学相关”通往“生物学因果”的桥梁。它系统地解答了以下几个关键问题:
机制是什么?显著SNP是否通过影响基因表达(eQTL)来发挥作用?
目标基因是谁?受影响的基因具体是哪一个?
调控路径如何?表观遗传修饰(如DNA甲基化、组蛋白修饰)如何中介这一调控过程?
细胞上下文是什么?这种调控关系在哪种细胞类型或组织中发生?
02、主要应用
GWAS+转录组+表观多组学的联合分析,其根本意义在于实现了对基因组“代码”的“功能注释”和“机制解读”。它将孤立的统计学信号转化为具有明确生物学背景、细胞上下文和分子路径的完整故事,是后GWAS时代解读复杂性状遗传机制的核心和标准范式。它极大地推动了精准医学和现代农业育种从理论发现向临床和应用实践的转化。


项目文章

期刊:iMeta
影响因子:33.2/Q1
发表单位:兰州大学王维民教授团队
分析内容:群体进化、GWAS、转录组、表观组(ATAC-Seq、CUT&Tag、Hi-C)。
研究路线:
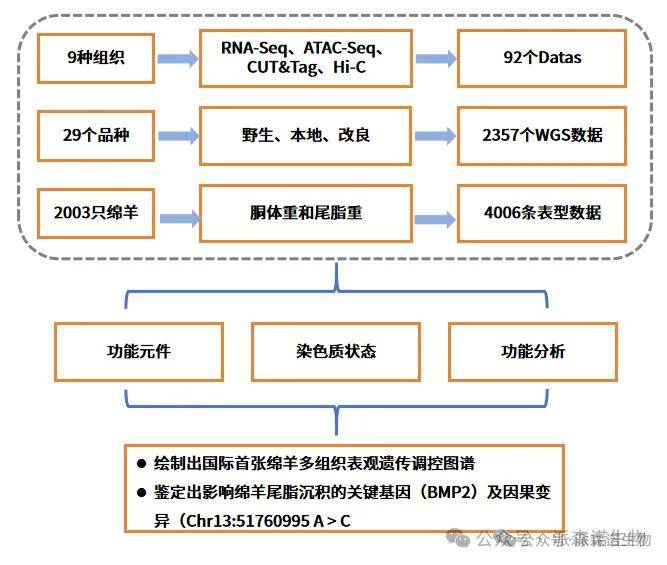
关键结果:
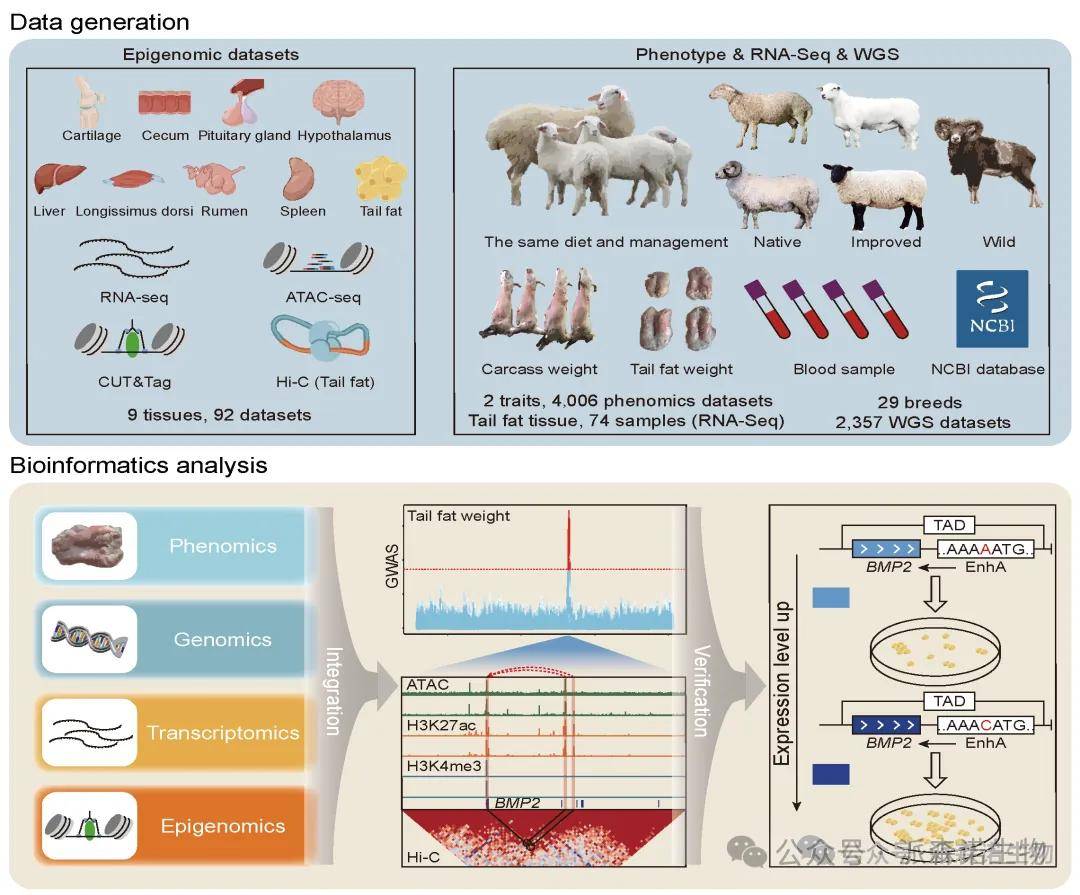
对9个重要组织(软骨、盲肠、垂体、下丘脑、肝脏、背最长肌、瘤胃、脾脏和尾脂)进行了RNA-Seq、ATAC-seq、两种组蛋白修饰(H3K27ac和H3K4me3)的CUT&Tag和Hi-C测序,生成了92个表观基因组数据集。启动子状态(TssA和TssW)在TSS处表现出最高的富集度,并且与其他染色质状态相比,其基因表达平均水平最高。
对来自全球的364只绵羊进行基于最大似然估计的群体结构分析。结果表明,所有个体被分为三个独立的簇:亚洲摩弗伦羊、中国地方绵羊品种和改良品种。剖析绵羊的驯化,计算了野羊和家养绵羊之间的固定指数(FST)、种群扩展单倍型纯合性(XP-EHH)和核苷酸多样性比率(π比率)。共确定了46个候选基因组区域,结果发现TssA、TssW和EnhA区域在受驯化影响的基因组区域中高度富集。
对168只脂尾型绵羊品种和170只瘦尾型绵羊品种进行了基于固定指数(FST)的选择性清除分析,将前5‰的窗口定义为候选基因组区域。结果发现两个信号最强的区域分别位于Chr13(51.15-52.05Mb)和Chr15(3.67-4.50Mb)。
为缩小影响绵羊尾脂沉积的基因组区域,本研究对2,003只具有尾脂重量和尾脂相对重(尾脂重/胴体重)的大规模队列进行GWAS分析。根据-log10(0.05/总SNP)=8.63的阈值,在Chr7、Chr9和Chr13染色体上筛选出984个和1086个与尾脂重量和尾脂相对重显著相关的SNPs位点,其中981个SNPs与尾脂重量和尾脂相对重均显著相关,且位于Chr13染色体的受选择区域。
为确定与尾脂沉积相关的候选因果变异并探讨其对尾脂沉积的作用,本研究整合了不同尾型的选择性清除分析结果、尾脂重性状的GWAS结果和绵羊尾部脂肪组织的表观基因组数据(即ATAC-seq、H3K27ac、H3K4me3和Hi-C数据)。结果仅发现两个SNPs(Chr13:51760995A>C和Chr13:51825895G>A)位于OCR和H3K27ac峰。这两个SNPs也位于与骨形态发生蛋白2(BMP2)、LOC101117953和LOC101118027基因相同的拓扑关联域(TAD)中。结果表明这两个SNPs可能与绵羊尾脂沉积密切相关。
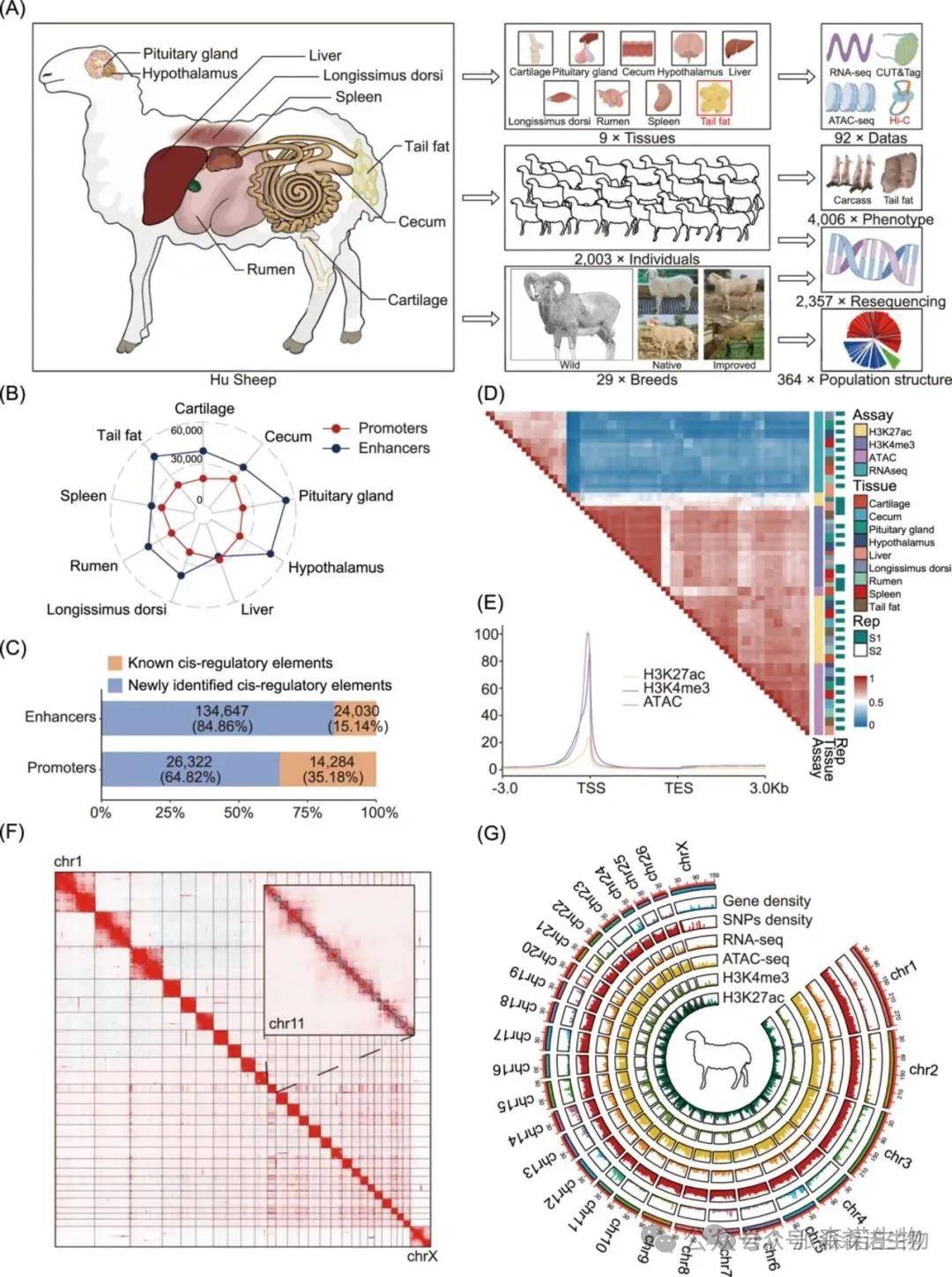
图1 绵羊多组织表观基因组图谱
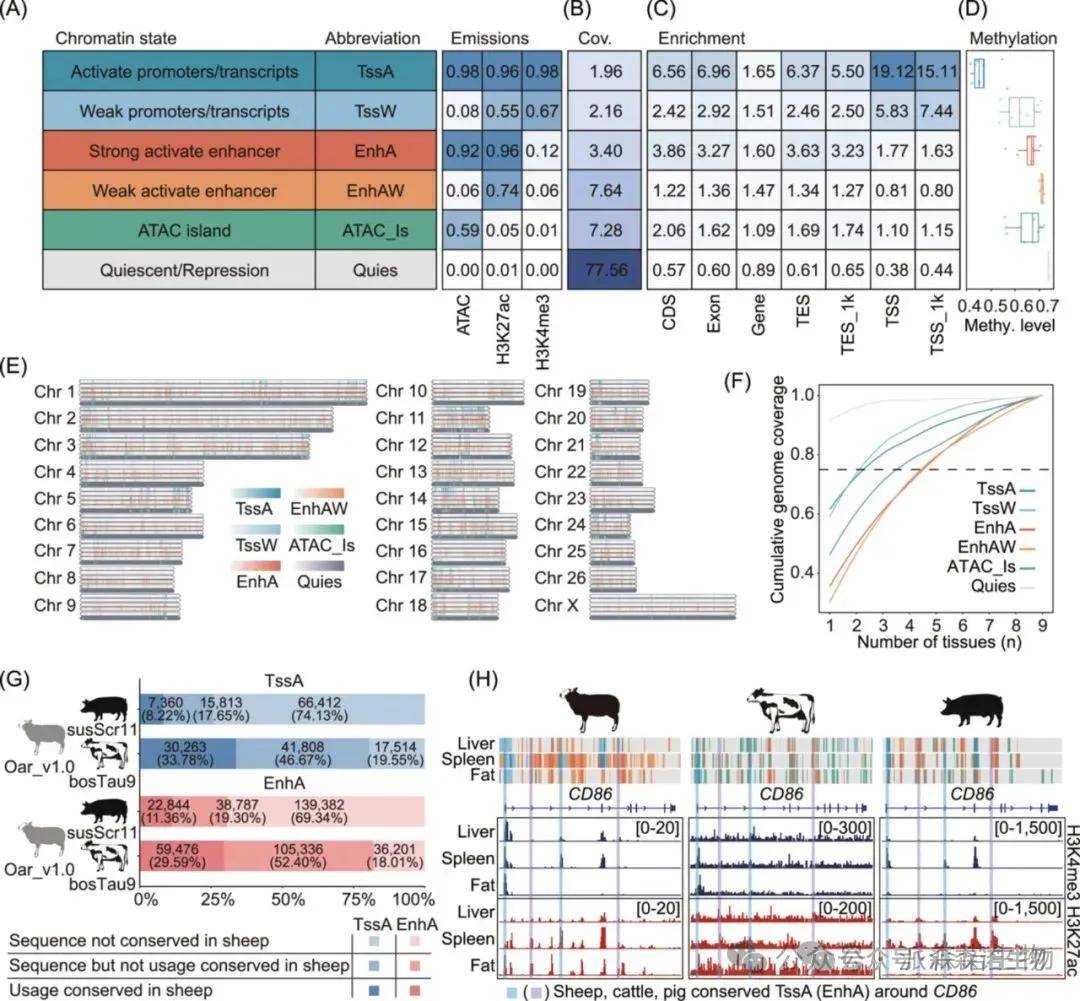
图2 九种组织的全基因组染色质状态图
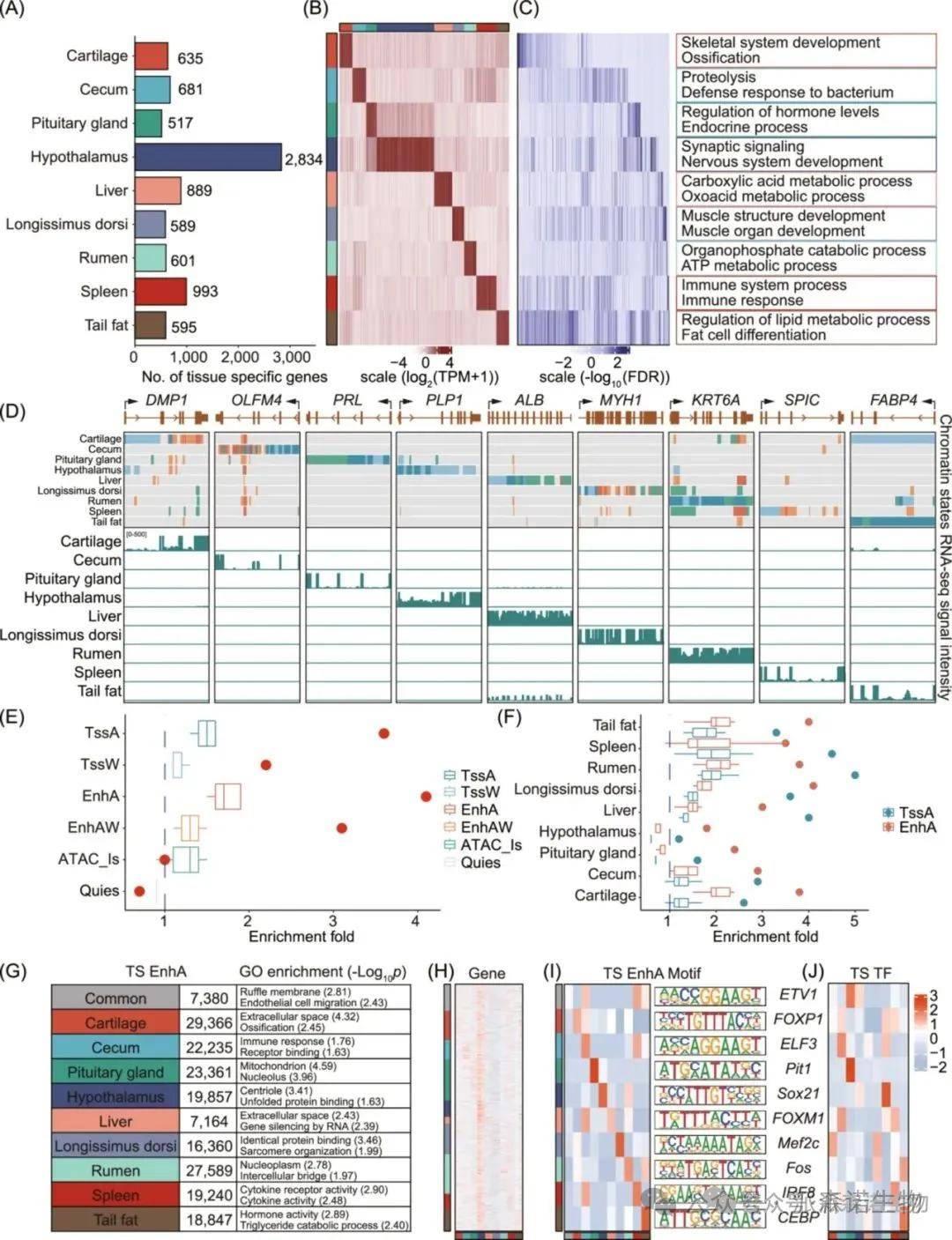
图3 组织特异性基因和染色质状态的功能表征
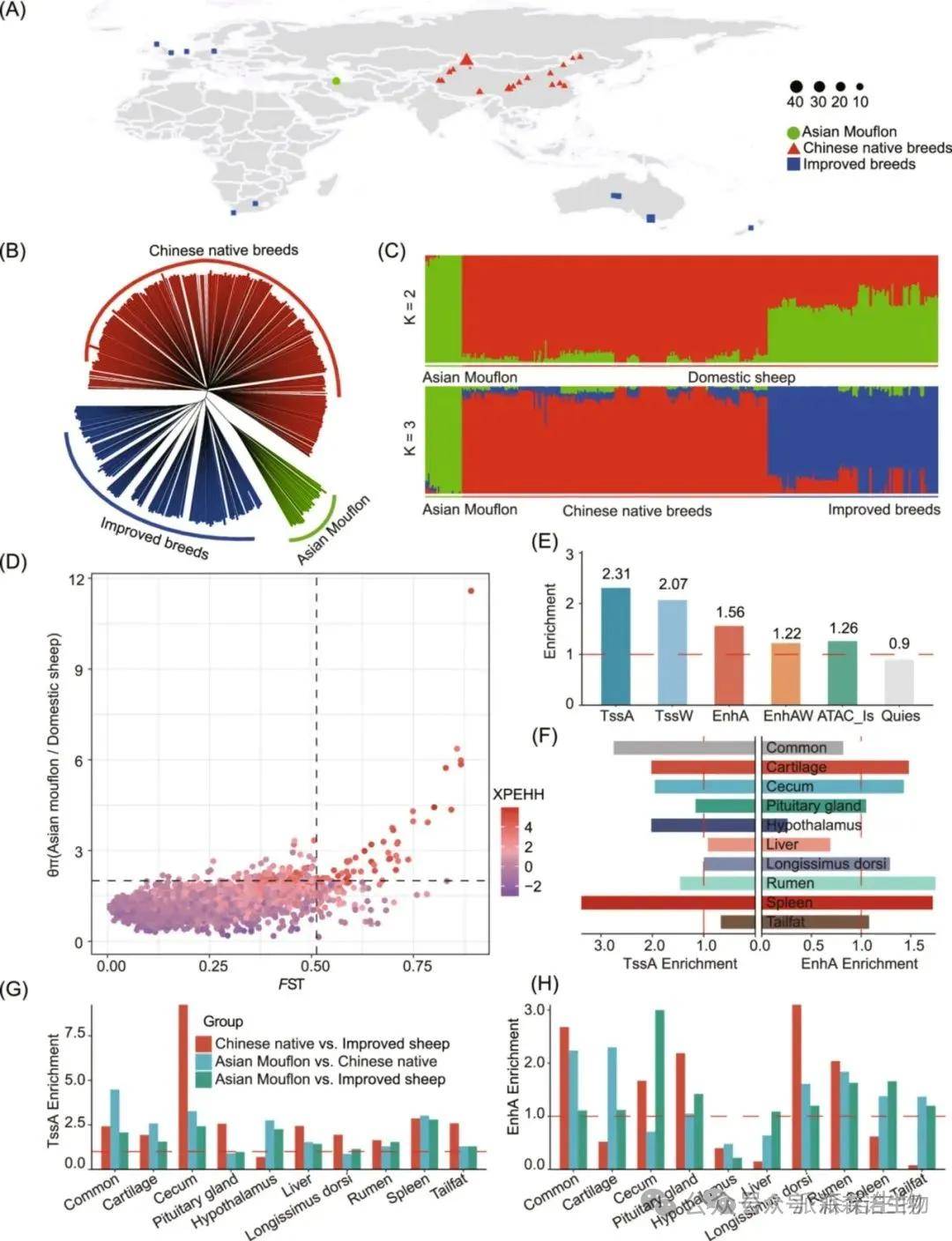
图4 绵羊驯化和改良过程中选定区域染色质状态的富集
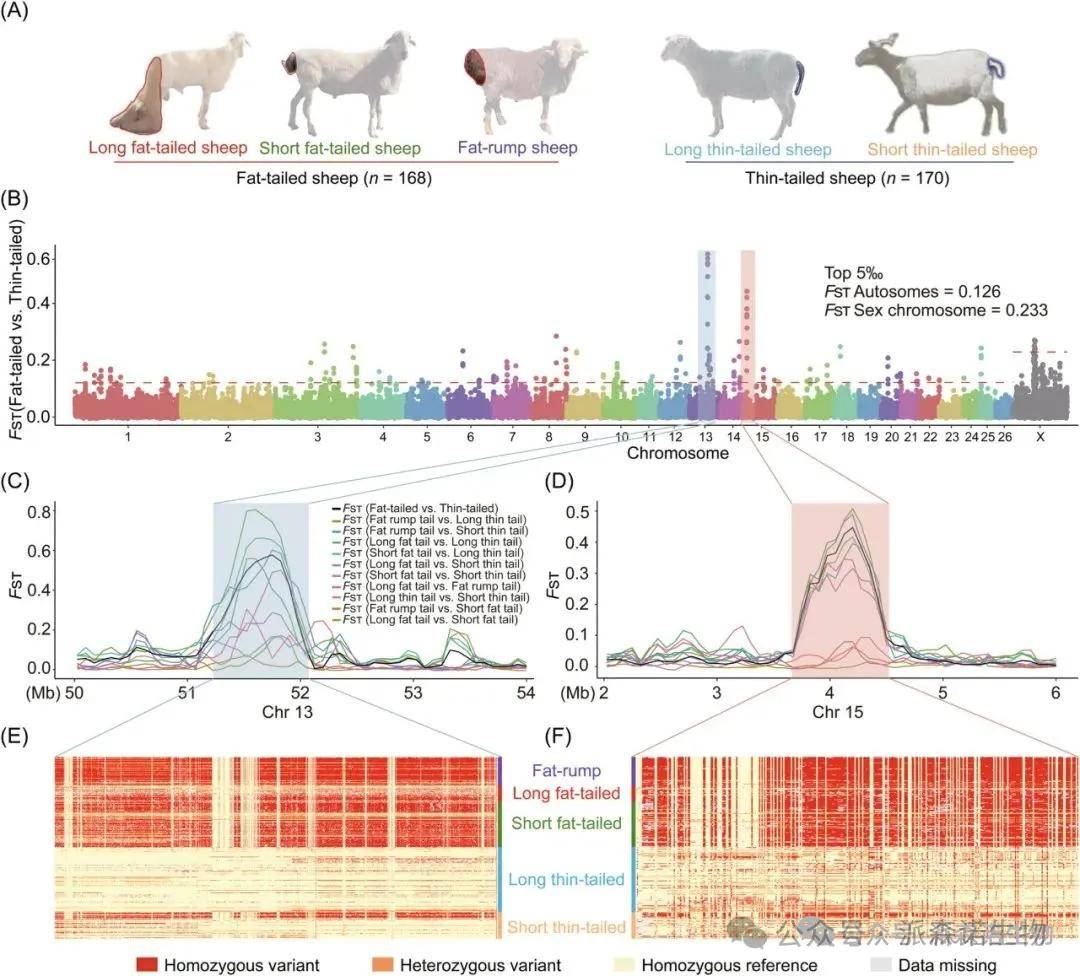
图5 不同尾型绵羊的全基因组选择信号分析
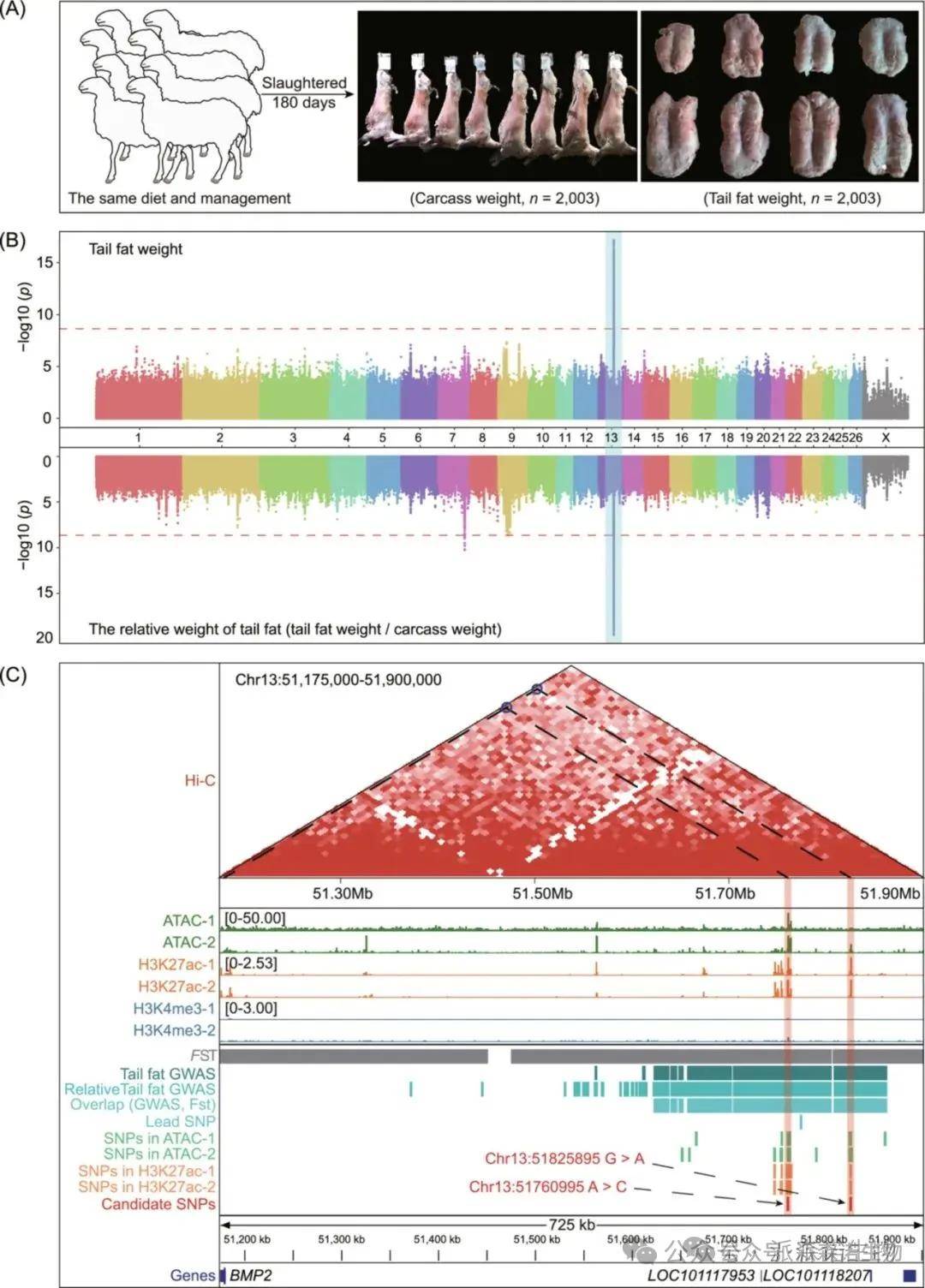
图6 整合全基因组关联研究(GWAS)和表观基因组数据,定位与绵羊尾脂沉积显著相关的候选因果变异
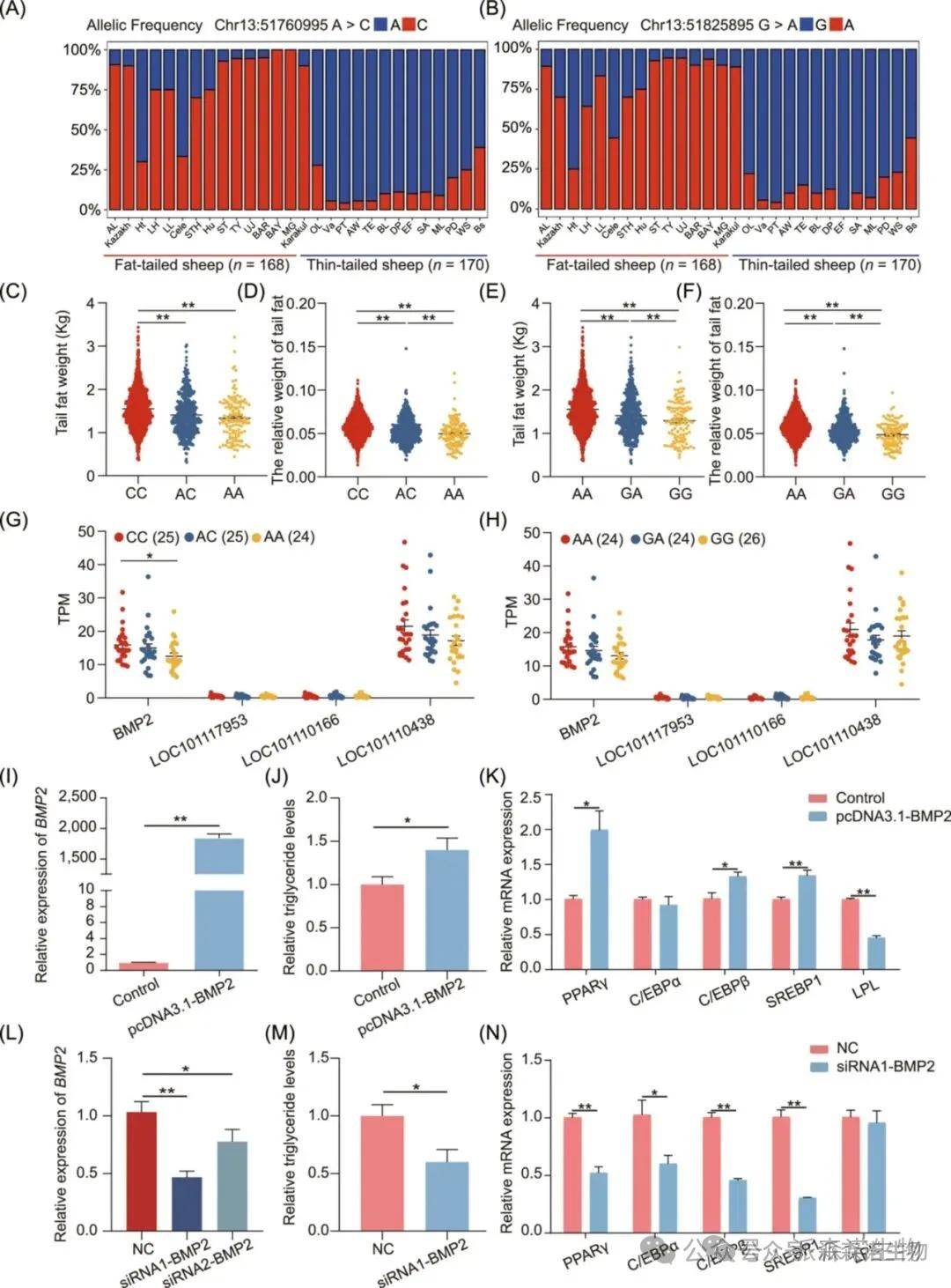
图7 候选功能位点鉴定及靶基因验证
参考文献:
Zhang D, Cheng J, Li X, et al. Comprehensive multi-tissue epigenome atlas in sheep: A resource for complex traits, domestication, and breeding. Imeta. 2024 Dec 15;3(6):e254. doi: 10.1002/imt2.254.




















