
2025-11-03

文章亮点
1.对25份具有广泛抗旱差异的玉米种质进行了高质量基因组从头组装,结合已有的31份基因组,共构建了包含56份材料的玉米泛基因组。
2.基于泛基因组和GWAS分析,鉴定到3个关键的玉米抗旱相关的功能基因,同时利用CRISPR等转基因技术对功能进行了验证。
3.派森诺提供泛基因组分析一站式解决方案:样本搜集、提取建库测序、基因组组装注释、图形泛基因组构建、GWAS分析、个性化分析等。
文章信息
文章题目:A pangenome of maize provides genetic insights into drought resistance
中文题目:玉米泛基因组为抗旱性提供遗传见解
发表期刊:Nature Genetics
影响因子:29
发表时间:2025年10月27日
摘 要
干旱对作物产量的稳定性构成严重威胁。识别遗传资源并解析作物抗旱性的分子机制至关重要。本研究构建了包含56个玉米种质(25个新测序+31个已有)的高质量泛基因组。分析揭示了在脱落酸相关或干旱响应基因中存在的稀有等位变异和广泛的调控多样性,这可能是导致不同种质抗旱性差异的原因。此外,研究人员鉴定出三个基因(ZmUGE2、ZmSIL2 和 ZmASI3),它们分别通过增强细胞壁的机械支撑、调控胁迫响应基因表达以及协调雄穗和雌穗的发育来增强玉米的抗旱性。因此,本研究为理解玉米不同生长阶段抗旱性的遗传调控提供了宝贵见解,同时为玉米基因组学研究提供了重要资源。
前 言
干旱是农业生产(尤其是在发展中国家)的最大威胁之一,造成的损失远超洪涝和病虫害;玉米作为全球最重要的主粮作物,其产量受干旱影响巨大,且对干旱的敏感性在增加;解析玉米抗旱性的遗传机制,对于应对气候变化、保障全球粮食安全至关重要。开花期是玉米对干旱最敏感的时期,但此阶段的抗旱遗传机制知之甚少。传统的全基因组关联分析(GWAS)主要依赖SNP,忽略了重要的结构变异(SV)。玉米基因组复杂(重复序列多、多样性高),使用单一参考基因组难以有效检测SV,无法全面反映物种的遗传多样性。泛基因组研究旨在通过对一组多样化的个体进行测序和分析来捕获物种内的完整遗传多样性,这些研究极大地增进了研究人员对包括玉米在内的不同物种重要性状遗传调控的理解。在本文中,研究人员基于56个玉米基因组,构建了目前最全面的玉米泛基因组,以阐明遗传变异如何影响这种作物的抗旱性。
玉米抗旱性的遗传多样性
研究人员选取了25个玉米种质进行从头基因组测序,这些种质代表了全球热带、亚热带和温带地区广泛的抗旱性谱系和遗传起源。研究构建了苗期和开花期的抗旱性评价体系,苗期以相对于参考品种B73的幼苗存活率(SR)作为关键指标。开花期以开花-吐丝间隔期(ASI)和不同水分条件(正常灌溉、中度干旱、严重干旱)下的籽粒产量作为关键指标。对53份材料进行抗旱性评估发现,19个种质为抗旱,16个种质为中间型,18个种质为干旱敏感型。此外,研究人员发现ASI与产量呈显著负相关,且这种关联性强于苗期SR与产量的关系,在这些种质中,GEMS54、GEMS36、05W002、CIMBL55、D863F和R15被鉴定为抗旱种质。
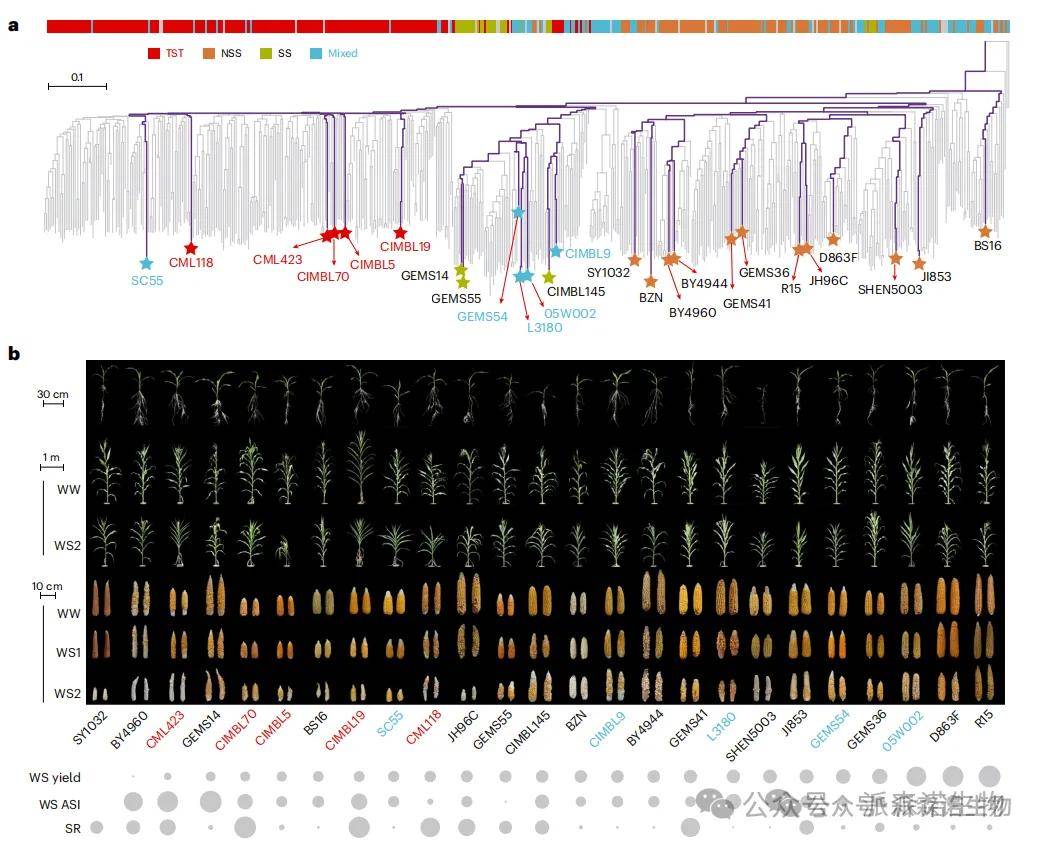
玉米抗旱性研究:25个核心种质的系统发育分析与表型特征解析
25个玉米基因组的从头组装与注释
基于平均深度98X的ONT长读长数据和平均深度60X的Illumina短读长数据,结合共线性挂载技术,研究人员构建了25个玉米种质的染色体级别的参考基因组。组装结果显示平均Conitg N50可达71.04Mb,Busco评估显示基因完整度平均为96.49%,LAI指数超20。基因组注释结果表面。平均每个基因组可以预测到约39880个蛋白质编码基因,BUSCO评估完整度为96.98%。与前人研究结果类似,玉米基因组中85.38%-85.90%的基因组内容被鉴定为重复序列,其中主要为逆转录转座子,占比高达74.87%。
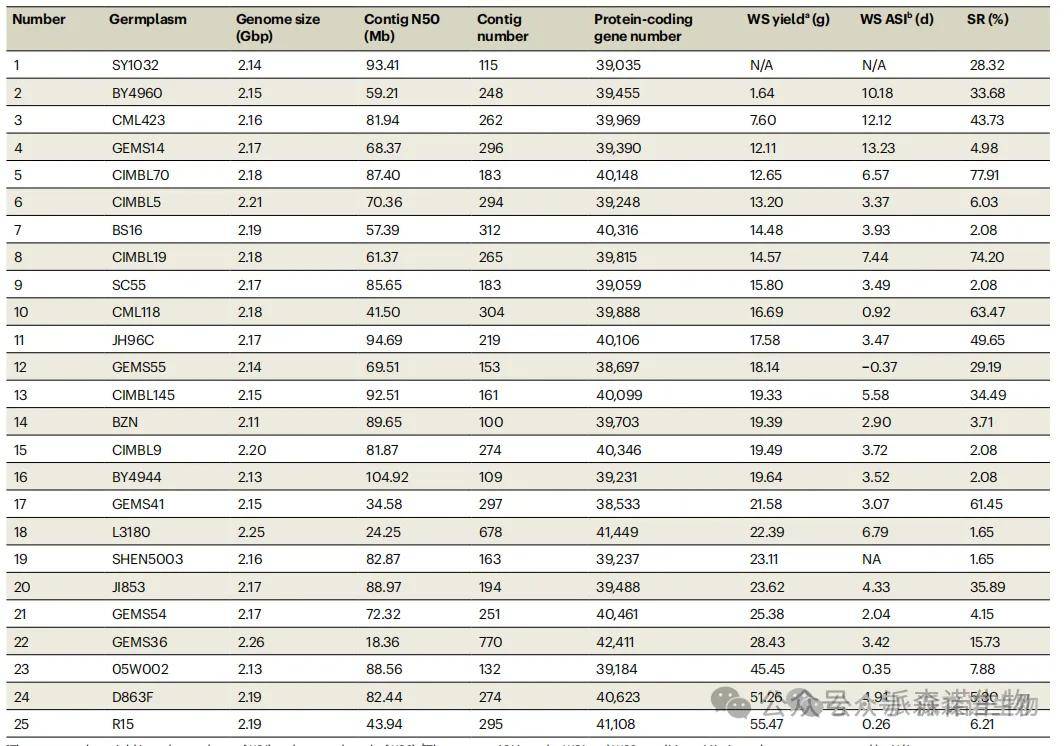
25个玉米参考价基因组组装结果
基于泛基因组视角的ABA相关或干旱相关基因分析
基于整合56个玉米种质构建的高质量泛基因组,本研究在基因和结构变异(SV)层面取得了系统性发现。首先,泛基因组分析显示,尽管经典的ABA或干旱响应基因在种质间高度保守(92.4%属于核心、软核心家族),但通过大规模SV鉴定(近300万个非冗余SV),仍在这些保守基因中发现了存在于特定抗旱或敏感种质中的稀有功能等位变异(如ZmNCED5重复、ZmDRO1缺失等),这些变异是传统SNP分析难以捕捉的。进一步通过构建图基因组优化SV分型后进行群体水平分析表明,SV能显著影响大量抗旱相关基因的表达(eQTL),且基于图基因组的SV数据集(pSV-2)对多种抗旱相关性状遗传力的解释力显著优于SNP和传统方法鉴定的SV。这共同证明,结构变异是驱动玉米抗旱性多样性的关键遗传基础,凸显了基因组结合图泛基因组分析在解析复杂性状中的核心价值。
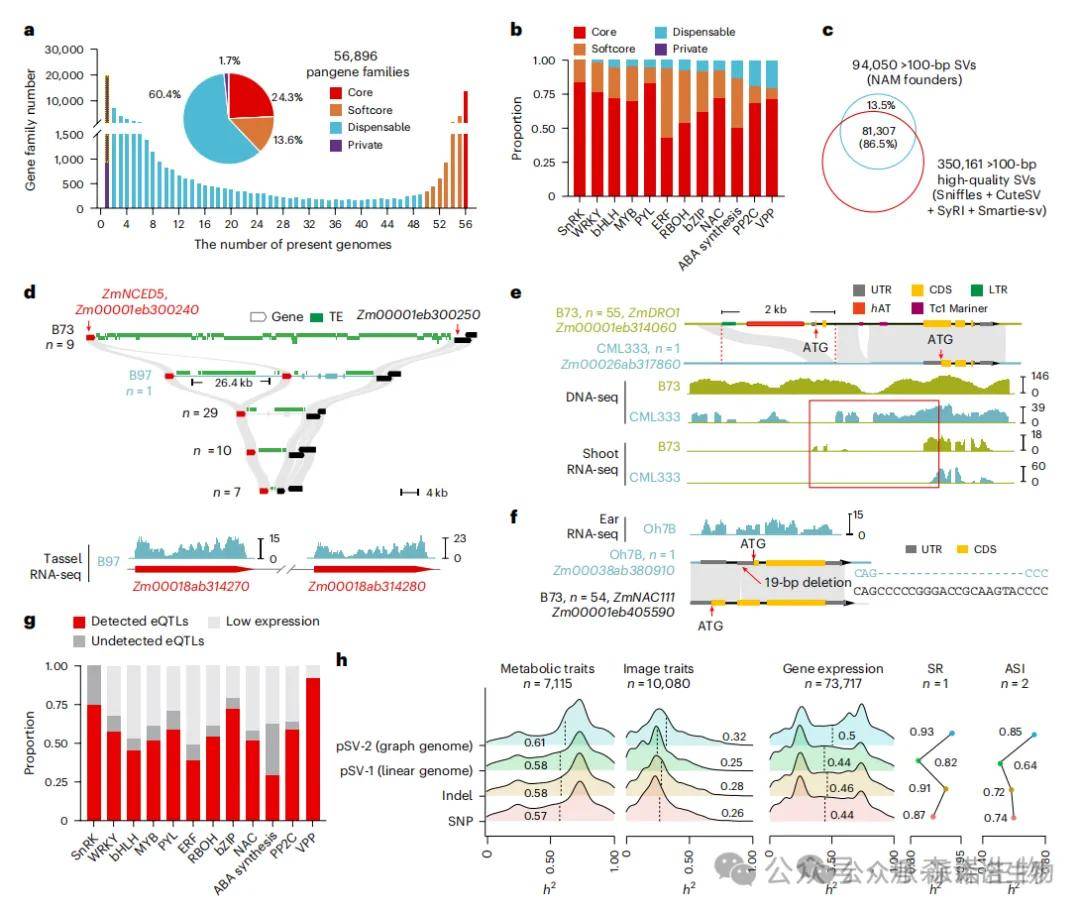
玉米泛基因组分析及其在ABA与干旱响应基因中的变异
ZmUGE2与ZmSIL2在苗期抗旱性中的作用
通过GWAS、单倍型分析和转基因功能验证,研究人员揭示了两个在玉米苗期抗旱发挥关键作用的基因。ZmUGE2为正向调控因子,编码UDP-葡萄糖4-差向异构酶,可能参与细胞壁代谢,过表达ZmUGE2的转基因玉米(ZmUGE2-OE)比野生型(WT)表现出更强的抗旱性,而利用CRISPR-Cas9技术敲除该基因的株系(Zmuge2-KO)则对干旱更敏感;ZmSIL2为负向调控因子,编码一个定位于细胞核的三螺旋转录因子,作为转录抑制子,通过直接结合靶基因启动子区的“CGCCGCCGC” motif,负向调控一系列胁迫响应基因的表达,敲除该基因反而能增强抗旱性,其3‘-UTR区域存在的结构变异(SV),特别是转座子插入,与不同的单倍型和抗旱表型相关,且这些变异在玉米祖先类蜀黍中已存在。
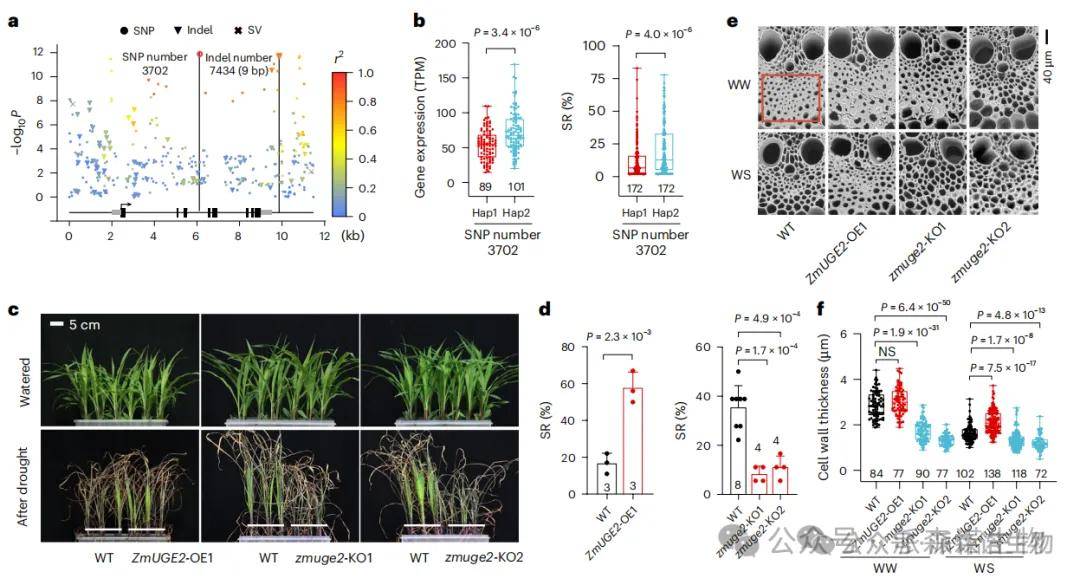
ZmUGE2的转基因研究
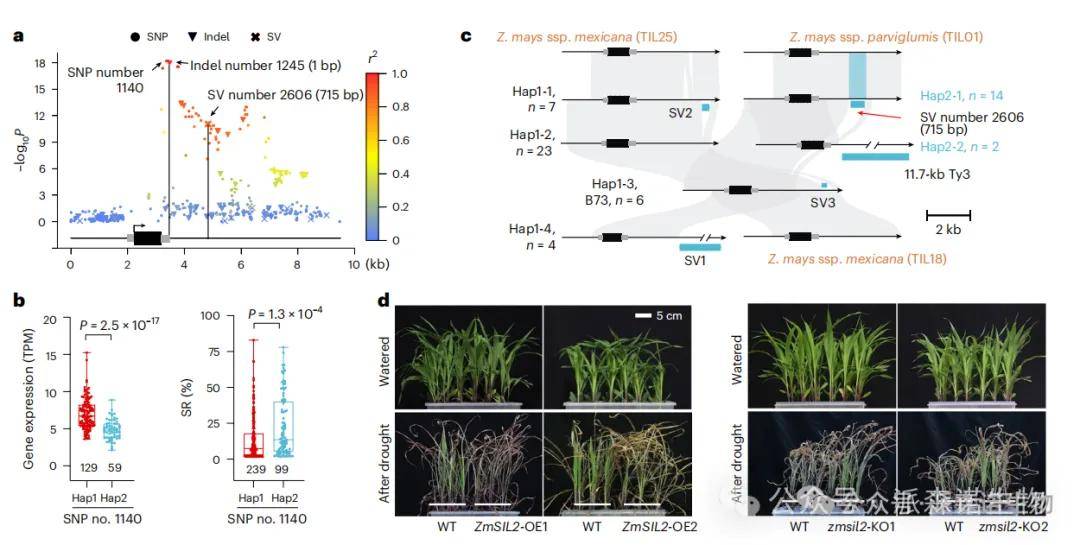
ZmSIL2的转基因研究
ZmASI3 在开花期抗旱性中的作用
基于GWAS分析,研究人员在3号染色体上鉴定到一个与ASI显著相关的区域。该区域在B73参考基因组中被错误注释为两个独立的基因,实际上是一个因巨大内含子5(28.8 kb,富含转座子)而被错误分割的单一基因,将其命名为ZmASI3。根据SNP特征,研究人员划分出HapA(ASI小,抗旱)和HapC(ASI大,敏感)两种主要单倍型。在干旱胁迫下,HapC单倍型由于内含子5的异常剪接,产生两种转录本:功能正常的全长蛋白(P01)和因保留内含子导致翻译提前终止的无功能截短蛋白(P02)。功能实验证实,只有全长P01蛋白具有ATP酶活性和染色质重塑活性。CRISPR-Cas9敲除实验证实,ZmASI3功能的丧失会显著延长ASI并增加产量损失,证明该基因对开花期抗旱性具有正向调控作用。
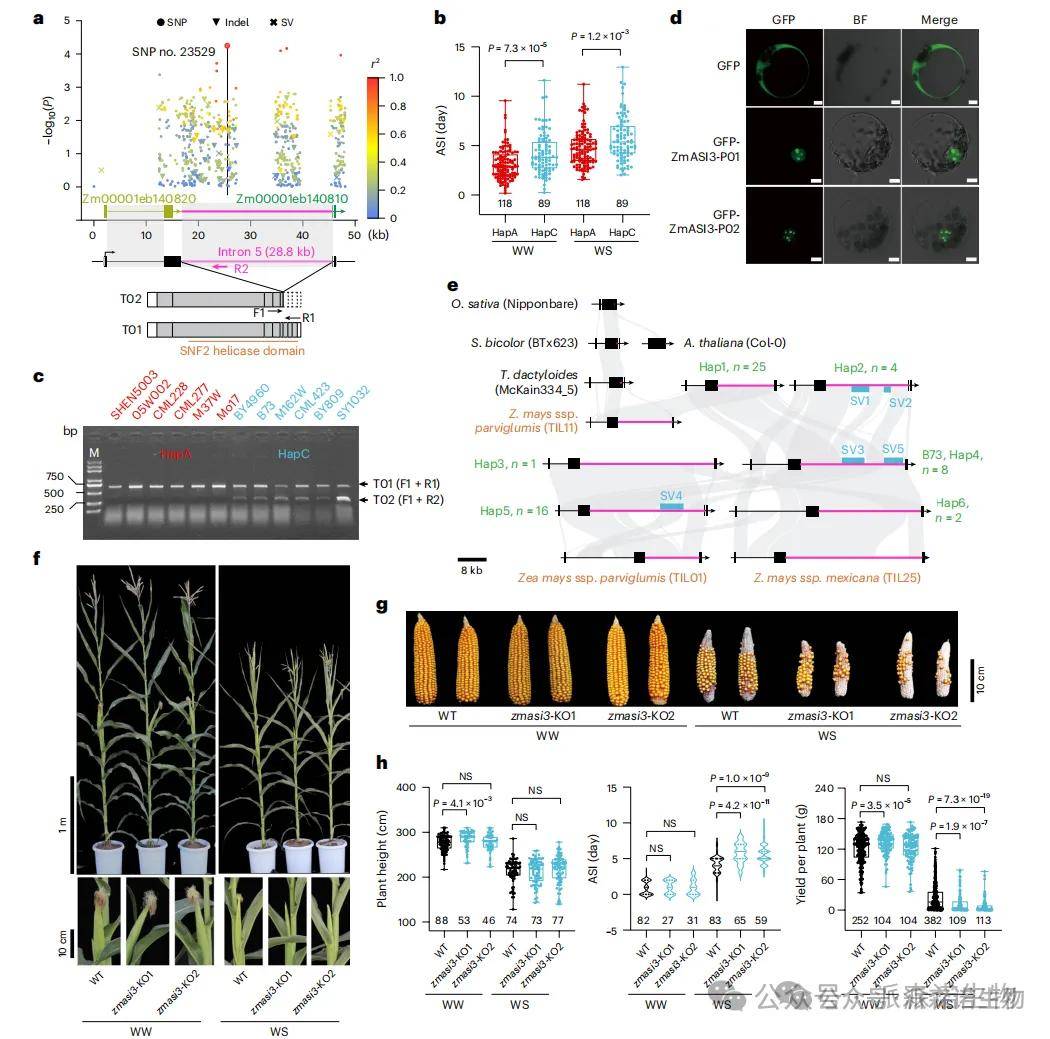
ZmASI3的自然变异调控玉米开花期抗旱性
总结与展望
本研究对25个具有广泛抗旱性差异的玉米种质进行了高质量基因组测序,同时基于泛基因组分析、GWAS、转基因等技术揭示了这种重要作物抗旱性的内在机制。这些成果不仅可用于全面的基因组分析和基因功能研究,而且增进了行业对玉米抗旱机制的理解,同时也为解析其他重要性状提供了宝贵的基因组信息资源。