
2025-12-26


文章亮点
1.研究构建了一个2.63 Gb、无缺口、覆盖全部20条染色体(包括X和Y)的猪基因组,填补了先前参考基因组(Sscrofa11.1)中194.42 Mb的未解析区域(PURs)。
2.完整组装了43.25 Mb的Y染色体,揭示了其重复序列结构、基因拷贝数变异(如HSFY和SRY),并与其他物种(人、羊、山羊)进行了比较。
3.派森诺提供T2T基因组分析一站式解决方案:样本处理、提取建库测序、基因组组装与注释、群体进化分析、性状关联、个性化分析等。
技术路线
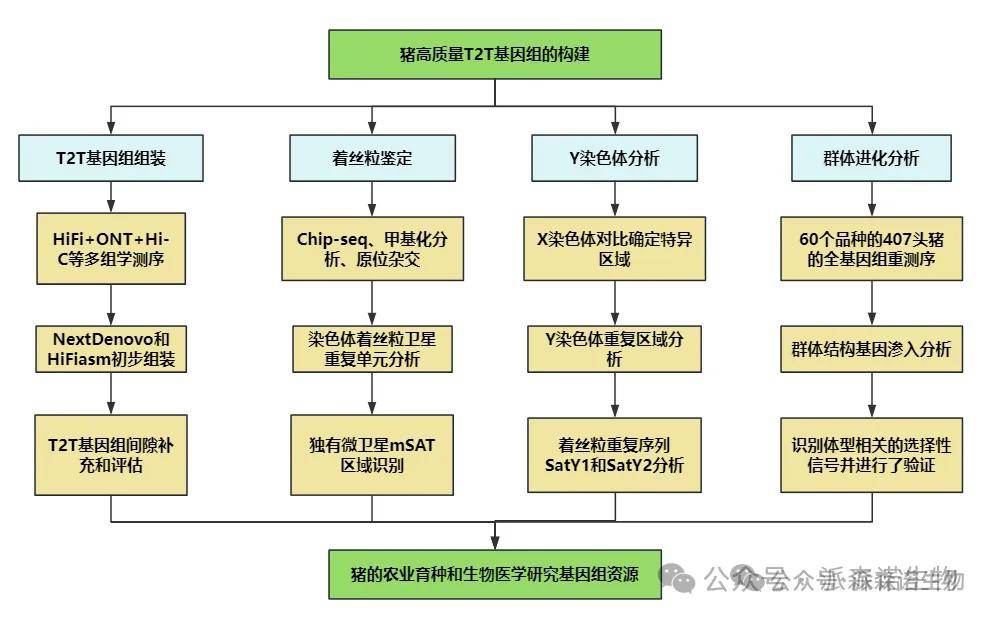
文章信息
文章题目:Telomere-to-telomere genome assembly of a male pig provides insight into population structure and selection for body stature
中文题目:雄性猪端粒到端粒(Telomere-to-telomere,T2T)全基因组组装揭示了群体结构及体型选择的遗传机制
发表期刊:Nature Genetics
影响因子:29
发表时间:2025年12月12日
摘要
本研究构建了首个高精度、无缺口的猪端粒到端粒(T2T)参考基因组 T2T-pig1.0(2.63 Gb),覆盖全部20条染色体,解析了大量以往未解析的基因组区域,并在现有参考基因组基础上新增了多种基因注释,尤其系统完善了Y染色体和着丝粒结构信息。通过在新解析区域中补充大量变异位点,研究更新了猪的群体结构,揭示了跨洲基因渗入现象,并通过选择扫掠分析鉴定出多条与体型性状相关的新选择区域和候选基因,其中 GALNT13 被证实在调控猪软骨细胞增殖与分化、影响体型形成中发挥重要作用。
研究背景
猪既是重要的农业经济动物,也是关键的生物医学模型,但现有猪参考基因组仍存在大量缺口,尤其在端粒、着丝粒、重复序列和性染色体等复杂区域,严重限制了结构变异解析、群体遗传研究及复杂性状(如体型)的遗传机制挖掘。随着长读长测序和组装技术的发展,端粒到端粒(T2T)无缺口基因组已在部分物种中实现,但猪仍缺乏高质量的T2T参考。与此同时,不同猪品种在体型上存在显著差异,如五指山猪等小型地方品种,是研究体型形成和人工选择的理想材料。因此,构建完整、精确的猪T2T基因组,并将其用于群体结构和体型选择信号分析,成为推动猪遗传改良和生物医学研究的迫切需求。
猪T2T基因组的组装和注释
研究人员以一头五指山公猪为材料,综合利用超长 Oxford Nanopore(ONT)、PacBio HiFi、Hi-C、Bionano 光学图谱及短读长测序数据,构建了首个覆盖18条常染色体及X、Y染色体的猪端粒到端粒(T2T)无缺口基因组 T2T-pig1.0(2.63 Gb),完整解析了全部40个端粒,显著提升了基因组的连续性与准确性,并新增了194.42 Mb 以往参考基因组中未解析的复杂区域,揭示了大量新的结构变异。在此基础上完成了系统的基因组注释,发现重复序列占基因组的48.25%,共注释到21,713个蛋白编码基因及多类非编码RNA,其中1,189个新增蛋白编码基因主要位于未解析区域且具有组织表达证据,整体注释完整性和功能覆盖度显著优于现有参考基因组,为猪的功能基因研究和遗传改良提供了更全面、可靠的基因组资源。
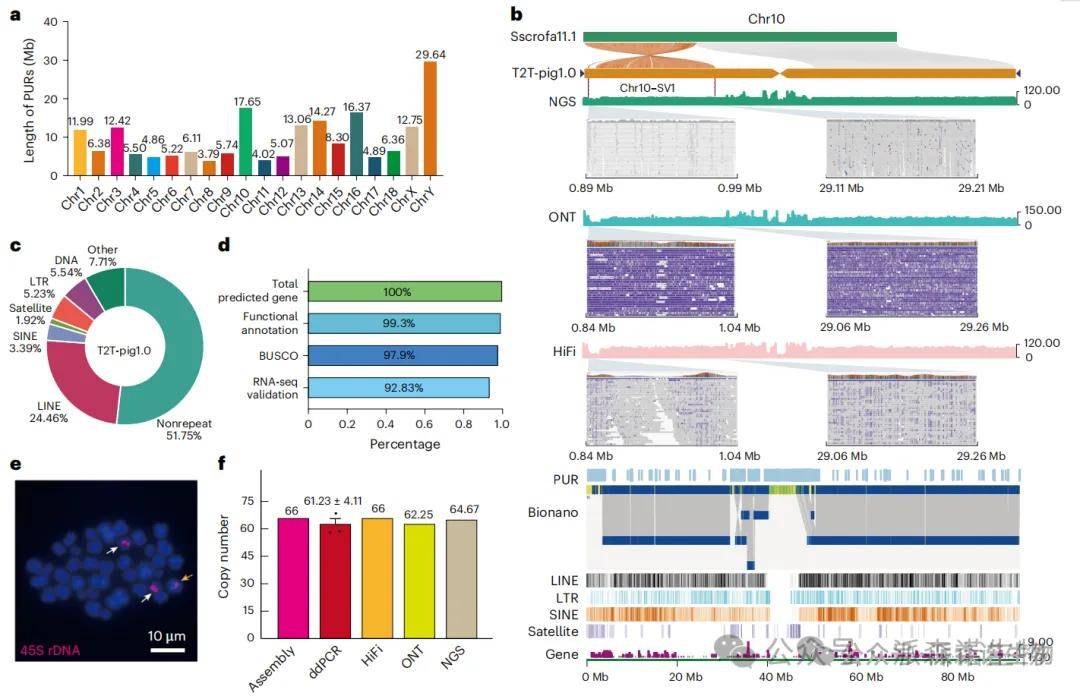
猪完整T2T基因组的组装
猪着丝粒的完整结构
基于T2T-pig1.0无缺口基因组,研究人员首次在全基因组尺度上完整解析了猪所有染色体的着丝粒结构。通过结合CENP-A ChIP–seq、HiFi甲基化信号及重复序列特征,明确鉴定出全部着丝粒区域,总长度约107.3 Mb,占基因组的4.0,并确定了猪染色体的核型类型(中着丝粒、近端着丝粒和端着丝粒)。猪着丝粒主要由多种卫星重复单元(SAT1、SAT2、SAT3)组成,且在不同染色体间呈现显著的分布偏好。尤为重要的是,端着丝粒染色体中发现了一种独特的组织模式“telomere–SAT1B–(mSAT)n–SAT3–q臂”,其中富集的微卫星序列(AGAAT)此前在其他动物T2T基因组的着丝粒中尚未报道。此外,着丝粒区域表现出明显的DNA高甲基化特征,并伴随年轻LTR转座子和DNA转座子的特异性侵入,显示猪着丝粒在组成和进化路径上与人、羊、山羊等物种存在显著差异。这些结果系统揭示了猪着丝粒的完整结构特征及其进化特性。
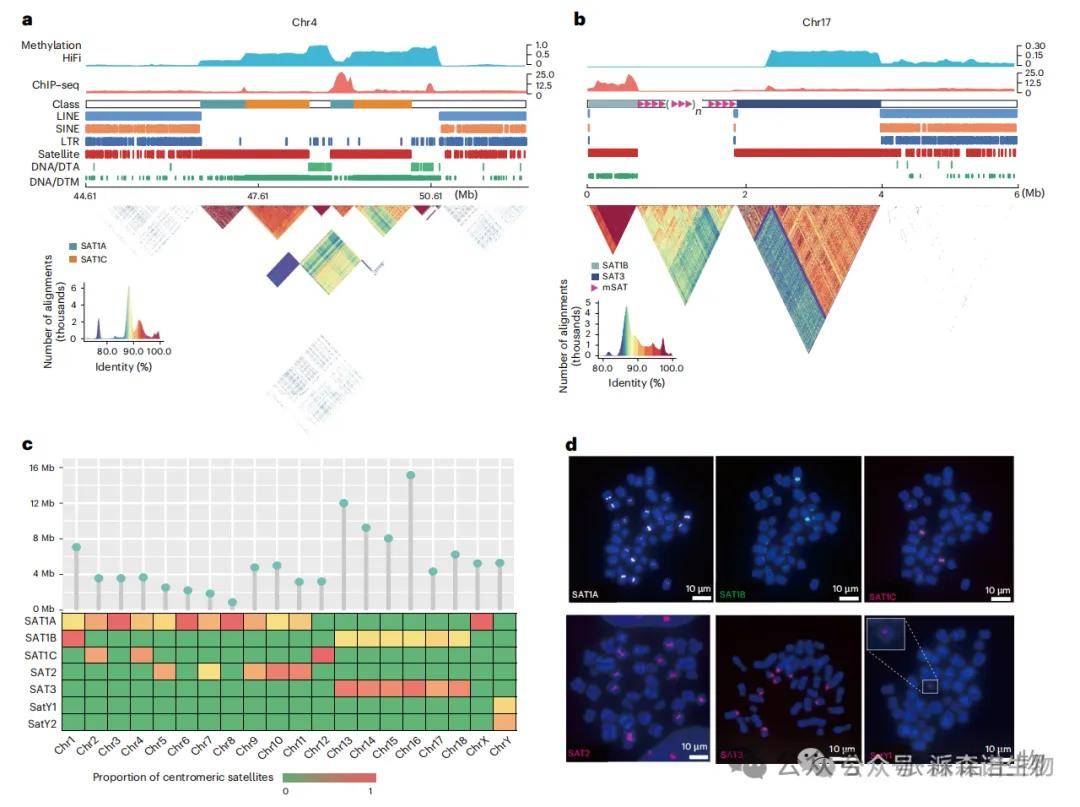
猪着丝粒结构特征
猪Y染色体结构
研究人员首次获得了完整连续的猪Y染色体序列(43.25 Mb),相较于参考基因组 Sscrofa11.1,大幅补齐了约29.6 Mb此前未解析的序列。通过解析X、Y染色体比对关系,明确划分了假常染色体区(PAR)和雄性特异区(MSY),并在chrY上注释到111个蛋白编码基因,其中42个为新发现基因,且多基因(如HSFY)呈现显著的多拷贝扩增。Y染色体以高度重复序列为主(约85%),其q臂富集大规模卫星重复阵列SatY3,结构上与人类Y染色体具有相似性。研究还鉴定出chrY特异的着丝粒重复单元SatY1和SatY2,并通过CENP-A ChIP–seq和甲基化信号精确定位其着丝粒区域。整体而言,该部分系统揭示了猪Y染色体在结构组成、重复序列特征及基因拷贝数变异方面的完整图谱,为理解猪性染色体进化及雄性生殖相关性状提供了关键基础。
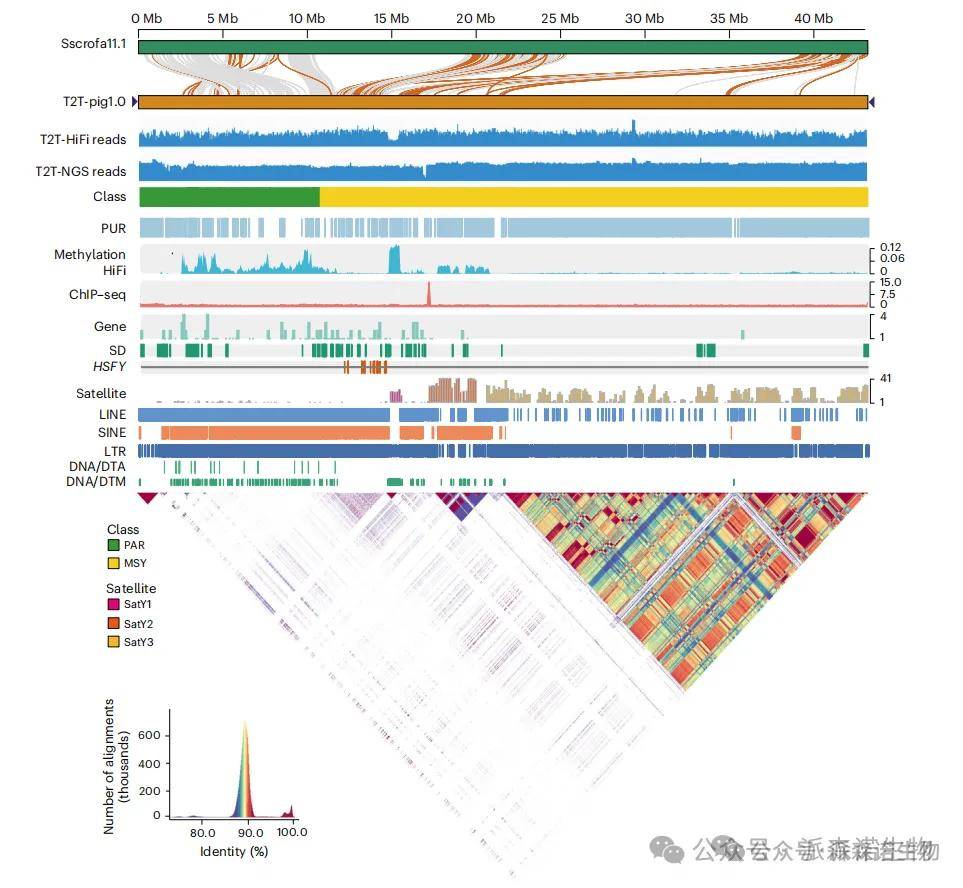
猪Y染色体的结构特征
跨物种比较基因组分析
研究人员通过比较猪(T2T-pig1.0)与人类、小鼠、马、狼、牛等哺乳动物的完整T2T基因组,揭示了猪基因组的进化关系和功能特征。系统发育分析表明五指山猪与杜洛克猪的分化时间约为210万年前,且T2T-pig1.0相比Sscrofa11.1鉴定出更多扩张的基因家族(541个),这些基因显著富集于“负调控细胞大小”等与体型相关的通路,为五指山猪的小体型提供了进化依据。性染色体比较发现chrY上存在11个保守的雄性特异性基因(如SRY、HSFY),支持其在哺乳动物中的功能保守性;假常染色体区(PAR)分析显示猪的chrY PAR最长(约10.20 Mb),而人类因基因丢失导致PAR缩短,同时四物种在PAR中共享11个涉及免疫和骨骼发育的基因(如SHOX)。宏观共线性分析进一步证实了基因组结构的保守性与物种特异性变异。
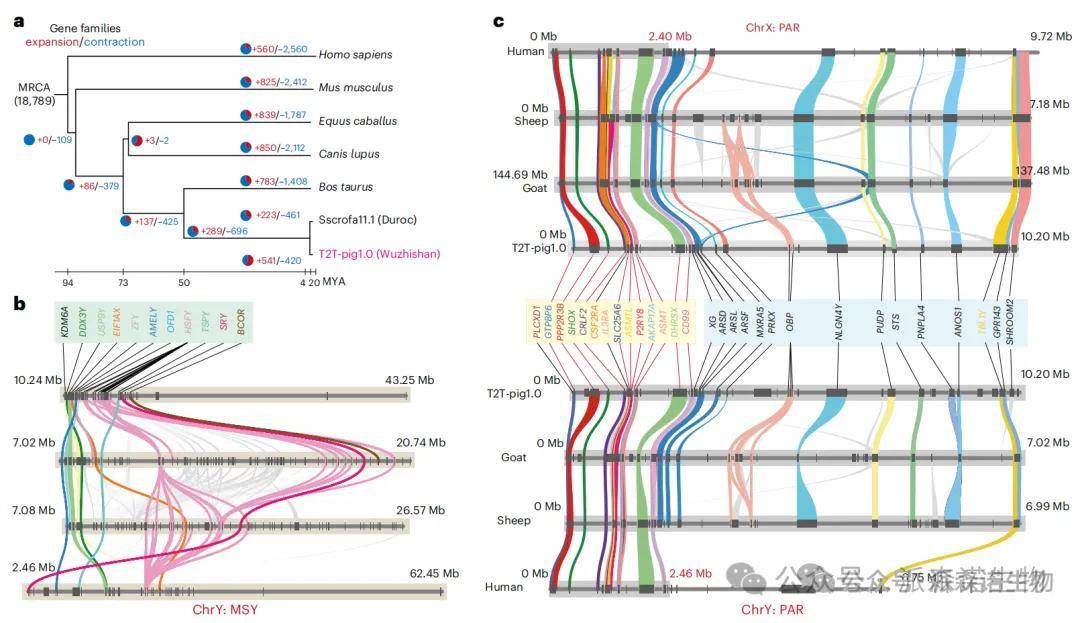
T2T-pig1.0与其他三个T2T基因组chrY的比较分析
GALNT13与猪软骨细胞发育的相关性
种群结构和分化
研究人员对来自全球三大洲60个品系的407份猪样本进行了群体遗传分析。与Sscrofa11.1相比,新参考基因组有效提升了比对质量,并识别出更多SNP(约1785万个),其中1.9%位于此前未覆盖的PUR区域。通过多方法综合分析(邻接树、主成分分析和ADMIXTURE),样本被划分为六大类群,包括亚洲、欧洲和北美地区的家猪与野猪。研究发现PUR区稀有等位基因在亚洲家猪、亚洲野猪和东南亚疣猪中较多,而在欧洲野猪和北美家猪中多为固定变异。亚洲家猪主要呈现三大祖先成分,分别以亚洲野猪、华南和华东猪种为主导,并检测到欧洲血统在东北亚群体中的渗入。相比旧版本,T2T-pig1.0显著提升了对猪遗传结构解析的分辨率,更清晰揭示了亚洲猪群间的基因交流情况。
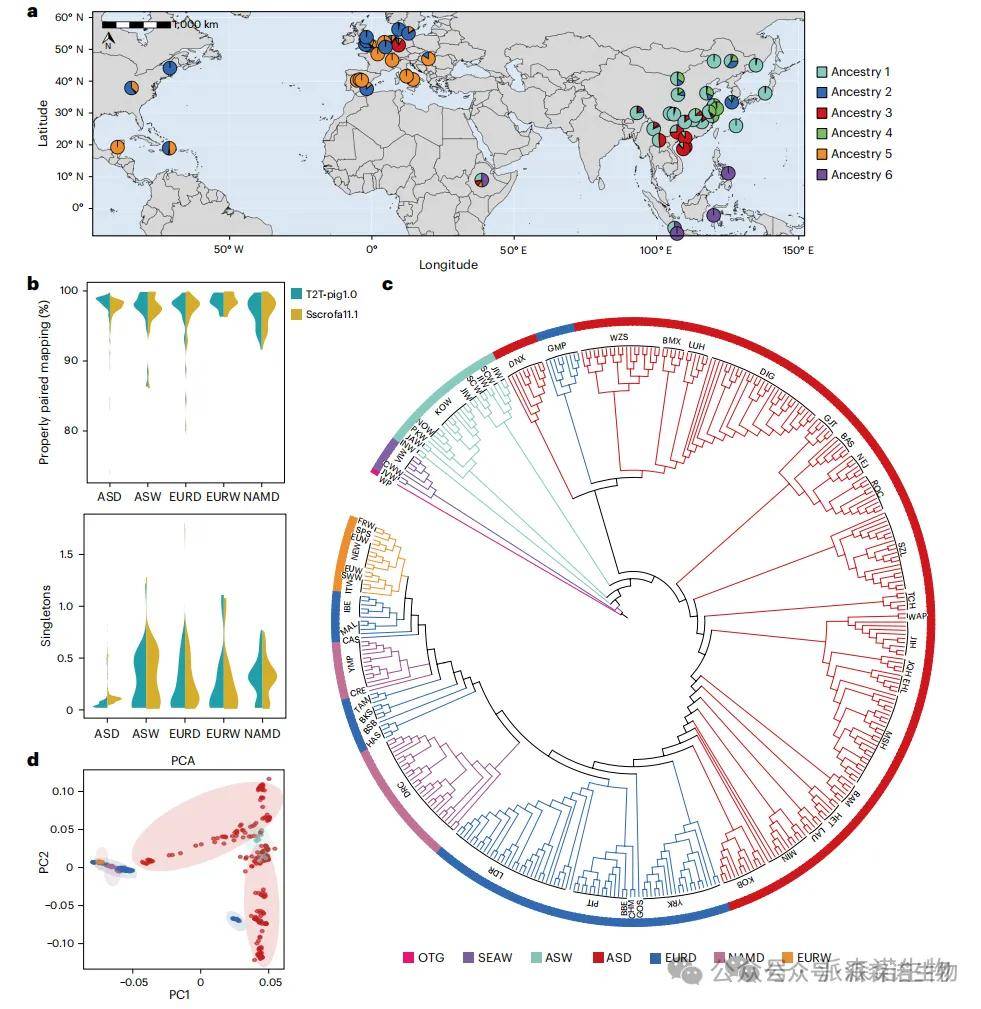
猪种群结构分析
猪体型相关的选择信号
研究通过比较小型猪(五指山猪、巴马香猪)与大型商业猪(杜洛克、约克夏)识别了与体型相关的选择信号,发现X染色体40–135 Mb区间存在大量被选择基因,这可能与该区域低重组率导致的选择信号积累有关。在这些候选基因中,BRCA1、CORIN和GALNT13表现出显著的选择特征,并且已知与体型和骨骼发育密切相关,提示它们可能在猪体型形成过程中发挥关键作用。在体外实验中,GALNT13过表达能够显著促进胸椎和骨骺软骨细胞中关键软骨标记基因(COL2A1、SOX9、RUNX2)的表达,从而增强软骨细胞分化,同时抑制细胞增殖。这表明GALNT13通过调控软骨细胞的增殖与分化参与猪体型形成的分子机制,为揭示猪体长和骨骼发育的遗传基础提供了重要线索,也为未来通过基因选择或功能研究优化猪体型提供了潜在的分子靶点。
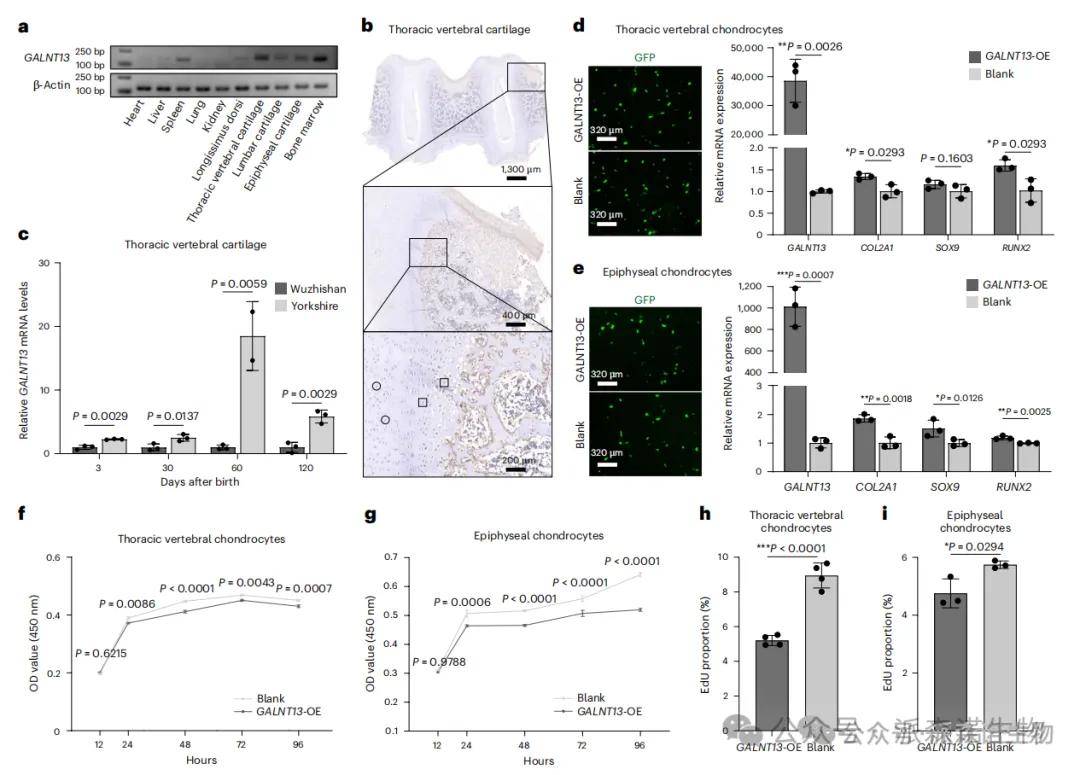
GALNT13与猪软骨细胞发育的相关性
总结
本研究通过T2T-pig1.0组装完成了无缺口猪基因组,揭示了包括PURs在内的未知序列及谱系特异性区域,精确表征了Y染色体和着丝粒重复序列,并发现了新的基因及结构变异。研究显示,猪Y染色体上的多拷贝基因(如HSFY和SRY)数量存在种间和品系间差异,这可能与雄性生殖性能相关。T2T-pig1.0提供了重要的基因组资源,为猪的农业育种和生物医学研究提供基础,同时也为未来深入解析PURs功能及研究着丝粒进化奠定了基础。