2017-02-17
简化基因组测序(Reduced-Representation Genome Sequencing, RRGS)是指利用生物信息学方法,设计分子标记开发方案,筛选特异性长度片段,应用高通量测序技术获得海量标签序列来充分代表目标物种全基因组信息的测序策略。简言之,简化基因组测序就是基因组重测序的简化版,小编称之为 “省钱的重测序”。
目前,主流的简化基因组建库方法包括 RAD 法(Restriction site Associated DNA),2b-RAD 法、GBS 法(Genotyping-By-Sequencing)、ddRAD 法(double-digest RAD,由于该种方法建库的流程更加类似于 GBS 法,所以也被称为 ddGBS 法)。今天,小编就给大家简要地介绍一下 GBS 分析的一般流程。
GBS分析流程的三个主要步骤:
1. 原始数据处理
原始数据通常包含各种类型的错误,如碱基错配、低质量碱基、接头污染。因此,在进行后续分析前,小编强烈建议对下机数据进行质量评估、接头序列去除、低质量数据过滤。目前主流的用于原始数据处理的软件包括:Trimmomatic、PRINSFQ、FastqMcf、FASTX-Toolkit和AdapterRemove2等。
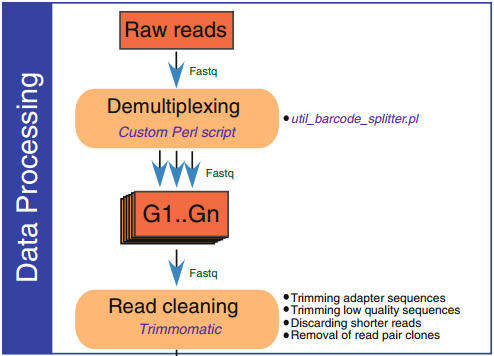
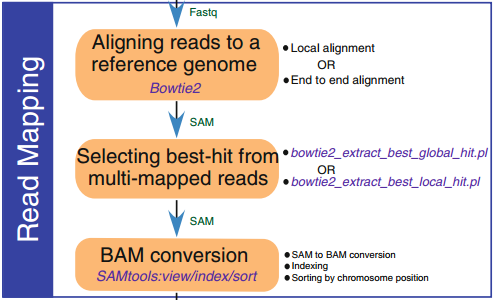
2. 比对到参考基因组
数据过滤后,对于有参考基因组的物种,我们可以将短读长比对到参考基因组,序列比对软件包括(MAP、mrsFast、STAMPY、Bowtie2、BWA和SOAP2等)。对于无参考基因组的物种,我们可以利用STACKS,UNEAK或者RApiD等软件将测序得到的 reads 拼接成 mini-contigs。这些mini-contigs可用于短读长序列比对和基因分型。
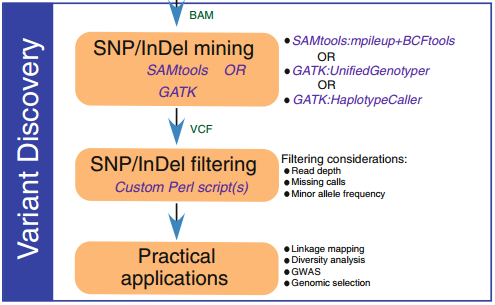
3. 变异检测
比对到参考基因组后,一般会生成BAM文件。然后就可以检测个体或群体的变异(包括SNPs和InDels)。用于变异检测的软件有SAMtools、GATK,SOAP,SNVer和GNUMAP等。
小结:
本文简单介绍了GBS分析的一般流程,以及在每一个流程中需要用到的一些软件。本流程中选择的软件均为运算效率较高的一些软件,具体软件见表1。
表1 GBS 分析流程中使用的软件

此外,小编还想给想要做 GBS或正在做 GBS 的老师们一些建议:
(1) 测序数据量很重要!很重要!!很重要!!!数据量 = 标签数*测序深度*读长;
(数据量太少,样品之间测到的标签会不在基因组的同一位置)
(2) 标签数不是随意定的,酶切位点决定标签数量,所以选择一款合适的酶很重要;
(3) 实验过程很关键,PCR 循环可能会引入大量的重复序列,所以在建库过程中,一定要降低 PCR 循环数!降低PCR 循环数!!降低PCR 循环数!!!
(重复序列的存在会影响变异的检测)
原文索引: DataKagale S , Koh C , Clarke WE. Analysis of Genotyping-by-Sequencing (GBS)[J]Methods in Molecular Biology, 2016


