
2016-08-23
在上一期的“5分钟小课堂”中,小编梳理了宏基因组学研究的关键步骤流程,并概述了挖掘宏基因组学大数据的四大“法宝”:拼接组装、功能注释、生物标记物筛选和菌株水平的精细解析。其中,首当其冲的就是宏基因组的拼接组装,可谓高通量测序中的“拼图游戏”。作为后续三大“法宝”的数据来源,拼接组装的效果将直接影响下游分析的可靠性,对研究意义的重要性不言而喻。今天,就让小编与您一起,探索宏基因组拼图游戏的奥秘,献上史上最全攻略!
1. 什么是拼接?
拼接组装是根据序列的一致性,将高通量测序产生的众多宏基因组DNA短片段依次有序地重叠连接在一起,从而“重建”获得较长的连续不间断序列,也就是传说中的“Contigs”。利用双端PE(Paired-end)序列携带的信息,可以估计Contigs之间的间隔长度(即Gaps),从而连接形成Scaffolds。
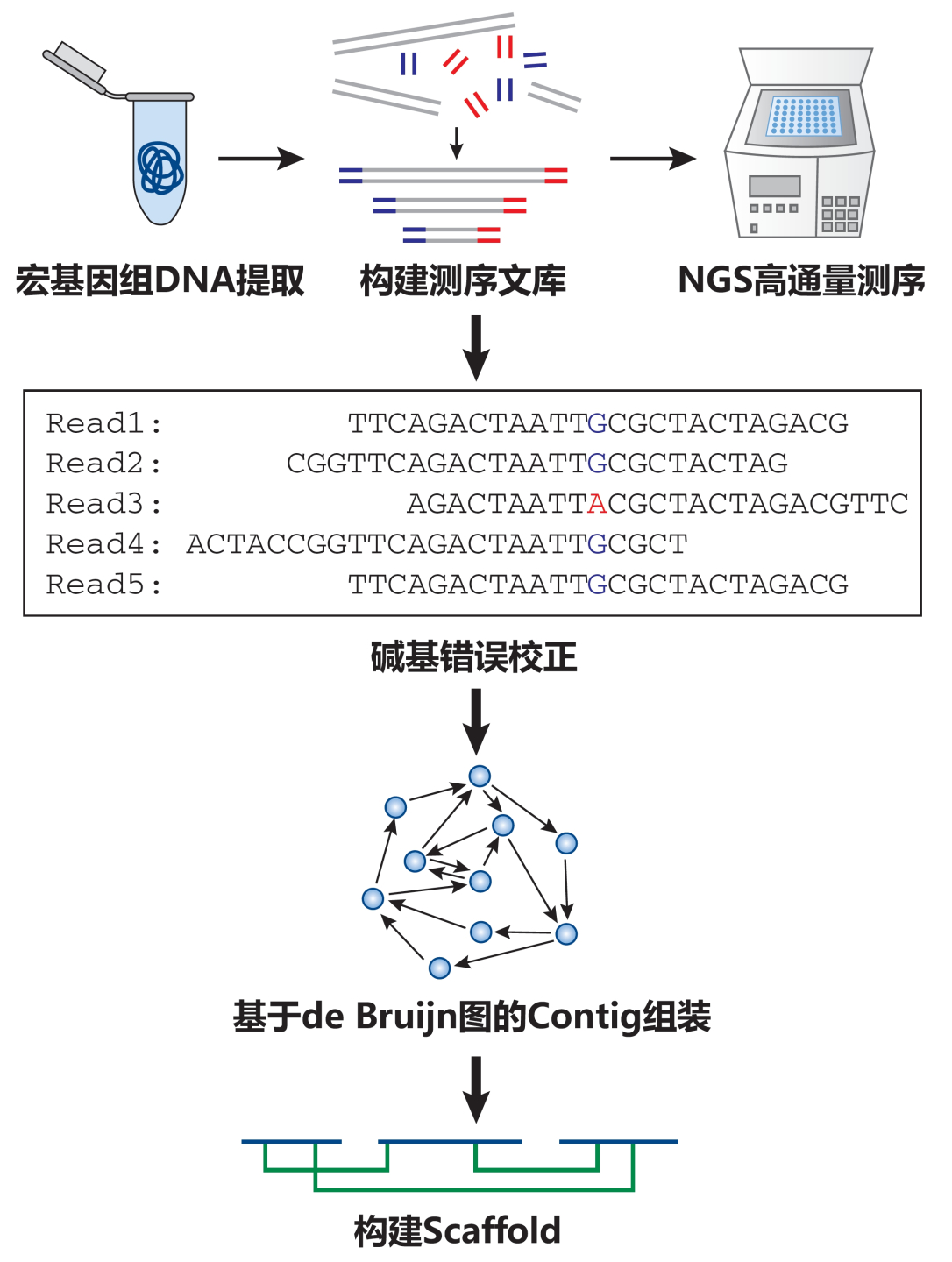
宏基因组的拼接组装流程图,修改自文献[1]
通过以上介绍,小伙伴们有没有发现,宏基因组拼接组装和拼图游戏真的有异曲同工之妙呢!这里,拼图的原材料就是短片段序列,拼出的图就是Contigs和Scaffolds,而游戏的通关秘诀,无疑就是选取合适的拼接组装算法啦!
2. “拼图游戏”的关键:de Bruijn图和一笔画问题
宏基因组包含成千上万种微生物,彼此之间的含量差异可达好几个数量级。拼接组装这样的“大杂烩”,不仅需要庞大的数据量,更需要选取精巧、合适的算法。
目前的宏基因组大数据通常由Illumina HiSeq测序仪产生,序列较短(2 × 150 bp)但通量极高。因此,科学家对序列拼接组装的算法做了针对性的优化,将拼图游戏简化为我们熟知的“一笔画问题”图论问题,下图就是个鲜活生动的例子:
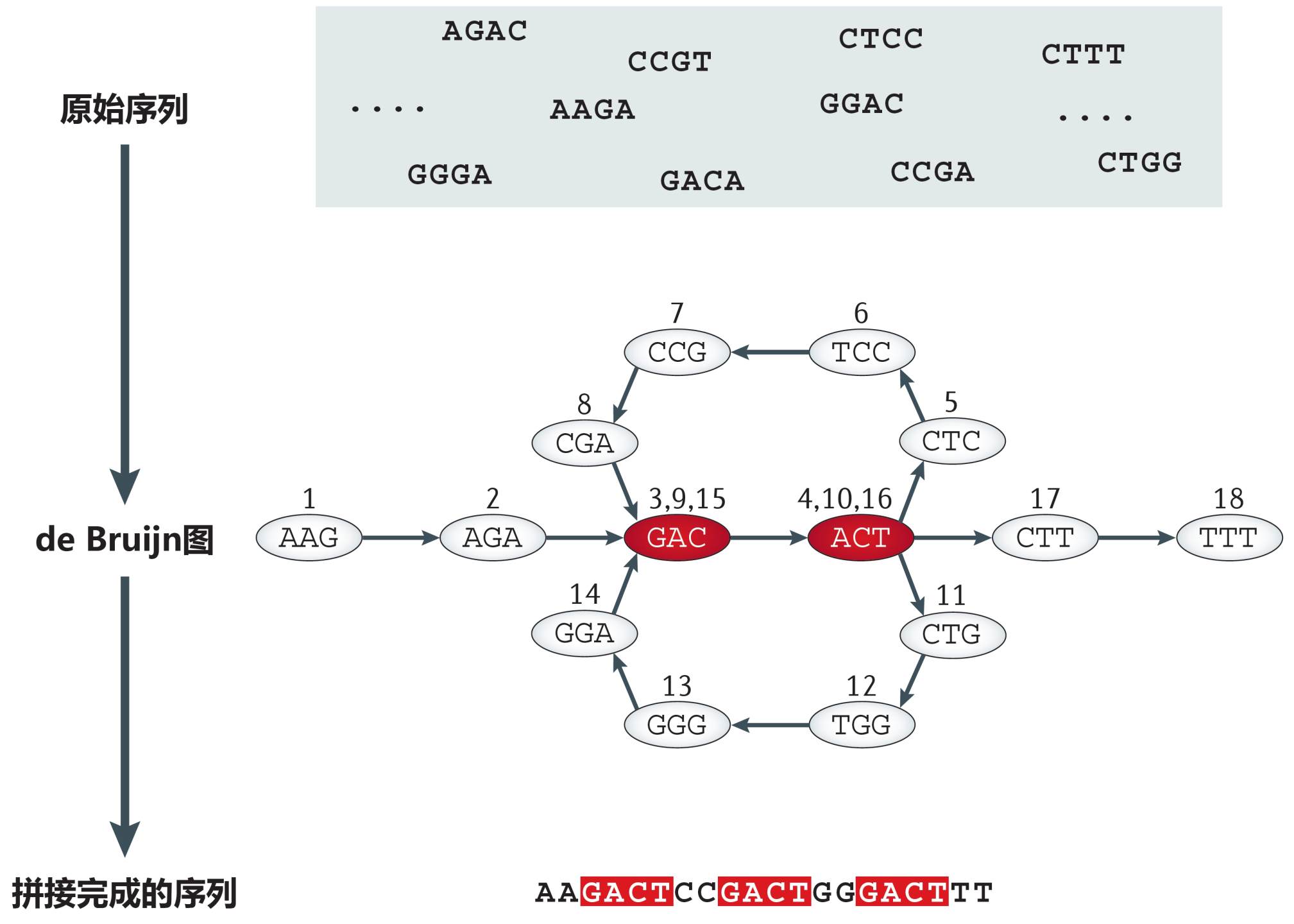
基于de Bruijn图的序列拼接组装示意图,修改自文献[2]
上图中,原始序列长度为4碱基。首先将每一条短序列都拆分为一系列长度为k的子片段(俗称k-mer),比如,图中的原始序列AAGA被拆分为AAG和AGA两个长度为3碱基的k-mer。然后根据全体k-mer之间的连接顺序和重叠关系构建de Bruijn图,尝试找到一次性遍历所有k-mer的“一笔画”路径,由此完成拼接组装,获得Contigs和Scaffolds序列(图中的红色部分为重复序列,可以看到,它们也被正确识别和拼接)。
3. 常用拼接组装工具简介
目前,基于de Bruijn图的序列拼接组装工具已成为主流,包括SOAPdenovo2[3]和IDBA-UD(Iterative De Bruijn graph Assembler for sequencing data with highly Uneven Depth)[4]等。这些工具都能对原始序列中隐含的测序错误进行校正,从而提升拼接组装的精确度。通常而言,SOAPdenovo2的拼接速度较快,而IDBA-UD采用了迭代算法,从一系列k-mer值中,选取最合适的k-mer参数进行拼接组装,同时针对宏基因组中不同物种测序深度不均一的现象进行了优化,因而被认为更适合于宏基因组的拼接组装。
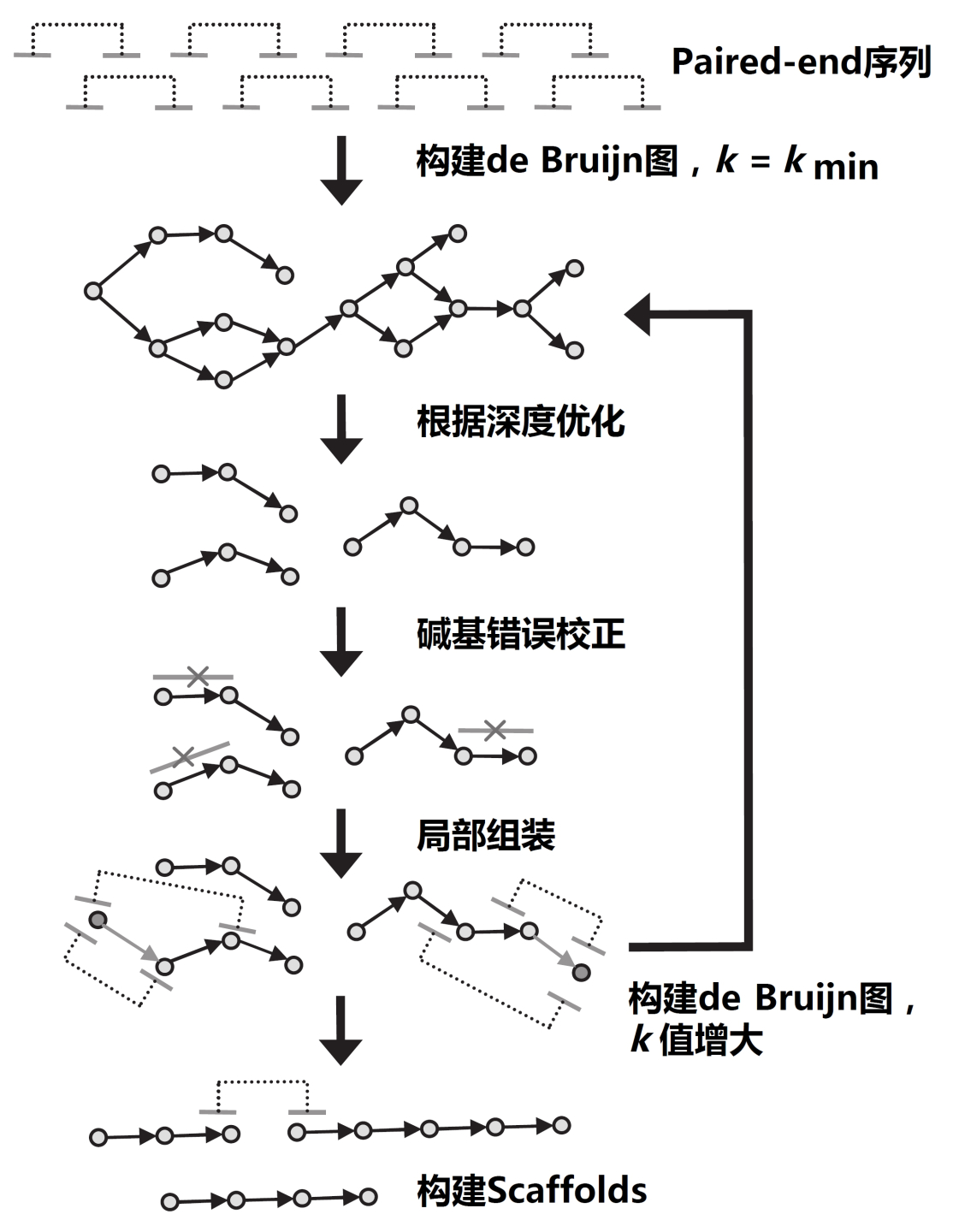
IDBA-UD拼接组装流程图,修改自文献[4]
当然,de Bruijn图也并非万能。对于最近日渐流行的三代单分子实时测序技术,由于其具有超长读长的特性,HGAP(Hierarchical Genome Assembly Process)[5]等根据序列比对寻找彼此之间重叠区域的方法更为合理。
4. 拼接效果的评价
正如游戏得分有高低,在拼接完成后,我们也需要对组装效果进行评估。显然,Contigs和Scaffolds长度是评价的重要标准之一。通常我们使用N50值来评估,将所有Contigs/Scaffolds序列按照长度从长到短依次排列后相加,当加和的长度达到总长度的50%时,最后一条Contigs/Scaffolds序列的对应长度即N50值。显然N50越长,拼接组装效果越好,宏基因组序列也就越完整。
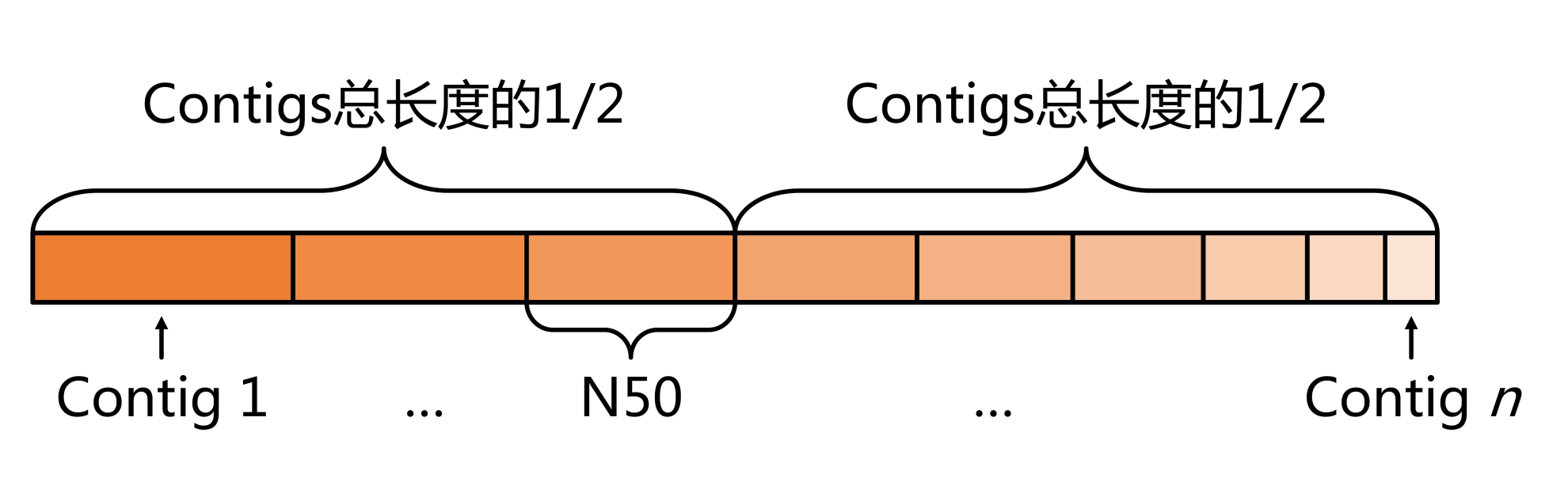
N50值计算示意图
当然,宏基因组的复杂程度将直接影响拼接组装的效果。比如,对于肠道宏基因组样本,迄今为止的几项大型研究(如MetaHIT、HMP和IGC等项目)得到的基因目录的N50值都在1 kb左右[6-8]。
结语
通过以上的讲解,小伙伴们对宏基因组的“拼图游戏”应该入门了吧!总体而言,拼图游戏虽然复杂,但只要选对合适的算法,通关也并非遥不可及!当然,随着科技的不断发展,我们也期待涌现更多更强大的宏基因组拼接组装工具。
至于拼接获得的Contigs/Scaffolds序列如何用于后续分析,且待下回分解,敬请各位小伙伴保持关注哦!
附:【5分钟小课堂】后续预告
l 看不见摸不着的它们,都在忙些啥?宏基因组功能注释为您解答!
l 茫茫菌群,谁是天使,谁是元凶,谁又是围观路人甲?
l 菌株水平的超高分辨率解析,宏基因组学就是这么高大上!
参考文献
1. Fan W, Li RQ (2012) Test driving genome assemblers. Nature Biotechnology 30: 330-331.
2. Berger B, Peng J, Singh M (2013) Computational solutions for omics data. Nature Reviews Genetics 14: 333-346.
3. Luo RB, Liu BH, Xie YL, Li ZY, Huang WH, et al. (2012) SOAPdenovo2: an empirically improved memory-efficient short-read de novo assembler. GigaScience 1: 6.
4. Peng Y, Leung HCM, Yiu SM, Chin FYL (2012) IDBA-UD: a de novo assembler for single-cell and metagenomic sequencing data with highly uneven depth. Bioinformatics 28: 1420-1428.
5. Chin CS, Alexander DH, Marks P, Klammer AA, Drake J, et al. (2013) Nonhybrid, finished microbial genome assemblies from long-read SMRT sequencing data. Nat Methods 10: 563-569.
6. Huttenhower C, Gevers D, Knight R, Abubucker S, Badger JH, et al. (2012) Structure, function and diversity of the healthy human microbiome. Nature 486: 207-214.
7. Li JH, Jia HJ, Cai XH, Zhong HZ, Feng Q, et al. (2014) An integrated catalog of reference genes in the human gut microbiome. Nature Biotechnology 32: 834-841.
8. Qin JJ, Li RQ, Raes J, Arumugam M, Burgdorf KS, et al. (2010) A human gut microbial gene catalogue established by metagenomic sequencing. Nature 464: 59-65.