2018-05-15
维恩图:也叫文氏图,韦恩图,用于显示元素集合重叠区域的图示。是不是不太容易理解?没关系,让我们用转录组测序结果来举例说明。
某高校老师设计实验如下:
处理类型 | 对照组 | 高温组 | 高盐组 |
对照组 | 1 | 2 | |
高温组 | 1 | 3 | |
高盐组 | 2 | 3 |
得到差异基因集合如下:
差异基因集合 | 差异基因ID |
1 | ABCDEFG |
2 | CDFGILKPW |
3 | ABCDJKM |
现在问题来了,如何知道哪些基因同时响应了高温和高盐胁迫呢?我们只需要明确差异基因集合1和2中有哪些基因是共有基因即可。Dangdangdang~~~此时就需要维恩图上场啦!方法有三种:
1. 用肉眼归类,找出集合1和集合2共有差异基因和特有基因;缺点的:这个策略仅仅对个位数的基因数有效,一旦差异基因达到几十,几百,估计是要头晕眼花了,而且出错的概率也会大大增加。
2. 使用R包,例如 VennDiagram,绘制维恩图;缺点:可以绘图,但是无法输出维恩图各个部分的基因ID的列表。
3. 编程统计,并绘图;缺点:阅读完一本perl语言入门,学会哈希的使用并编写脚本分析,这个时间至少要耗费数个星期,数月也是有可能的。
下面要说的是一种可以避开上述所有难处、缺点和不足的维恩图制作方法。![]()
1. 打开venny制作网站http://bioinfogp.cnb.csic.es/tools/venny/
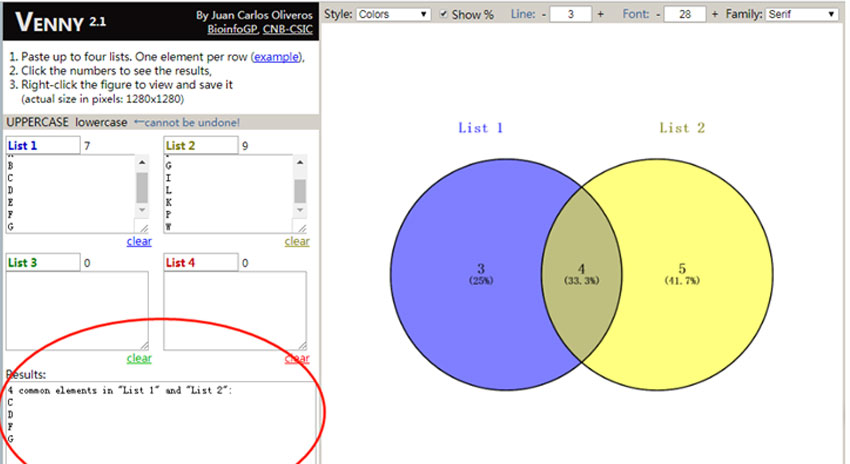
2. 将集合1的差异基因ID复制到list1中,将集合2的差异基因ID复制到list2中,即可得知集合1和集合2共有和特有差异基因的数目。

3.如果想知道具体共有或者特有的基因对应的ID,很简单,只需要用鼠标轻轻点击一下维恩图中特定的数字,然后就可以看到result中会出现维恩图中特定区域对应的基因ID。
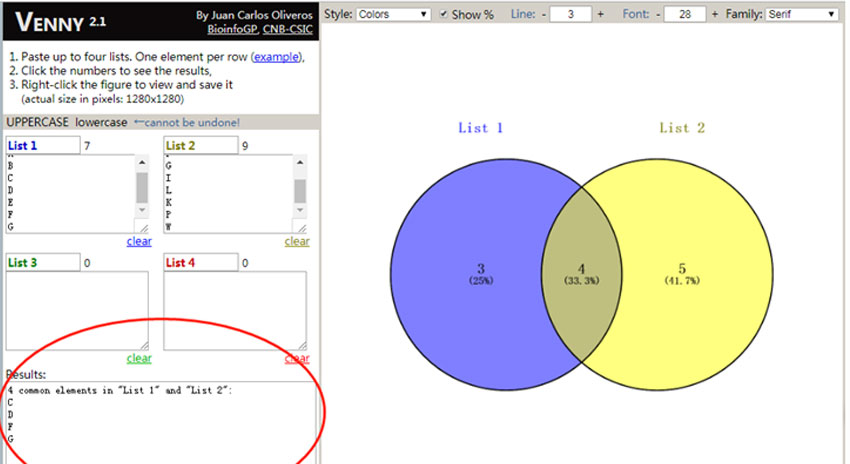
现在还有很多在线网站可以让各位老师和同学自己动手DIY维恩图,例如:
http://bioinfogp.cnb.csic.es/tools/venny/
http://bioinformatics.psb.ugent.be/webtools/Venn/
http://www.biovenn.nl/index.php
http://www.cmbi.ru.nl/cdd/biovenn/index.php
各家网站做出来的维恩图外观可能会和老师心里的维恩图长得不太一样,这个时候不用担心,我们已经获得了共有和特有基因统计结果,只需结合各种画图软件(AI,PS或者PPT),想怎么画就怎么画,随意打造我们自己心目中的维恩图。


