2018-05-17
2018年1月1日起,微生物系统分类的国际权威《International Journal of Systematic and Evolutionary Microbiology》在其官网发布新闻“Genome sequencing data required with Taxonomic Descriptions”,国际系统与进化微生物学杂志(IJSEM)要求作者提供基因组测序数据,并在分类学描述中提供新的分类单元的描述。
新的分类单元确定常用的方法主要有DNA-DNA杂交(简称DDH),16S rRNA鉴定以及ANI分析。但DDH分析复杂耗时,16S rRNA 鉴定存在局限性(如某些物种的16S rRNA相似度高达99%,但DDH值却很低),因此ANI分析逐渐受到科研工作者的青睐。
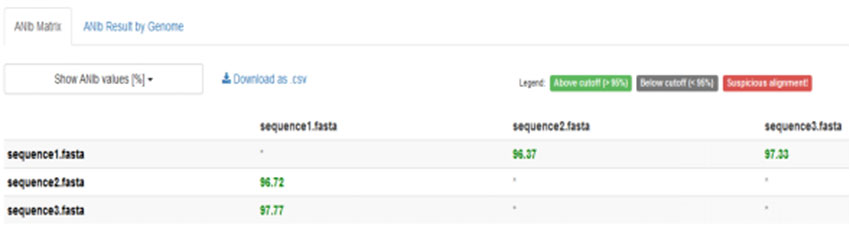
ANI分析全称Average nucleotide identity,即平均核苷酸多态性,主要用于在全基因组水平间评估物种间的亲缘关系,ANI值为95%被视为界定种的标准。
ANI值的计算主要由几种不同的方法实现,Jspecies是其中较常被使用的一种,Jspecies主要有软件版和在线版。今天小编主要给大家介绍的就是用于计算ANI值的一个快速且易于使用的在线网站JspeciesWS(http://jspecies.ribohost.com/jspeciesws/),通过计算ANI值判断两个或多个基因组是否属于同一物种。JspeciesWS的基因组数据库取自ftp://ftp.ncbi.nlm.nih.gov/genomes/genbank,类似于NCBI的基因组序列数据,包含了组装基因组序列及其相关注释数据。对于每个条目,网站提供该领域的界、门、纲、目、科、属和种的分类学信息。
下面就由小编带领大家来一探究竟吧!
1、JSpeciesWS不需要用户注册,界面友好。点击Start a Analysis即可开始分析。

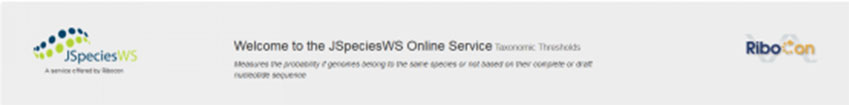
2、点击开始后,会链接到一个“Genome cart””会话框。
“Genome cart””会话框中用户可以上传自己测得的基因组序列或直接从JSpeciesWS参考数据库“GenomesDB”中添加基因组序列。“Genome cart””会话框中在闲置14天后自动失效(不会根据提供的会话代码重新访问),并且所有数据都将被删除。目前“Genome cart””会话框的基因组数量限制为15。
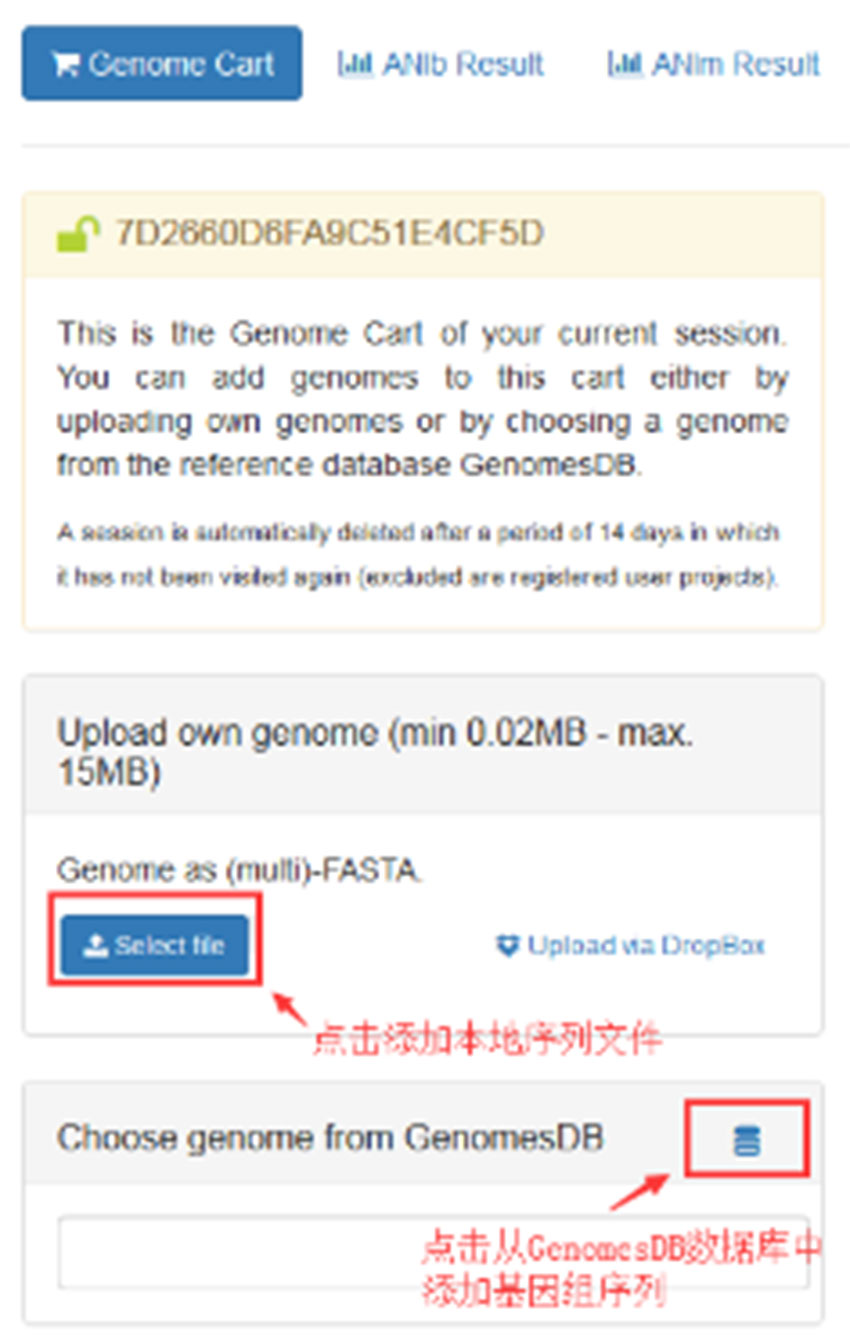
3、一键完成ANI值计算
网站可一键完成“Genome cart””会话框中所有基因组之间成对的ANIb,ANIm计算。
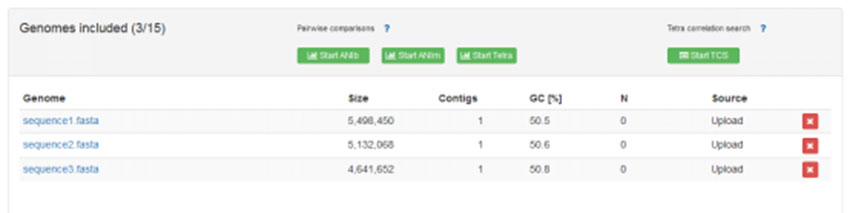
以对3条序列进行的ANIb计算为例,点击“start ANIb”之后,需选择成对分析的计算方式:
l “All vs all”即分别以sequence1,2,3为参考基因组与另外两个序列进行成对分析,分析次数为“6”;
l “Sequence1.fasta”即以Sequence1为参考基因组。
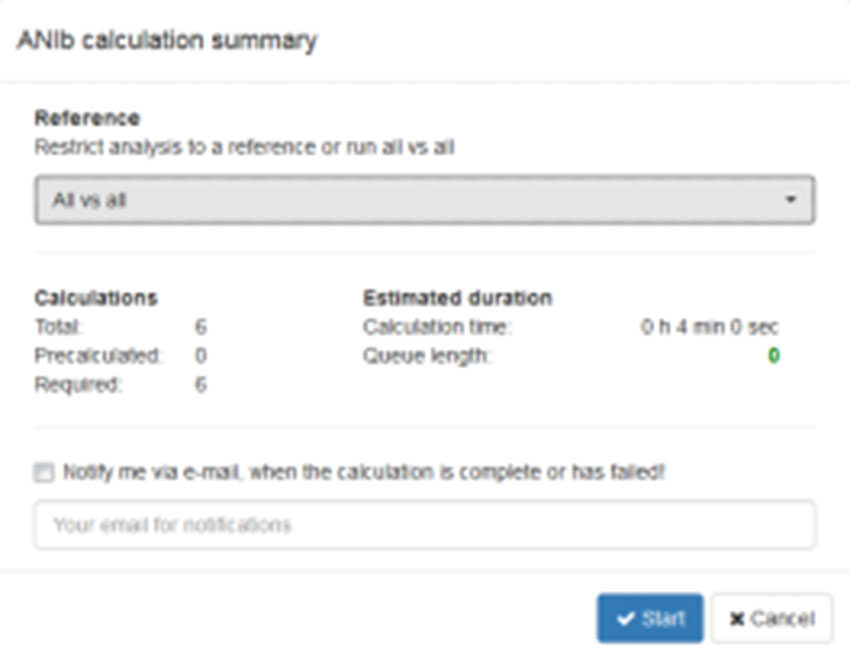
开始分析后,状态字段会定期更新,最终用户会通过弹出消息通知计算完成,也可提供电子邮件地址以获得有关完成计算的信息。
以15个基因组为例,对其基因组全部两两比较(210次计算),采用目前的系统,计算ANIb约2小时,计算ANIm的约30分钟。所有结果都可以重新访问、导出以供进一步处理,并在用户之间共享。
![]()
JspeciesWS代表了JSpecies在可用性、灵活性和效率方面的新发展。想要使用JSpecies又懒得安装软件的童鞋们快来试试吧!


