2018-08-30

文案 | 动植物基因组事业部
之前给大家总结了群体进化中群体遗传结构分析的内容,今天接着进行遗传多样性、基因流、选择性清除等分析内容的学习。
群体进化结果展示
群体遗传结构分析-个体基因组序列差异的度
主要包括:系统进化树、主成分分析(PCA)、群体遗传结构分析
遗传多样性分析
▶ 期望/观测杂合度
随机抽取的两个样本的等位基因不相同的概率。反映了群体中的遗传变异程度,杂合度越高,表明群体内遗传多样性越高。
▶ 核苷酸多样性(π)
是指两个序列间每个位点上核苷酸差异的平均值。在一个随机交配群体中,π是核苷酸水平上的杂合度,反映了群体的多态性高低。
▶ 群体分化指数
Fst代表一个种群内亚群间的遗传分化程度,Fst值一般在 0~1 之间。Fst值越小,代表亚群间的遗传分化越小。
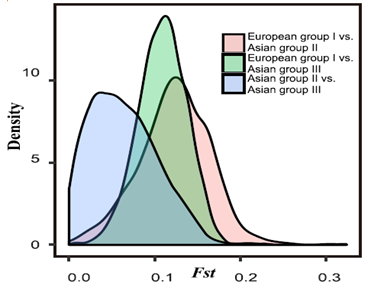
(Wu et al., 2018)
图1:梨品系群体遗传分化Fst值
欧洲group I、亚洲group II和亚洲group III是基于PCA、进化树、群体遗传结构的结果对亚洲野生梨和欧洲野生梨的分组。上图可以看出欧洲 group I 与亚洲 group II之间、欧洲group I 与亚洲 group III之间 、亚洲 group III与亚洲 group II之间的Fst值依次减小,这表明亚洲和欧洲梨的分歧时间比亚洲梨group II和亚洲梨group III的分歧时间早。
基因流分析
生物个体或群体之间由于杂交而导致的基因交流,或指将基因从一个群体的基因库传递到另一个群体的基因库,增加群体间的相似性。
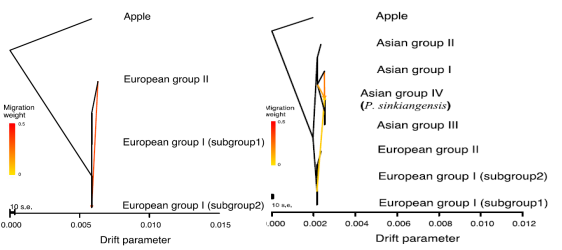
(Wu et al., 2018)
图2:欧洲梨与亚洲梨的基因流分析
颜色的深浅表示基因交流的强弱程度,红色表示强烈的基因交流,黄色表示微弱的基因交流。箭头的方向表明基因交流的方向。上图可以看出欧洲梨group I中的subgroup2与欧洲梨group II之间发生了强烈的基因交流(左图);欧洲梨与亚洲梨之间的基因交流较微弱(右图)。
选择性清除分析-群体水平基因组不同区域遗传多样性变化规律
▶ 连锁不平衡分析(有参)
连锁不平衡又称等位基因关联,是指同一条染色体上,两个等位基因间的非随机关联。
LD 强度与两个SNP 间的距离有关,距离越小,发生重组的机会越小,LD 就越强;反之,距离越大,发生重组的机会越大,LD 就越弱。选择作用会使LD变强。
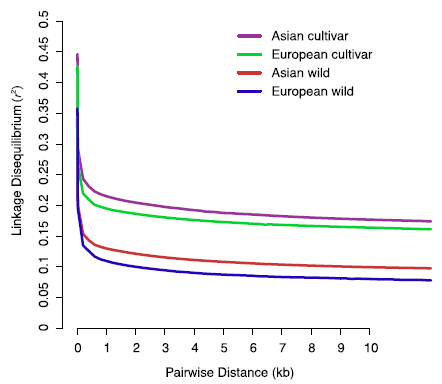
(Wu et al., 2018)
图3 梨品系的连锁不平衡分析
上图为典型的LD decay的展示图,横坐标为SNP对之间的物理距离,纵坐标为SNP对之间的相关系数。上图可以看出野生梨的衰减比栽培种快,栽培种衰减较慢说明群体受到人工选择。梨的基因组的LD距离短、衰减速度快,说明梨在进化过程中受到的人工选择相对较弱。
▶ 选择性清除分析及候选基因功能注释(有参)
基因组某区域由于受到了选择而消除多态化过程,即遗传多样性降低,在群体种出现高频的等位基因和低频的等位基因。
主要用于挖掘驯化过程中、物种适应性进化过程中受选择的基因。
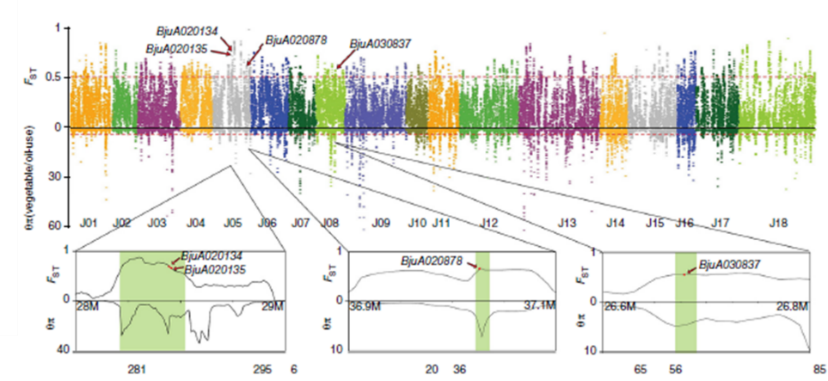
(Yang et al., 2016)
图4:芥菜全基因组Fst值和π比率。图中放大区域分别显示了GSL代谢相关基因和两种脂类代谢相关基因的同源性表达优势和强烈的选择性信号。
有效群体大小分析
有效群体大小指与实际群体有相同基因频率方差或相同杂合度衰减率的理想群体含量,通常小于绝对的群体大小。
有效群体大小的研究有助于了解种群进化历史和复杂性状的遗传机制。
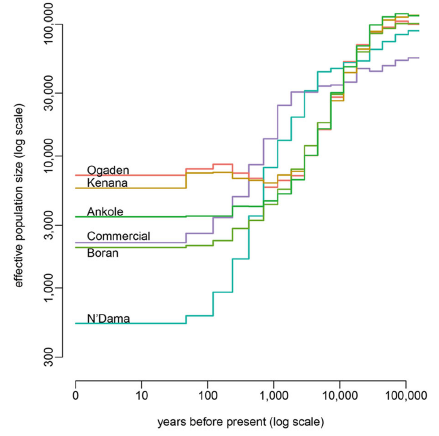
(Kim et al., 2017)
图5:五种非洲牛的有效群体大小的估算(与商业牛比较)
上图展示的是经典的有效种群大小估算的图。横坐标为距今的时间,纵坐标为估算的有效群体大小。上图可以看出N’Dama 牛与其它非洲牛相比,经历了强烈的种群数量的衰减,这与种群的瓶颈效应有关。距今1000年前,Ogaden 和 Kenana 种群大小有略微的扩张,这与非洲大陆的瘤牛入驻有关。大约公元前10000年,所有种群经历了种群数量衰减,可能与当时发生的新石器时代驯化事件相关。
群体进化的礼包大放送,小伙伴们有木有对群体进化有进一步的了解?
派森诺群体进化项目内容丰富,分析多样化,实验方案个性化,你心动了吗?

解析种群信息,揭示群体进化历程
追溯种群历史,寻找进化基因
派森诺竭诚为您服务
期待您的到来~
参考文献
参 考 文 献
[1] Kim J, Hanotte O, Mwai O A, et al. The genome landscape of indigenous African cattle [J]. Genome Biology, 2017, 18(1):34.
[2] Wu J, Wang Y, Xu J, et al. Diversification and independent domestication of Asian and European pears[J]. Genome Biology, 2018, 19(1):77.
[3] Yang J, Liu D, Wang X, et al. The genome sequence of allopolyploid Brassica juncea and analysis of differential homoeolog gene expression influencing selection[J]. Nature Genetics, 2016, 48(10):1225.



