
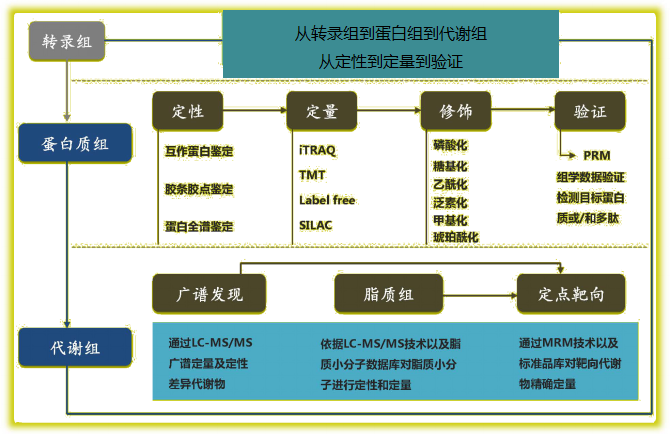
2018-09-25
根据客户提供差异基因(转录本),差异蛋白,差异代谢物可进行如下关联分析:
方案一 转录组与蛋白质组关联分析
方案二 转录组与代谢组关联分析
方案三 蛋白质组与代谢组关联分析
方案四 转录组、蛋白质组与代谢组三者关联网络分析
方案五 代谢组-微生物多样性联合分析
方案一 转录组与蛋白质组联合分析示例
1.1 mRNA与蛋白表达相关性分析
将所有的差异表达蛋白的 mRNA 表达量计算出来,比较两者之间的相关性。我们将基因和蛋白能够完全对应,表达趋势完全一致的列出:
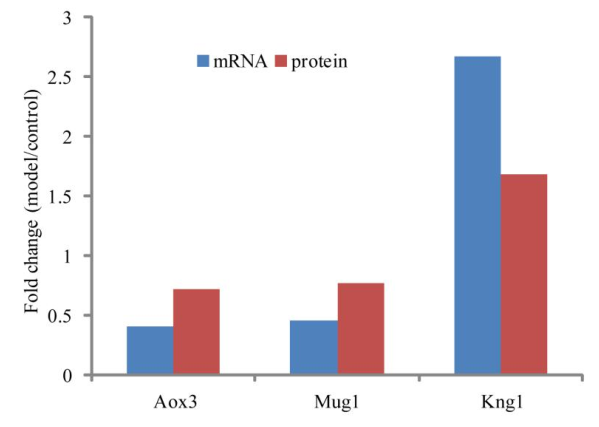
Fig.1 差异蛋白与差异基因表达相关性分析
1.2 差异基因与差异蛋白 pathway 分析
将差异基因与差异蛋白同时使用 GenMAPP v2.1 向 KEGG pathway 数据库映射, 获得他们的共同的 pathway 信息。

Fig.2 差异基因与差异蛋白 pathway 图,带☆的是蛋白,红色的是上调,蓝色的是下调
1.3 差异基因与差异蛋白互作网络构建
整合构建 mRNA 与 protein 互作网络如下:
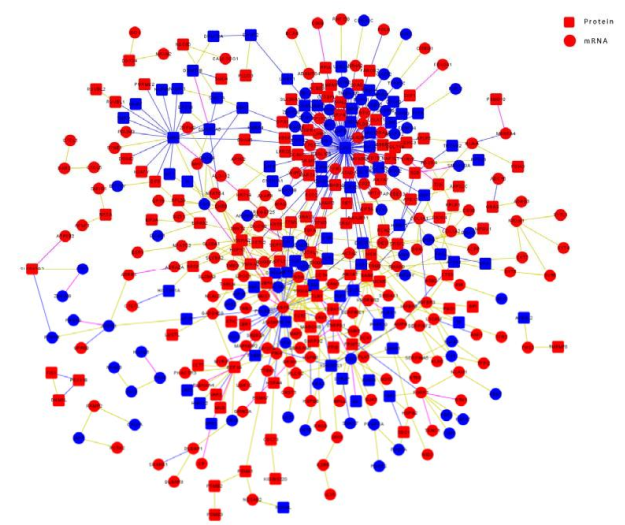
Fig.3 差异基因与差异蛋白整合调控网络构建,圆点是基因,矩形点是蛋白
方案二 转录组与代谢组联合分析示例
2.1 mRNA 与代谢物表达相关性分析
将所有的差异表达代谢物的酶基因对应 mRNA 表达量计算出来,比较两者之间的相关性。我们将基因和代谢物能够完全对应,表达趋势完全一致的列出:
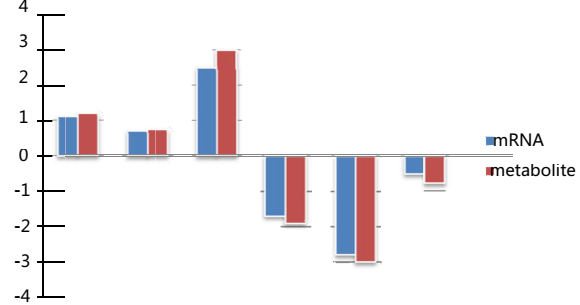
Fig.4 差异代谢物与差异基因表达相关性分析
2.2 差异基因与差异代谢物 pathway 分析
将差异基因与差异代谢物同时使用 GenMAPP v2.1 向 KEGG pathway 数据库映射,获得他们的共同的 pathway信息。
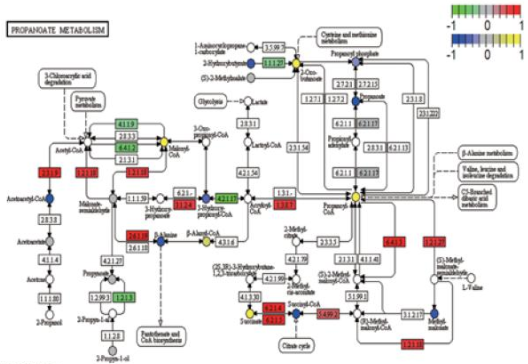
Fig.5 差异基因与差异代谢物 pathway 图,圆点是代谢物,长方形是酶基因
2.3 差异基因与差异代谢物互作网络构建整合构建 mRNA 与代谢物互作网络如下:
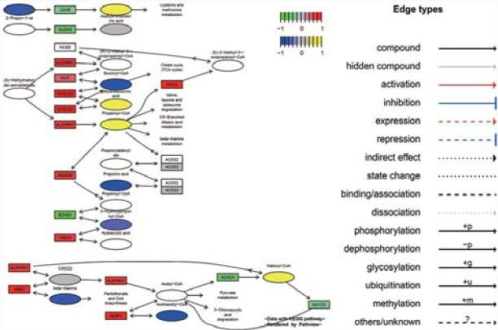
Fig.6 差异基因与差异代谢物整合调控网络构建,长方形是基因,椭圆是代谢物
方案三 蛋白组与代谢组联合分析示例
参考转录组与代谢组关联分析
方案四 转录组、蛋白组与代谢组联合分析示例
综合上述得到的基因、蛋白与代谢之间的调控关系,构建三者的调控网络。

Fig7. IPA 软件构建的基因、蛋白、代谢物互作网络
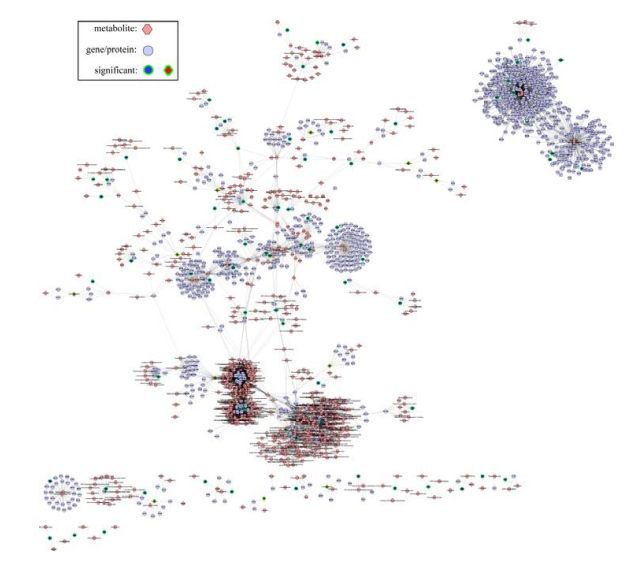
Fig8. metscape 软件构建的基因、蛋白、代谢物互作网络
方案五 代谢组-微生物多样性联合分析示例
1. Mantel test
用于检验来源于相同样本集的两个距离矩阵间的相关性强弱。
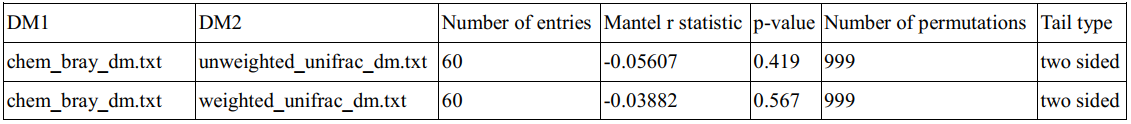
第一和第二列为用于相关性检验的两套数据所对应的距离矩阵;
第三列为样本量;
第四列为Mantel检验得到的相关系数r,r的绝对值越接近1,表明两套数据间的相关性越高; 第五列为P值,P值越接近于零,表明相关性检验的统计显著性越高,结果越可靠;
第六列为置换检验的次数;第七列表明检验类型为单侧或双侧检验。
2. Procrustes分析
可以通过计算各样本在两套数据上的间距平方和(M2),来量化两套数据的一致性高低。还可以对样本进行置换检验,从而评估两套数据之间相似度的统计显著性。
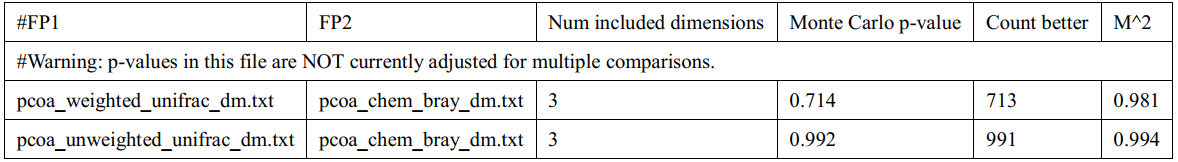
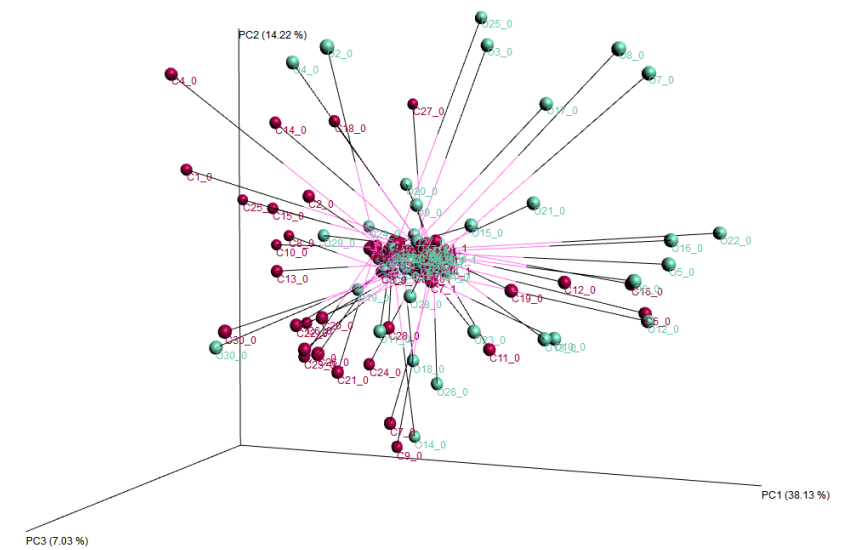
Fig9.Procrustes 分析结果的三维排序图
3. CIA 协惯量分析
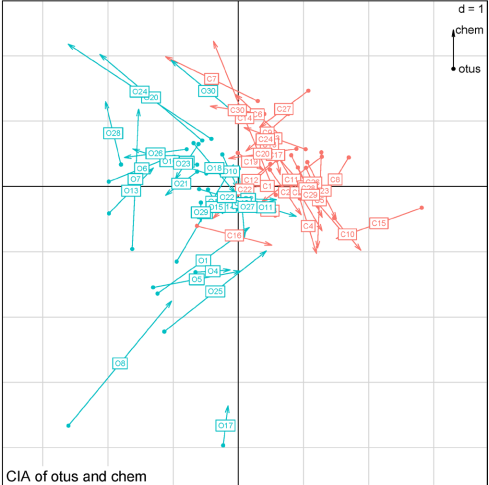
Fig10.CIA 分析结果的二维排序图
参考文献
[1] Sunia A. Trauger,Ewa Kalisak ,J,Gary Siuzdak et. al.Correlating theTranscriptome, Proteome, and Metabolome in the Environmental Adaptation of aHyperthermophile. Journal of Proteome Research 2008, 7, 1027–1035 1027.
[2] Cho K et al. Integrated transcriptomics, proteomics, and metabolomicsanalyses to survey ozone responses in the leaves of rice seedling. J ProteomeRes. 2008Jul;7(7):2980-98.
[3] Coen M et al. Integrated application of transcriptomics and metabonomics yields new insight into the toxicity due to paracetamol in the mouse. J Pharm Biomed Anal. 2004 Apr 1;35(1):93-105.
[4] Espinoza C et al. Interaction with diurnal and circadian regulation resultsin dynamic metabolic and transcriptional changes during cold acclimation in Arabidopsis. PLoS One. 2010 Nov 23;5(11):e14101.
[5] Bono H et al. Comprehensive analysis of the mouse metabolome based on the transcriptome. Genome Res. 2003 Jun;13(6B):1345-9.
[6] Kanani H et al. Individual vs. combinatorial effect of elevated CO2conditions and salinity stress on Arabidopsis thaliana liquid cultures:comparing the early
[7] Mantel, N., The Detection of Disease Clustering and a Generalized Regression Approach. Cancer Research, 1967. 27(2 Part 1): p. 209-220.
[8]Gower, J.C., Generalized procrustes analysis. Psychometrika, 1975. 40(1): p. 33-51.
[9]Dray, S., D. Chessel, and J. Thioulouse, Co-inertia analysis and the linking of ecological data tables.Ecology, 2003. 84(11): p. 3078-3089.