2018-11-07
文案 | 动植物基因组事业部
在基于SNP等分子标记的群体遗传进化分析中,有时会产生构建的系统发育树与预期不符的情况,包括分子系统树与基于表型的系统树不符、与基于氨基酸序列的系统树不符、与基于叶绿体/线粒体序列的系统树不符等情况。对于这一类情况,排除采样和测序造成的误差,我们需要考察造成这种现象的原因,并针对不同原因找到解决方法。
通常情况下,可能的原因有趋同进化、ILS效应、长枝吸引效应等。
趋同进化
趋同进化是指是指亲缘关系较远的生物,由于生活环境、生活方式相似而在长期的适应过程中所形成的体形或器官等异常相似的现象。通常情况下,氨基酸序列比核苷酸序列更容易受到趋同进化的影响。因此可能造成核苷酸进化树与氨基酸进化树不一致的情况。
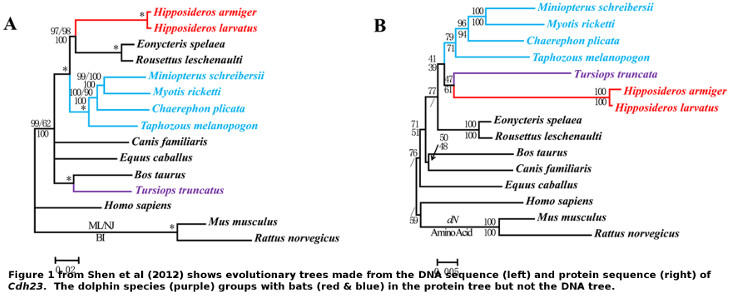
图1: 基于DNA序列(A)与基于蛋白序列(B)的进化树不一致;海豚与蝙蝠在蛋白树中聚类到一起但在DNA树中没有聚在一起
为了进一步鉴定趋同进化的信号,可以采用选择压力分析或祖先序列与现存序列比较等方法。例如,在选择压力分析中,通过计算非同义突变与同义突变率(ω),鉴定位点受选择的情况(ω接近0:纯化选择;ω大于1:正向选择)。趋同进化位点的ω>1且与氨基酸进化树显著相关。

图2:纵轴表示对基因树的支持率,横轴表示ω值;绿点为趋同进化信号位点,蓝点受到纯化选择,红点受到中性选择
ILS效应
不完全谱系分类(incomplete lineage sorting, ILS)是指基于单个基因的系统树与基于物种或群体的系统树不一致的现象。当一个具有等位基因多态性的祖先种分化为两个类群时,两个等位基因在后代群体中都得到了保留,当后代群体进一步分化时,该基因的系统发育关系可能与物种进化关系不符。
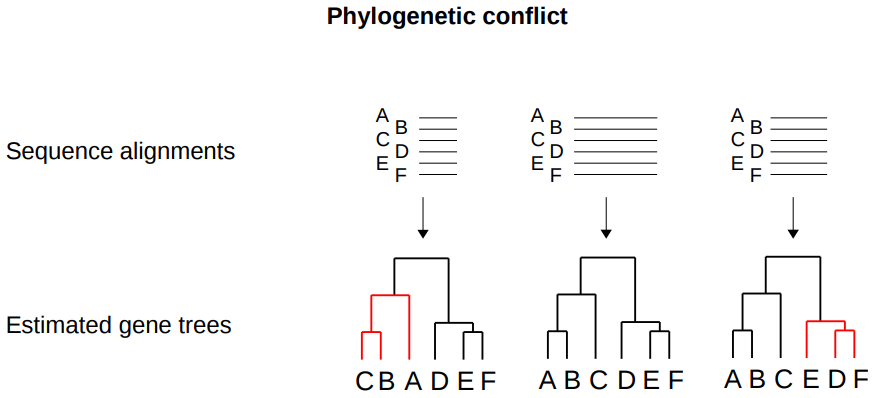
图3:基于不同基因组片段构建进化树间不一致
ILS效应常由于物种快速形成,或经历了快速分化,单个基因的水平上没有演化出足够多的序列差异造成。
为了检测ILS效应,可对全部SNP使用非重叠滑窗法或抽样法生成若干SNP子集,基于每个子集的SNP分别构建进化树,即基因树。若存在ILS效应,不同基因树的拓扑结构之间将存在严重的不一致现象。在这种情况下,可整合全部基因树,计算每个分枝上支持不同拓扑结构的SNP的比例,从而证明ILS的作用大小。
长枝吸引效应
系统发育分析中,“长枝吸引”(Long-branch Attraction, LBA)假象是最为困扰研究者的问题。“长枝吸引”假象由Felsenstein(1978)首次提出,是指在用系统发育分析方法分析一个有限数据集时,由于高频率的相似变化(如趋同、平行进化)和加速的进化速率等因素的存在,使序列达到相同状态,从而人为地将这些不是来自于共同祖先的序列的代表分类元聚在一起,使这些分类元之间相互“吸引”。
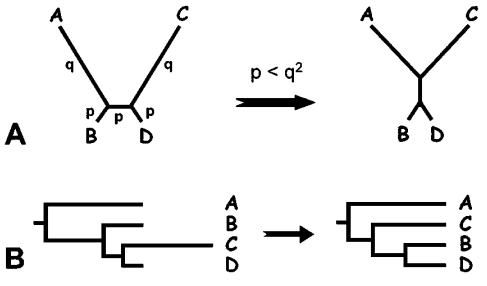
图4:无根树和有根树的长枝吸引
因此,在进行系统发育分析时,应尽可能避免“长枝吸引”假象的产生,从而构建出可靠的系统发育树。
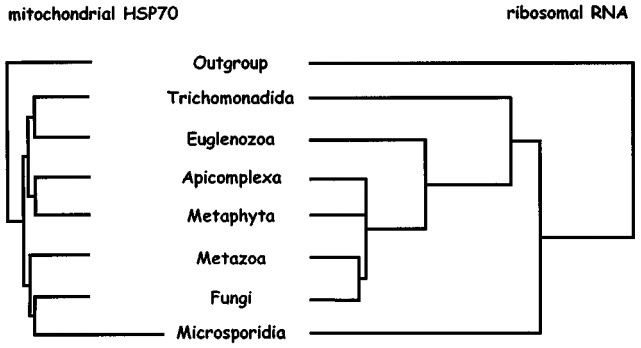
图5:小孢子虫的线粒体和核糖体系统树的长枝吸引,示Microsporidia, Trichomonadida, Euglenozoa三个群体的rDNA树有错位现象
有研究表明似然法不容易产生“长枝吸引”现象,但并不绝对,因此需要将几种方法结合使用来避免“长枝吸引”。
▶ 模型优化法:分析中考虑位点替换速率的异质性,通过设定gamma分布参数优化核苷酸替换模型;
▶ 排除法:包括剔除序列中第三密码子位点、剔除分类群中进化速率较快的长枝分类元等;
▶ 打断法:增加与长枝分类元关系近的分类元以打断长枝;
▶ 无关联数据整合分析法:如结合形态和分子两方面的数据联合分析。
看了以上内容,仍然觉得意犹未尽?
欢迎关注
(☟ 点击下方标题跳转详情)
(上海,2018年11月14日~16日)
在这里,你可以:
参加培训课程,掌握群体进化分析流程
与生信分析工程师面对面交流,解决你的分析相关问题
参观派森诺工作环境,体验生信分析工作氛围
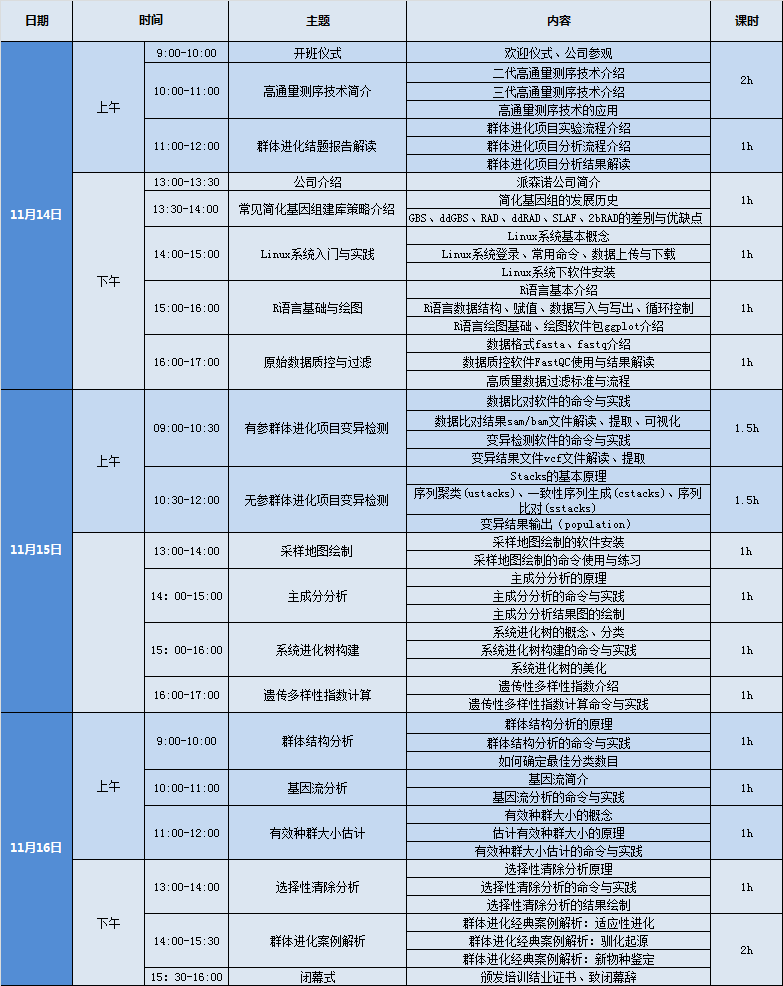
培训内容
参考文献
[1].Shen, Y-Y, Liang, L, Li, G-S, Murphy, RW, and Zhang, Y-P (2012) Parallel Evolution of Auditory Genes for Echolocation in Bats and Toothed Whales. PLoS Genetics 8(6): e1002788. doi:10.1371/journal.pgen.1002788
[2].Liu Y, Cotton J A, Shen B, et al. Convergent sequence evolution between echolocating bats and dolphins[J]. Current Biology, 2010, 20(2):R53-R54.
[3].Scornavacca C, Galtier N. Incomplete Lineage Sorting in Mammalian Phylogenomics.[J]. Systematic Biology, 2016, 66(1):syw082.
[4].Bergsten J. A review of long-branch attraction[J]. Cladistics-the International Journal of the Willi Hennig Society, 2010, 21(2):163-193.
[5].Philippe H. Opinion: long branch attraction and protist phylogeny[J]. Protist, 2000, 151(4):307-316.


